Core germplasm construction of Populus tomentosa based on the fluorescent SSR markers
-
摘要:目的 研究毛白杨核心种质的取样策略,在进行相应的遗传分析基础上,构建核心种质,分析其遗传多样性,为毛白杨种质资源库建设、引种和新品种选育提供科学依据,亦为其他树种核心种质构建提供参考。方法 基于16对荧光SSR引物,利用毛细管电泳技术分析272份毛白杨和白杨杂种种质不同取样比例的遗传多样性参数。根据期望杂合度,计算每个样品对总体遗传多样性的贡献值,然后对所有样品根据贡献值从大到小进行排序。通过比较贡献率最高的前50%、45%、40%、35%、30%、25%、20%和15%取样比例获得的平均有效等位基因数(Ne)、平均Shannon信息指数(I)、平均期望杂合度(He)等,分析核心种质的代表性,确定其合适的取样比例。结果 随着取样比例的降低,Ne、I和He值均在升高,均大于原始种质相应数值,而且He值均大于0.5,表明具有丰富的遗传多样性,而原始种质的He值小于0.5。按照25%取样比例,得到前68名种质,其中包含18份杂种种质以及所有省份选出的部分优异种质。所得到的Ne、I和He分别是2.761、1.094和0.539,均大于原始种质的相应值2.075、0.825和0.432。t检测结果表明,核心种质与原始种质的遗传多样性无显著差异,表明这68份种质在遗传多样性方面具有可靠的代表性,可以作为核心种质。北京的种质与河北的种质遗传一致度最高,为0.997;与山西次之,为0.990。结论 毛白杨核心种质的最佳取样比例是25%,最佳取样范围为20% ~ 40%,如果种质资源数目较大,可以适当降低至15%,如果基数较小,可以升高至45%。He、Ne、I等均表明这些核心种质具有丰富的遗传多样性。杂种种质遗传变异丰富,聚合了亲本的优良等位基因,首次从分子水平证明了杂种种质是毛白杨遗传改良的重要育种资源。建议相关部门或者育种者要高度重视白杨杂种种质的收集、保存和再利用。Abstract:Objective To study the sampling proportion and to determine the sampling strategy on the basis of corresponding genetic analysis, analyzing the genetic diversity and constructing the core germplasm of P. tomentosa can provide molecular foundation for the introduction, genetic breeding, collection and preservation of Leuce germplasm resources. It also can provide reference for the core germplasm construction of other tree species.Method Based on 16 pairs of fluorescent SSR primers, the genetic diversity of 272 accessions of P. tomentosa and poplar hybrid was analyzed by capillary electrophoresis. Based on expected heterozygosity data, all samples were ranked according to their contribution to the overall genetic diversity. The sampling ratios from top to low were 50%, 45%, 40%, 35%, 30%, 25%, 20% and 15%. The representativeness of core germplasm was analyzed by comparing the average effective allele number Ne, the average Shannon information index I and the average expected heterozygosity He, et al. Then the appropriate sampling proportion was determined.Result With the decrease of sampling proportion, the values of Ne, I and He were all increasing, which were all greater than the corresponding values of the original germplasm. The values of He were all greater than 0.5, indicating abundant genetic diversity, while the values of the original germplasms were less than 0.5. According to 25% sampling ratio, the first 68 germplasms were obtained, including 18 hybrid germplasms and some excellent germplasms selected by all provinces. The Ne, I and He values were 2.761, 1.094 and 0.539, respectively, which were all higher than the corresponding values of the original germplasm 2.075, 0.825 and 0.432. It was showed by the t-test that there was no significant difference between the genetic diversity of the core germplasm and the original germplasm resources, indicating that these 68 germplasms have a very reliable representation in terms of genetic diversity and can be used as the core germplasm. The genetic consistency between Beijing germplasm and Hebei germplasm was 0.997, followed by Shanxi of 0.990.Conclusion The best sampling proportion of core germplasm of P. tomentosa is 25%, and the best sampling range is 20%−40%. If the number of germplasm resources is large, it can be reduced to 15%; if the base is small, it can be increased to 45%. He, Ne, I, etc. all indicate that the above core germplasm has abundant genetic diversity. The genetic variation of hybrid germplasm is rich, which aggregates the good alleles of parents. It is proved at the molecular level that hybrid germplasm is an important breeding resource for genetic improvement of P. tomentosa. We suggest that relevant departments or breeders should attach great importance to the collection, preservation and reuse of poplar hybrid germplasm.
-
Keywords:
- Populus tomentosa /
- SSR /
- molecular marker /
- genetic diversity /
- core germplasm
-
毛白杨(Populus tomentosa)是我国重要的乡土树种,具有树干通直圆满、抗干旱、抗病虫、材质优良、树形美观等优良特点,被广泛应用于我国城乡绿化、生态防护林及工业用材林建设中[1-2]。中国具有丰富的毛白杨种质资源,育种研究者如何从中选择出合适的育种材料,既减少工作量,又不过于缩减遗传基础,是目前亟待解决的科学问题。林木种质资源包括森林植物的栽培种、野生种的繁殖材料以及利用上述繁殖材料人工创造的遗传材料,是选育新品种的基础材料,是国家重要的基础战略资源。因此全国各个省、市、自治区越来越重视林木种质资源的收集、保护和保存。由于土地数量有限,租地成本日益攀升,劳务费上涨,所以利用一些技术手段筛选并构建核心种质,具有重要的现实意义。
核心种质是一个作物种质资源库中的具有最小遗传重复的子集,且能代表该种质库的遗传多样性[3]。构建核心种质的标准是筛选出最少的遗传资源来代表整个原始种群的遗传多样性,能够最大限度地保存原始种群的遗传多样性[4]。核心种质构建的依据是在收集表型数据或(和)分子数据的基础上,利用表型或(和)遗传多样性参数,确定取样策略,然后进行统计学检验,如果与原始种质差异不显著,则表明具有代表性。目前Razieh等[5]基于18个形态学标记从所收集的104份伊朗核桃种质中筛选出27份核心种质。钟永达等[6]基于4个形态学标记从所收集的872份中国樟树种质中筛选出217份核心种质。形态学标记简单、直观、易于区分种质资源,然而易受到环境影响而产生一些非遗传变异[7],另外观测记录表型数据需要耗费大量的时间和人力,而且受人为的主观性影响较大。而分子标记是DNA水平遗传多态性的直接反映,不受环境、季节、人为等因素影响,被广泛应用于揭示林木种内的遗传多样性[8-11]。前人在林木分子标记方面取得了一些研究结果,但是所用标记,大部分是显性标记。简单重复序列(SSR)标记是共显性标记,具有多态性高、杂合度大和遗传多样性检测效果高等优点。Liang等[12]基于SSR标记构建了55份苹果(Malus domestica)核心种质。Wang等[13]基于EST-SSR标记成功构建了荔枝(Litchi chinensis)的核心种质。Liu等[14]利用19对SSR标记从251份降香黄檀(Dalbergia hupeana)种质中成功筛选出31份核心种质。传统SSR标记是使用聚丙烯酰胺凝胶电泳,但是不如荧光SSR标记分辨率高。本研究利用荧光SSR标记探讨毛白杨核心种质构建,进而分析其遗传多样性,以期为毛白杨种质资源保存保护、遗传改良提供科技支撑,为其他树种核心种质构建提供参考。
1. 材料与方法
1.1 试验材料
试验材料共有272个毛白杨和白杨杂种无性系,有252份是北京、河北、山东、河南、山西、陕西、甘肃、安徽、江苏省9个省(市)选出的毛白杨优树。其中1 ~ 25号来自北京市,共25份;26~81号来自河北省,共56份;82、83、221、222、232、234、235、237、242 ~ 249、251、254、255号来自山东省,共19份;84 ~ 114、223、225~230、257、258、259来自河南省,共41份;115 ~ 157号和260号来自山西省,共44份;158 ~ 203号、238、239、261号来自陕西省,共49份;204 ~ 208号来自甘肃省,共5份;209 ~ 217来自安徽省,共9份;218 ~ 220、263来自江苏省,共4份。265是新疆杨(P. bolleana)。19份白杨杂种分别是224(毛新杨 × 鲁毛50杨)((P. tomentosa × P. bolleana)× P. tomentosa ‘Lumao 50’)、231(84K)(P. alba × P. glandulosa ‘84K’)、233(银腺杨3号)(P. alba × P. glandulosa 3)、236(毛新杨 × 截叶毛白杨)((P. tomentosa × P. bolleana)× P. tomentosa ‘Truncata’)、240(毛新杨 × 银灰杨)((P. tomentosa × P. bolleana)× P. canescens)、241(毛新杨 × 银灰杨)、250(银腺杨1号 × 新疆杨)((P. alba × P. glandulosa 1)× P. bolleana)、252(银腺杨6号)(P. alba × P. glandulosa 6)、253(银腺杨1号 × 新疆杨)((P. alba × P. glandulosa 1)× P. bolleana)、256(毛新杨 × 截叶毛白杨)((P. tomentosa × P. bolleana)× P. tomentosa ‘Truncata’)、262(银腺杨2号 × 新疆杨)((P. alba × P. glandulosa 2)× P. bolleana)、264(毛新杨 × 截叶毛白杨)((P. tomentosa × P. bolleana)× P. tomentosa ‘Truncata’)、266(毛新杨1号)(P. tomentosa × P. bolleana 1)、267(毛新杨2号)(P. tomentosa × P. bolleana 2)、268(银腺杨1号)(P. alba × P. glandulosa 1)、269(银腺杨2号)(P. alba × P. glandulosa 2)、270(银腺杨5号)(P. alba × P. glandulosa 5)、271(毛新杨 × 截叶毛白杨)((P. tomentosa × P. bolleana)× P. tomentosa ‘Truncata’)和272(响叶杨 × 毛新杨)(P. adenopoda ×(P. tomentosa × P. bolleana))。
试验所用叶片全部采集于山东省冠县国有毛白杨林场。
1.2 试验方法
1.2.1 基因组DNA提取
将硅胶干燥过的叶片按照植物组织基因组DNA提取试剂盒(磁珠法)说明书〔英芮诚生化科技(上海)有限公司,货号PTED−6 030〕操作步骤,提取所有试验材料的DNA。
1.2.2 引物来源
16对引物是由北京林业大学林木育种国家工程实验室基于基因组数据,利用Primer 3软件,在SSR位点侧翼序列设计,并经过多态性和通用性筛选后得到的。详情见表1。
表 1 16对引物序列信息Table 1. Sequence information of 16 pairs of SSR primers引物编号
Primer No.重复基元
Repeat motif5′—3′引物序列
5′−3′ primer sequence3′—5′引物序列
3′−5′ primer sequence荧光
FluorochromePtr_1_SSR1 (CCT)5 AAAGCTTGTGTTCCACTTGT CAGATCTACCTCCTCCATCA TAMRA Ptr_1_SSR2 (AAGA)6 TCTCAATTACATCCCAATCC GGTTGATTCACCAGCAGTAT FAM Ptr_3_SSR13 (TA)10 AGTTGTTTGGGCTGTGTATC GTGCAATTCCCTGATTTAAG FAM Ptr_7_SSR14 (ATT)6 TCCCACAAGCACTCTTAACT CCTTTGCAGCACAGTAGTAA HEX Ptr_7_SSR15 (TG)12 CACTTGCTCTTACTCCTGCT CCAGGACAAATGCAATACTT FAM Ptr_9_SSR3 (CGA)7 TCGAATTCTCCGATAGTGTT AATGCTTTCTTATGCTGCTC FAM Ptr_10_SSR1 (GAC)9 CCAACAACAAGTACCCTCAT AGCTAGAGTCCTGTCTGCTG TAMRA Ptr_11_SSR1 (TA)29 TGAATGAAATACATTGCTGC GCTATTATTGGATTTGCCTG HEX Ptr_11_SSR8 (AT)30 AAATGGAGACTTGTGTGGAC GTGCAATAAAGCAGTGTGAA FAM Ptr_13_SSR6 (GAA)8 TGGTACTCTCCTCTTGCCTA CCTTCAGTTTCTGCTTCATC FAM Ptr_14_SSR7 (GCG)6 TGATTCTACTGGATCCAACC TATCCGATTCTCTAAGGCAA FAM Ptr_14_SSR11 (TCC)8 CTCTCAGTTCTCTTGGATCG GAGGTCTCATTTGTTGAAGC HEX Ptr_14_SSR12 (AT)13 TACTGGTGGTGCTCAATACA AAAGCAAACGCAGTAATAGC HEX Ptr_16_SSR3 (AT)10 GGGATTTCACCACTTATTGA TTATATTTGTCTGGGAGCGT FAM Ptr_18_SSR17 (AT)13 TTTCTGATGCTGTAGCTGTG ATCAGTATGCTTTGCCTGTT TAMRA Ptr_19_SSR1 (TCA)8 ATTTCTTTCGCCTCATAACA CCCTCTTTGTGTGGAATTTA TAMRA 1.2.3 荧光引物PCR扩增与PCR产物毛细管电泳检测
试验所用荧光引物和2 × Taq PCR MasterMix是由北京市睿博兴科生物技术有限公司生产,引物的纯化方式为PAGE。PCR 10 μL体系包括2 × Taq PCR MasterMix 5 μL,DNA(20 ng/μL)0.5 uL,正反引物各0.1 μL,灭菌双蒸水4.3 μL。使用Touchdown模式进行PCR扩增:95 ℃预变性5 min;95 ℃变性30 s,57 ℃退火30 s(每个循环降低0.5 ℃),72 ℃延伸40 s,14个循环;95 ℃变性30 s,50 ℃退火30 s,72 ℃延伸40 s,20个循环;72 ℃延伸10 min。使用毛细管电泳仪(ABI公司的3730XL)检测PCR产物。
1.3 统计分析
使用软件Genemarker 2.2.0分析毛细管电泳数据;使用GeneALeX软件[15]计算平均等位基因数(Na)、实际观察杂合度(Ho)、Nei’s遗传一致度、Ne、、He和I;使用软件DPS对所构建的不同比例核心种质资源的遗传多样性参数进行t检验。
1.4 核心种质取样策略
首先基于期望杂合度数值,通过R语言包Genetic Subsetter计算每个样品对总体遗传多样性的贡献值,然后对所有样品根据贡献值从大到小顺序排序[16]。然后通过比较贡献率最高的前50 %、45 %、40 %、35 %、30%、25%、20%和15%取样比例获得的核心种质的代表性,确定其合适的取样比例。
2. 结果与分析
2.1 毛白杨核心种质排序
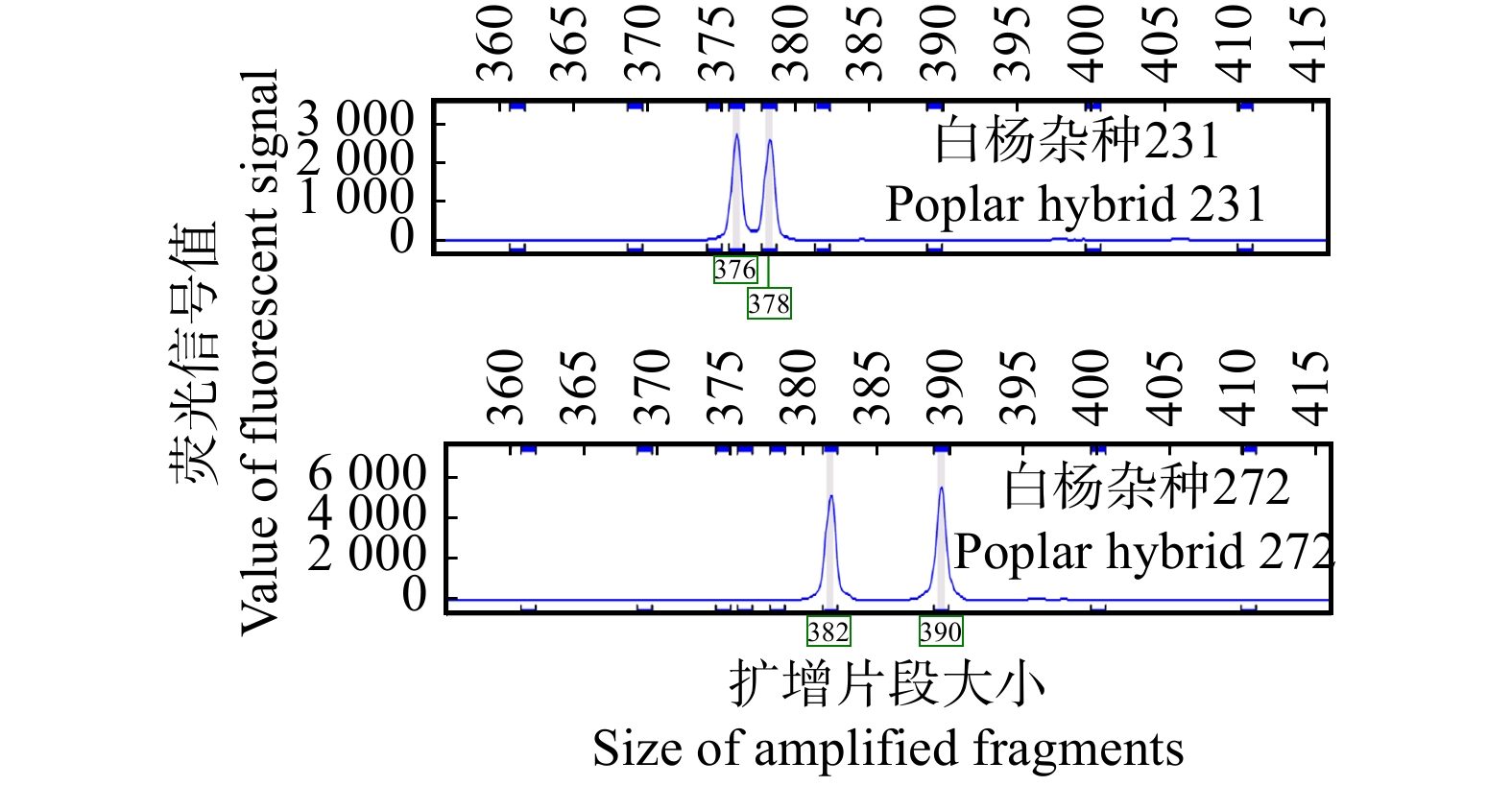
基于16对荧光SSR引物,根据272个样品对总体遗传多样性的贡献程度,按照50%的取样比例,得到前136名排序结果。由表2可以看出,此136份种质包含北京的种质1份(共25份),河北17份(共56份),山东9份(共19份),河南25份(共41份),山西19份(共44份),陕西32份(共49份),甘肃2份(共5份),安徽7份(共9份),江苏4份(共4份),新疆杨1份,杂种种质19份(共19份)。部分种质在Ptr−1−SSR2位点的等位基因变异见图1。其他不同取样比例所得核心种质数量以及不同种源所占数量见表3,可以看出当取样比例为40%时,不再包括北京的任何种质;当取样比例由50%逐渐降至15%时,始终包含江苏种质4份,表明这4份种质具有很好的遗传代表性。另外当取样比例降至15%时,陕西、河南和山东的种质分别有7份、3份和3份入选,具有较好的代表性。
表 2 核心种质排序Table 2. Order of core germplasm顺序
Order品种编号
Variety No.顺序
Order品种编号
Variety No.顺序
Order品种编号
Variety No.顺序
Order品种编号
Variety No.1 156 35 227 69 146 103 85 2 258 36 264 70 224 104 162 3 236 37 211 71 194 105 153 4 253 38 196 72 144 106 84 5 266 39 208 73 216 107 150 6 262 40 176 74 182 108 83 7 261 41 195 75 137 109 99 8 231 42 138 76 106 110 128 9 34 43 193 77 132 111 79 10 241 44 152 78 175 112 53 11 271 45 184 79 130 113 78 12 242 46 180 80 214 114 97 13 252 47 181 81 127 115 77 14 186 48 89 82 100 116 221 15 240 49 26 83 174 117 76 16 237 50 178 84 119 118 91 17 268 51 126 85 212 119 70 18 265 52 161 86 118 120 88 19 263 53 113 87 172 121 67 20 233 54 160 88 112 122 200 21 225 55 259 89 210 123 10 22 267 56 203 90 171 124 66 23 218 57 158 91 107 125 63 24 222 58 110 92 170 126 254 25 270 59 155 93 101 127 61 26 197 60 272 94 209 128 168 27 256 61 202 95 169 129 59 28 269 62 154 96 96 130 248 29 220 63 255 97 166 131 226 30 250 64 151 98 93 132 58 31 191 65 201 99 204 133 165 32 219 66 47 100 86 134 243 33 72 67 147 101 164 135 52 34 215 68 109 102 92 136 105 注:表中所有无性系的来源见1.1试验材料。Note: the source of all clones in the table is shown in chapter 1.1: materials. 表 3 不同取样比例不同种源所占数量Table 3. Number of different provenances at varied sampling proportions比例Ratio/% 北京Beijing 河北Hebei 山东Shandong 河南Henan 山西Shanxi 陕西Shaanxi 甘肃Gansu 安徽Anhui 江苏Jiangsu 新疆Xinjiang 杂交种Hybrid 总计Total 100 25 56 19 41 44 49 5 9 4 1 19 272 50 1 17 9 25 19 32 2 7 4 1 19 136 45 1 11 6 23 19 30 2 7 4 1 19 123 40 0 4 5 20 18 29 2 7 4 1 19 109 35 0 4 4 14 16 29 1 7 4 1 19 96 30 0 4 4 10 14 24 1 4 4 1 19 82 25 0 4 4 8 8 18 1 2 4 1 18 68 20 0 3 3 6 4 14 1 2 4 1 17 55 15 0 2 3 3 1 7 1 2 4 1 17 41 2.2 毛白杨核心种质遗传多样性分析
由16个SSR位点不同取样规模的遗传多样性指标平均值可以看出,当取样比例为20%及以上时,所有取样比例的Na值均大于6(表4)。但是当取样比例为15%时,Na值则小于6,而且观测杂合度也由50%时的0.608逐渐降低至0.601。随着取样比例的降低,Ne、I和He值均在升高,均大于原始种质相应数值。而且He值均大于0.5,表明这些不同取样比例的种质均具有丰富的遗传多样性,具有可靠的代表性,可以作为核心种质资源。但是272份种质的Ne、I和He值分别是2.075、0.825和0.432,表明原始群体具有中度丰富的遗传多样性。利用DPS软件对遗传多样性参数进行t检验,由表5、表6和表7可知,不同取样比例的核心种质遗传多样性参数与原始种质差异均不显著(P > 0.05),能够充分代表原始种质。由表7可知,当取样比例为25%时,Na值大于6,Ne、I和He值分别是2.761、1.094和0.539,表明这个群体的遗传多样性丰富。而且此时数量较少,共68份,保存这些核心种质,占地相对较少。
表 4 不同取样比例的遗传多样性分析Table 4. Analysis of genetic diversity with different sampling proportions样本量
Sample
number
(N)取样比例
Sampling
ratio/%等位基因数
Allele
number
(Na)有效等位基因数
Effective number
of allele
(Ne)Shannon信息指数
Shannon
information index
(I)观测杂合度
Observed
heterozygosity
(Ho)期望杂合度
Expected
heterozygosity
(He)272 100 6.625 2.075 0.825 0.561 0.432 136 50 6.375 2.456 0.982 0.608 0.502 123 45 6.375 2.479 0.998 0.606 0.507 109 40 6.375 2.537 1.023 0.607 0.516 96 35 6.313 2.597 1.047 0.607 0.525 82 30 6.313 2.690 1.083 0.607 0.537 68 25 6.063 2.761 1.094 0.605 0.539 55 20 6.063 2.916 1.141 0.605 0.557 41 15 5.875 3.145 1.205 0.601 0.584 表 5 初始样品及核心种质(20%)的遗传多样性比较Table 5. Comparison of genetic diversity of the primary samples and core germplasm(20%)群体
PopulationN Na Ne I Ho He 初始种质
Primary germplasm272 6.625 2.075 0.825 0.561 0.432 核心种质
Core germplasm55 6.063 2.916 1.141 0.605 0.557 保留率
Retention rate/ %20 91.52 140.53 138.30 107.84 128.94 表 6 初始样品及核心种质(40%)的遗传多样性比较Table 6. Comparison of genetic diversity of the primary samples and core germplasm(40%)群体
Popaul sationN Na Ne I Ho He 初始种质
Primary
germplasm272 6.625 2.075 0.825 0.561 0.432 核心种质
Core germplasm109 6.375 2.537 1.023 0.607 0.516 保留率
Retention rate/%40 96.23 122.27 124.00 108.20 119.44 表 7 初始样品及核心种质(25%)的遗传多样性比较Table 7. Comparison of genetic diversity of the primary samples and core germplasm(25%)群体 Population N Na Ne I Ho He 初始种质 Primary germplasm 272 6.625 2.075 0.825 0.561 0.432 核心种质 Core germplasm 68 6.063 2.761 1.094 0.605 0.539 保留率 Retention rate/% 25 91.52 133.06 132.61 107.84 124.77 2.3 毛白杨不同种源及杂种种质的遗传一致度分析
由表8可知,北京的种质与河北的种质遗传一致度最高,为0.997;与山西次之,为0.990;与杂种种质的遗传一致度为0.756。江苏的种质与河北的种质遗传一致度最高,为0.936;与北京次之,为0.934;与杂种种质的遗传一致度为0.834。
表 8 Nei’s遗传一致度Table 8. Nei’s genetic identity种源
Provenance北京Beijing 河北Hebei 山东Shandong 河南Henan 山西Shanxi 陕西Shaanxi 甘肃Gansu 安徽Anhui 江苏Jiangsu 新疆Xinjiang 杂交种Hybrid 北京 Beijing 1.000 河北 Hebei 0.997 1.000 山东 Shandong 0.978 0.987 1.000 河南 Henan 0.969 0.979 0.982 1.000 山西 Shanxi 0.990 0.996 0.993 0.987 1.000 陕西 Shaanxi 0.938 0.954 0.962 0.984 0.966 1.000 甘肃 Gansu 0.974 0.985 0.988 0.983 0.986 0.957 1.000 安徽 Anhui 0.940 0.950 0.971 0.979 0.958 0.957 0.974 1.000 江苏 Jiangsu 0.934 0.936 0.927 0.934 0.932 0.926 0.921 0.913 1.000 新疆 Xinjiang 0.694 0.707 0.728 0.733 0.720 0.719 0.722 0.728 0.722 1.000 杂交种 Hybrid 0.756 0.774 0.787 0.796 0.790 0.806 0.784 0.783 0.834 0.745 1.000 3. 讨 论
种质资源收集和保存对于保存物种遗传多样性和育种具有重要意义。但是对于一个物种来说,种质资源基因库中的全部种质,是基础群体,数目巨大,不方便开展具体遗传改良研究。所以基于合适的技术手段构建核心种质,对于深入挖掘优异种质资源,提高种质资源的利用价值和效率具有重要的实践意义[4]。有关核心种质的取样比例,前人已经做了一些研究。Balakrishnan等[17]、张洪亮等[18]均认为应根据种质资源总数来确定核心种质取样比例。如果总数很大,则核心种质取样比例可小一些;如果资源较少,则取样比例可相对大一些。当核心种质所占比例超过总数的50%时,则样本量会很大,这不符合核心种质的特征要求[19]。Li等[20]认为农作物的核心种质取样比例为5% ~ 40%,10%左右最佳。本研究从50%取样比例开始,依次降低5%,直至15%,共设计了8个取样比例,依次计算各遗传多样性参数。所有取样比例的全部参数均表明这些取样种质比初始种质(仅是中度丰富)具有更丰富的遗传多样性。通过比较参数Na、Ne、平均I和He,认为毛白杨核心种质的最佳取样比例为25%,最佳取样范围为20% ~ 40%。如果种质资源数目较大,可以适当降低至15%,如果基数较小,可以升高至45%。
遗传多样性分析结果表明,随着取样比例的逐渐降低,其Ne、I和He值均在逐渐的升高,没有重复的数值,可以作为遗传多样性的重要参考指标。然而Na和Ho在取样比例不同时,数值有时却是相同的。例如Ho在取样比例为25%和20%时,数值却是相同的,均为0.605。表明这两个参数只能作为评价遗传多样性的辅助参考。
基于16对荧光SSR引物,根据272个样品对总体遗传多样性的贡献大小,按照50%和45%的取样比例,分别得到136份和123份核心种质。然而仅包含1份北京的种质(总共25份)。所以我们推测北京的毛白杨种质与河北或者其他省份的遗传相似度很高。当取样比例小于等于40%时,不再包含北京的种质,而来自江苏的4份种质(总共4份),从50%比例一直降到15%时,总是包含此4份种质。所以我们推测江苏的种质与其他种源的种质遗传相似度较低,遗传变异较丰富。Nei’s遗传一致度计算结果证明了我们以上的推测是完全正确的。
一般而言,构建核心种质的步骤主要有:收集数据、选择遗传多样性参数、确定取样策略、构建准核心种质和进行代表性检验。本研究的创新之处是首先利用每份种质对总体遗传多样性的贡献大小进行排序,然后通过比较贡献率最高的前50%、45%、40%、35%、30%、25%、20%和15%取样比例获得的核心种质的代表性,初步确定准核心种质,接着利用遗传多样性参数进行验证,最后利用t检验进行统计学上的验证。所以构建的核心种质更具有代表性。
林惠斌等[21]研究表明用毛白杨做母本进行杂交,存在严重败育问题,而用白杨杂种作母本,选用双杂交和回交是有效的育种的方式,因此本研究也收集了部分白杨杂种作为试验材料。从50%取样比例逐渐降到30%时,一直包含19份杂种种质(总共19份);当取样比例为20%和15%时,核心种质数量分别是55份和41份,依然有17份杂种种质入选,表明这些杂种种质遗传变异丰富,聚合了来自父本和母本的优良等位基因。本文首次从分子水平证明了林惠斌和朱之悌先生关于杂种种质是毛白杨遗传改良的重要育种资源的观点。建议相关部门或者育种者要高度重视白杨杂种种质的收集、保存和再利用。
-
表 1 16对引物序列信息
Table 1 Sequence information of 16 pairs of SSR primers
引物编号
Primer No.重复基元
Repeat motif5′—3′引物序列
5′−3′ primer sequence3′—5′引物序列
3′−5′ primer sequence荧光
FluorochromePtr_1_SSR1 (CCT)5 AAAGCTTGTGTTCCACTTGT CAGATCTACCTCCTCCATCA TAMRA Ptr_1_SSR2 (AAGA)6 TCTCAATTACATCCCAATCC GGTTGATTCACCAGCAGTAT FAM Ptr_3_SSR13 (TA)10 AGTTGTTTGGGCTGTGTATC GTGCAATTCCCTGATTTAAG FAM Ptr_7_SSR14 (ATT)6 TCCCACAAGCACTCTTAACT CCTTTGCAGCACAGTAGTAA HEX Ptr_7_SSR15 (TG)12 CACTTGCTCTTACTCCTGCT CCAGGACAAATGCAATACTT FAM Ptr_9_SSR3 (CGA)7 TCGAATTCTCCGATAGTGTT AATGCTTTCTTATGCTGCTC FAM Ptr_10_SSR1 (GAC)9 CCAACAACAAGTACCCTCAT AGCTAGAGTCCTGTCTGCTG TAMRA Ptr_11_SSR1 (TA)29 TGAATGAAATACATTGCTGC GCTATTATTGGATTTGCCTG HEX Ptr_11_SSR8 (AT)30 AAATGGAGACTTGTGTGGAC GTGCAATAAAGCAGTGTGAA FAM Ptr_13_SSR6 (GAA)8 TGGTACTCTCCTCTTGCCTA CCTTCAGTTTCTGCTTCATC FAM Ptr_14_SSR7 (GCG)6 TGATTCTACTGGATCCAACC TATCCGATTCTCTAAGGCAA FAM Ptr_14_SSR11 (TCC)8 CTCTCAGTTCTCTTGGATCG GAGGTCTCATTTGTTGAAGC HEX Ptr_14_SSR12 (AT)13 TACTGGTGGTGCTCAATACA AAAGCAAACGCAGTAATAGC HEX Ptr_16_SSR3 (AT)10 GGGATTTCACCACTTATTGA TTATATTTGTCTGGGAGCGT FAM Ptr_18_SSR17 (AT)13 TTTCTGATGCTGTAGCTGTG ATCAGTATGCTTTGCCTGTT TAMRA Ptr_19_SSR1 (TCA)8 ATTTCTTTCGCCTCATAACA CCCTCTTTGTGTGGAATTTA TAMRA 表 2 核心种质排序
Table 2 Order of core germplasm
顺序
Order品种编号
Variety No.顺序
Order品种编号
Variety No.顺序
Order品种编号
Variety No.顺序
Order品种编号
Variety No.1 156 35 227 69 146 103 85 2 258 36 264 70 224 104 162 3 236 37 211 71 194 105 153 4 253 38 196 72 144 106 84 5 266 39 208 73 216 107 150 6 262 40 176 74 182 108 83 7 261 41 195 75 137 109 99 8 231 42 138 76 106 110 128 9 34 43 193 77 132 111 79 10 241 44 152 78 175 112 53 11 271 45 184 79 130 113 78 12 242 46 180 80 214 114 97 13 252 47 181 81 127 115 77 14 186 48 89 82 100 116 221 15 240 49 26 83 174 117 76 16 237 50 178 84 119 118 91 17 268 51 126 85 212 119 70 18 265 52 161 86 118 120 88 19 263 53 113 87 172 121 67 20 233 54 160 88 112 122 200 21 225 55 259 89 210 123 10 22 267 56 203 90 171 124 66 23 218 57 158 91 107 125 63 24 222 58 110 92 170 126 254 25 270 59 155 93 101 127 61 26 197 60 272 94 209 128 168 27 256 61 202 95 169 129 59 28 269 62 154 96 96 130 248 29 220 63 255 97 166 131 226 30 250 64 151 98 93 132 58 31 191 65 201 99 204 133 165 32 219 66 47 100 86 134 243 33 72 67 147 101 164 135 52 34 215 68 109 102 92 136 105 注:表中所有无性系的来源见1.1试验材料。Note: the source of all clones in the table is shown in chapter 1.1: materials. 表 3 不同取样比例不同种源所占数量
Table 3 Number of different provenances at varied sampling proportions
比例Ratio/% 北京Beijing 河北Hebei 山东Shandong 河南Henan 山西Shanxi 陕西Shaanxi 甘肃Gansu 安徽Anhui 江苏Jiangsu 新疆Xinjiang 杂交种Hybrid 总计Total 100 25 56 19 41 44 49 5 9 4 1 19 272 50 1 17 9 25 19 32 2 7 4 1 19 136 45 1 11 6 23 19 30 2 7 4 1 19 123 40 0 4 5 20 18 29 2 7 4 1 19 109 35 0 4 4 14 16 29 1 7 4 1 19 96 30 0 4 4 10 14 24 1 4 4 1 19 82 25 0 4 4 8 8 18 1 2 4 1 18 68 20 0 3 3 6 4 14 1 2 4 1 17 55 15 0 2 3 3 1 7 1 2 4 1 17 41 表 4 不同取样比例的遗传多样性分析
Table 4 Analysis of genetic diversity with different sampling proportions
样本量
Sample
number
(N)取样比例
Sampling
ratio/%等位基因数
Allele
number
(Na)有效等位基因数
Effective number
of allele
(Ne)Shannon信息指数
Shannon
information index
(I)观测杂合度
Observed
heterozygosity
(Ho)期望杂合度
Expected
heterozygosity
(He)272 100 6.625 2.075 0.825 0.561 0.432 136 50 6.375 2.456 0.982 0.608 0.502 123 45 6.375 2.479 0.998 0.606 0.507 109 40 6.375 2.537 1.023 0.607 0.516 96 35 6.313 2.597 1.047 0.607 0.525 82 30 6.313 2.690 1.083 0.607 0.537 68 25 6.063 2.761 1.094 0.605 0.539 55 20 6.063 2.916 1.141 0.605 0.557 41 15 5.875 3.145 1.205 0.601 0.584 表 5 初始样品及核心种质(20%)的遗传多样性比较
Table 5 Comparison of genetic diversity of the primary samples and core germplasm(20%)
群体
PopulationN Na Ne I Ho He 初始种质
Primary germplasm272 6.625 2.075 0.825 0.561 0.432 核心种质
Core germplasm55 6.063 2.916 1.141 0.605 0.557 保留率
Retention rate/ %20 91.52 140.53 138.30 107.84 128.94 表 6 初始样品及核心种质(40%)的遗传多样性比较
Table 6 Comparison of genetic diversity of the primary samples and core germplasm(40%)
群体
Popaul sationN Na Ne I Ho He 初始种质
Primary
germplasm272 6.625 2.075 0.825 0.561 0.432 核心种质
Core germplasm109 6.375 2.537 1.023 0.607 0.516 保留率
Retention rate/%40 96.23 122.27 124.00 108.20 119.44 表 7 初始样品及核心种质(25%)的遗传多样性比较
Table 7 Comparison of genetic diversity of the primary samples and core germplasm(25%)
群体 Population N Na Ne I Ho He 初始种质 Primary germplasm 272 6.625 2.075 0.825 0.561 0.432 核心种质 Core germplasm 68 6.063 2.761 1.094 0.605 0.539 保留率 Retention rate/% 25 91.52 133.06 132.61 107.84 124.77 表 8 Nei’s遗传一致度
Table 8 Nei’s genetic identity
种源
Provenance北京Beijing 河北Hebei 山东Shandong 河南Henan 山西Shanxi 陕西Shaanxi 甘肃Gansu 安徽Anhui 江苏Jiangsu 新疆Xinjiang 杂交种Hybrid 北京 Beijing 1.000 河北 Hebei 0.997 1.000 山东 Shandong 0.978 0.987 1.000 河南 Henan 0.969 0.979 0.982 1.000 山西 Shanxi 0.990 0.996 0.993 0.987 1.000 陕西 Shaanxi 0.938 0.954 0.962 0.984 0.966 1.000 甘肃 Gansu 0.974 0.985 0.988 0.983 0.986 0.957 1.000 安徽 Anhui 0.940 0.950 0.971 0.979 0.958 0.957 0.974 1.000 江苏 Jiangsu 0.934 0.936 0.927 0.934 0.932 0.926 0.921 0.913 1.000 新疆 Xinjiang 0.694 0.707 0.728 0.733 0.720 0.719 0.722 0.728 0.722 1.000 杂交种 Hybrid 0.756 0.774 0.787 0.796 0.790 0.806 0.784 0.783 0.834 0.745 1.000 -
[1] 徐纬英. 杨树[M]. 哈尔滨: 黑龙江人民出版社, 1988. Xu W Y. Populus spp.[M]. Harbin: Heilongjiang People’s Publishing House, 1988.
[2] 李善文, 张志毅, 何承忠, 等. 中国杨树杂交育种研究进展[J]. 世界林业研究, 2004, 17(2):37−41. doi: 10.3969/j.issn.1001−4241.2004.02.010 Li S W, Zhang Z Y, He C Z, et al. Advances in hybrid breeding of poplar in China[J]. World Forestry Research, 2004, 17(2): 37−41. doi: 10.3969/j.issn.1001−4241.2004.02.010
[3] Frankel O H. Genetic perspectives of germplasm conservation[M]. Cambridge: Cambridge University Press, 1984: 161−170.
[4] Frankel O H, Brown A H D. Plant genetic resources today: a critical appraisal[C]//Holden J H W, Williams J T. Crop genetic resources: conservation and evaluation. London: George Allan and Unwin, 1984: 249−257.
[5] Razieh M, Mohammad R D, Darab H, et al. Development of a core collection in Iranian walnut (Juglans regia L.) germplasm using the phenotypic diversity[J]. Scientia Horticulturae, 2019, 249(4): 439−448.
[6] 钟永达, 赵善文, 程泽龙, 等. 基于种苗表型初步构建中国樟树核心种质[J]. 江西农业大学学报, 2019, 41(1):81−89. Zhong Y D, Zhao S W, Cheng Z L, et al. Preliminary construction of core collection of Cinnamomum camphora based on seedling phenotypic data[J]. Acta Agriculturae Universitatis Jiangxiensis, 2019, 41(1): 81−89.
[7] 毛秀红. 刺槐不同来源无性系种质的表型变异与遗传多样性分析[D]. 北京: 中国林业科学研究院, 2017. Mao X H. Analysis of phenotypic variation and genetic diversity for Robinia pseudoacacia clonal germplasms from different sources[D]. Beijing: Chinese Academy of Forestry, 2017.
[8] 邵辰光, 丁西朋, 白昌军, 等. 利用EST-SSR技术分析银合欢种质资源遗传多样性[J]. 云南农业大学学报(自然科学), 2016, 31(6):1031−1037. Shao C G, Ding X P, Bai C J, et al. Analysis of genetic diversity in Leucaena germplasm using stylosanthes EST-SSRs[J]. Journal of Yunnan Agricultural University (Natural Science), 2016, 31(6): 1031−1037.
[9] Sun R X, Lin F R, Huang P, et al. Moderate genetic diversity and genetic differentiation in the relict tree Liquidambar formosana Hance revealed by genic simple sequence repeat markers[J]. Frontiers in Plant Science, 2016, 7: 1411−1421.
[10] 刘宝尧, 刘小利, 魏海斌, 等. 青海地方核桃种质资源遗传多样性研究[J]. 西北林学院学报, 2017, 32(2):130−135. doi: 10.3969/j.issn.1001−7461.2017.02.22 Liu B Y, Liu X L, Wei H B, et al. Genetic diversity of germplasm resources of walnut in Qinghai[J]. Journal of Northwest University of Forestry, 2017, 32(2): 130−135. doi: 10.3969/j.issn.1001−7461.2017.02.22
[11] 骆鹏, 曹玉婷, 莫家兴, 等. 柳杉无性系指纹图谱的构建及遗传多样性分析[J]. 南京林业大学学报, 2017, 32(2):130−135. Luo P, Cao Y T, Mo J X, et al. Analysis of genetic diversity and construction of DNA fingerprinting of clones in Cryptomeria fortune[J]. Journal of Nanjing Forestry University (Natural Sciences Edition), 2017, 32(2): 130−135.
[12] Liang W, Dondini L, Franceschi P D, et al. Genetic diversity, population structure and construction of a core collection of apple cultivars from Italian germplasm[J]. Plant Molecular Biology Reporter, 2014, 33(3): 1−16.
[13] Wang Y, Fu J X, Pan L J, et al. Construction of core collection of lychee by SSR marker[J]. Acta Horticulturae, 2014, 1029: 87−92.
[14] Liu F M, Zhang N N, Liu X J, et al. Genetic diversity and population structure analysis of Dalbergia odorifera germplasm and development of a core collection using microsatellite markers[J]. Genes, 2019, 10(4): 281−295. doi: 10.3390/genes10040281
[15] Peakall R, Smouse P E. GenAlEx6.5[J]. Bioinformatics, 2012, 28: 2537−2539. doi: 10.1093/bioinformatics/bts460
[16] Maria M A, Alfonso C M, Jefferey B E, et al. The USDA Barley core collection genetic diversity, population structure, and potential for genome-wide association studies[J/OL]. PLoS ONE, 2014, 9(4): 1−13[2019−09−25]. https://pubmed.ncbi.nlm.nih.gov/24732668/.
[17] Balakrishnan R, Nair N V, Sreenivasan T V. A method for establishing a core collection of Saccharum officinarum L. germplasm based on quantitative-morphological data[J]. Genetic Resource and Crop Evolution, 2000, 47(1): 1−9. doi: 10.1023/A:1008780526154
[18] 张洪亮, 李自超, 曹永生, 等. 表型水平上检验水稻核心种质的参数比较[J]. 作物学报, 2003, 29(2):252−257. doi: 10.3321/j.issn:0496-3490.2003.02.016 Zhang H L, Li Z C, Cao Y S, et al. Comparison of parameters for testing the rice core col lection in phenotype[J]. Acta Agronomica Sinica, 2003, 29(2): 252−257. doi: 10.3321/j.issn:0496-3490.2003.02.016
[19] 李慧峰, 陈天渊, 黄咏梅, 等. 基于形态性状的甘薯核心种质取样策略研究[J]. 植物遗传资源学报, 2013, 14(1):91−96. doi: 10.3969/j.issn.1672-1810.2013.01.014 Li H F, Chen T Y, Huang Y M, et al. Sampling strategies of sweet potato core collection based on morphological traits[J]. Journal of Plant Genetic Resources, 2013, 14(1): 91−96. doi: 10.3969/j.issn.1672-1810.2013.01.014
[20] Li Z C, Zhang H L, Zeng Y W, et al. Studies on sampling schemes for the establishment of core collection of rice landraces in Yunnan, China[J]. Genetic Resources and Crop Evolution, 2002, 49(1): 67−74. doi: 10.1023/A:1013855216410
[21] 林惠斌, 朱之悌. 毛白杨杂交育种战略的研究[J]. 北京林业大学学报, 1988, 10(3):97−101. Lin H B, Zhu Z T. Studies on breeding strategies of Populus tomentosa[J]. Journal of Beijing Forestry University, 1988, 10(3): 97−101.
-
期刊类型引用(3)
1. 吴昊,姜俊慧,罗莉,崔益萌,张婉莹. UV-B辐射增强和氮添加及其互作对空心莲子草-马唐竞争的影响. 西南农业学报. 2024(02): 344-352 .  百度学术
百度学术
2. 吴昊,谢闪闪,苏仪函. 氮添加对茶树与入侵杂草空心莲子草种间竞争的影响. 南方农业学报. 2024(05): 1335-1346 .  百度学术
百度学术
3. 吴昊,刘雨欣,李利军,余梅鹏,杨依琳,庄启国,杜奎. 人为干扰对猕猴桃园杂草多样性与土壤因子关系的影响. 西南农业学报. 2024(06): 1313-1321 .  百度学术
百度学术
其他类型引用(0)



 下载:
下载: