Expression patterns and salt tolerance analysis of BpPAT1 gene in Betula platyphylla
-
摘要:目的 GRAS家族是植物特有的具有高度保守羧基末端的转录因子家族,已有研究表明GRAS转录因子是植物胁迫反应中关键的转录调节因子之一。本研究拟对白桦中GRAS转录因子基因BpPAT1基因是否具有耐盐能力进行分析,为阐明白桦GRAS转录因子响应盐胁迫的分子调控机制奠定基础,进一步丰富木本植物GRAS转录因子响应逆境胁迫分子机制的研究。方法 从盐胁迫白桦转录组数据中筛选并获得了1条GRAS转录因子基因,将其命名为BpPAT1。利用蛋白多序列比对及系统进化树来分析BpPAT1与其他GRAS家族蛋白的亲缘关系。利用实时荧光定量PCR(qRT-PCR)技术分析盐胁迫及非胁迫条件下白桦根、茎和叶组织中BpPAT1的表达模式,初步鉴定其是否响应盐胁迫。为了进一步分析BpPAT1的抗逆功能,构建其植物过表达载体(pROKII-BpPAT1)与抑制表达载体(pFGC5941-BpPAT1),利用农杆菌介导的高效瞬时遗传转化体系,获得BpPAT1基因瞬时过表达、抑制表达及对照白桦植株。在盐胁迫下分别对BpPAT1瞬时表达及对照植株的耐盐相关生理指标进行测定,鉴定BpPAT1是否能调控白桦的耐盐能力。结果 多序列比对及系统进化树分析结果表明BpPAT1蛋白具有GRAS家族的序列特征,且与拟南芥中AtPAT1蛋白的亲缘关系较近。qRT-PCR结果表明:在盐胁迫6 h后,BpPAT1在白桦植株中的表达量显著上升(P < 0.05),说明该基因能响应盐胁迫。抗逆生理指标的测定结果表明:在白桦中过表达BpPAT1能够使过氧化物酶(POD)及超氧化物歧化酶(SOD)活性显著增强(P < 0.05),同时增加了白桦组织中的脯氨酸含量,降低了电解质渗透率及丙二醛含量。结论 白桦BpPAT1基因能响应盐胁迫,过表达BpPAT1显著增加了白桦POD、SOD酶活性和脯氨酸含量,降低了电解质渗透率及丙二醛含量,进而提高了ROS清除能力,有效增强了白桦的耐盐能力。Abstract:Objective GRAS family is a plant-specific transcription factor family, characterized by a highly conserved carboxyl terminus domain. Previous studies have shown that GRAS transcription factor is one of the key transcriptional regulators in plant stress response. The purpose of this study is to analyze the salt tolerance of GRAS transcription factor gene BpPAT1 gene in Betula platyphylla, so as to lay a foundation for elucidating the molecular regulation mechanism of GRAS transcription factor in response to salt stress. Our work enriched the research on the molecular mechanism of the GRAS transcription factors of woody plant in response to stress.Method In this study, one GRAS transcription factor gene was screened from the transcriptome data of B. platyphylla under salt stress and named as BpPAT1. Multiple sequence alignment and phylogenetic tree were used to analyze the genetic relationship between BpPAT1 and other organism’s GRAS family genes. Real-time fluorescence quantitative PCR (qRT-PCR) method was used to analyze the expression pattern of BpPAT1
in root, stem and leaf tissues of B. platyphylla under salt stress and normal condition, to identify whether it responded to salt stress or not. In order to further analyze the stress tolerance function of BpPAT1, plant overexpression vector (pROKII-BpPAT1) and inhibitory expression vector (pFGC5941-BpPAT1) were constructed. Transient overexpression and inhibitory expression of BpPAT1 gene and control B. platyphylla plants were obtained by Agrobacterium tumefaciens-mediated transient genetic transformation system. The physiological indexes related to salt tolerance were measured to identify whether the BpPAT1 was associated with salt tolerance in transient expression of BpPAT1 and control plants under salt stress. Result The results of multiple sequence alignment and phylogenetic tree analysis showed that BpPAT1 protein had the sequence characteristics of GRAS family and was closely related to AtPAT1 protein in A. thaliana. The result level of qRT-PCR showed that the expression of BpPAT increased significantly in B. platyphylla plants after 6 hours of salt stress, indicating that BpPAT1could respond to salt stress signal. The measurement results of the physiological indexes of stress resistance showed that the overexpression of BpPAT1 in B. platyphylla could significantly increase the activity of peroxidase (POD) and superoxide dismutase (SOD), increased the content of proline, and decreased electrolyte leakage and malondialdehyde (MDA) content. Conclusion The BpPAT1gene can respond to salt stress, overexpression of BpPAT1 significantly enhances POD, SOD enzyme activities and proline content, decreases electrolyte leakage and MDA content under salt stress, thus improves ROS scavenging ability and salt tolerance of B. platyphylla. -
Keywords:
- Betula platyphylla /
- GRAS transcription factor /
- BpPAT1 /
- gene expression /
- salt stress response
-
植物在生长过程中会遭遇各种逆境,如寒冷、干旱、高盐等非生物胁迫,在漫长的进化过程中植物自身形成了一个高效的逆境胁迫响应及应答机制,使其能在各种逆境条件下生存。转录因子(transcription factors,TFs)又称反式作用因子,能够直接或间接与基因启动子区域顺式作用元件发生特异性结合,并调控基因的转录。研究转录因子在植物逆境表达调控中的作用对揭示植物抗逆机制至关重要[1-2]。
GRAS是近年来发现的一种高等植物特有的转录因子家族,命名来源于其功能成员GAI(gibberellicacid insensitive)、RGA(repressor of GA1-3mutant)和SCR(scarecrow)[3]。GRAS蛋白一般由400 ~ 700个氨基酸残基组成,并且在其C末端含有多个高度保守的基序,如LHRI(leucine heptad repeat I)、VHIID、LHRII(leucine heptad repeat II)、PFYRE和SAW[4]。目前,已经在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、大白菜(Brassica rapa ssp. pekinensis)等植物中鉴定出了GRAS家族基因[5-6]。目前关于GRAS家族基因的研究主要集中在草本模式植物中,对木本植物GRAS转录因子的研究较少,特别是GRAS家族中大多数亚家族的具体作用机制尚不完全清楚。GRAS转录因子作为植物中特有的一类转录因子,在植物的抗逆、生长发育和信号转导过程中起到重要作用[7]。例如,过表达SlGRAS40增强了番茄(Solanum lycopersicum)对干旱和盐胁迫的耐受性[8]。ZjCIGR1基因通过调控包括COR基因在内的下游基因的表达,提高了结缕草(Zoysia japonica)的耐寒性[9]。AtSHR和AtSCR基因参与维持拟南芥根尖分生组织的活性[10-11]。在小麦(Triticum aestivum)中沉默TaSCL14基因会抑制植物生长,降低光合能力,降低对光以及干燥胁迫的耐受性[12]。AtSCL13基因主要负责正向调控光敏色素B的信号传递途径,促使脱黄化的拟南芥下胚轴伸长[13]。
白桦(Betula platyphylla)是一种适应性强、分布广泛、生长迅速的阔叶树种,是我国东北地区具有重要应用价值的树种之一[14]。目前关于白桦GRAS家族的生物信息学分析以及逆境胁迫相关研究还未见报道。本研究在盐胁迫白桦转录组数据中获得了一条GRAS转录因子基因,通过进化树分析发现其与拟南芥AtPAT1的亲缘关系较近,将其命名为BpPAT1。通过qRT-PCR技术,对盐胁迫下BpPAT1基因在不同胁迫时间点及不同组织部位的表达情况进行分析,初步鉴定其是否能响应盐胁迫,然后利用瞬时转化技术获得BpPAT1瞬时表达的白桦植株,并对其耐盐功能进行研究。本研究将为阐明白桦GRAS转录因子响应盐胁迫的分子机制奠定基础,进一步丰富木本植物GRAS转录因子响应逆境胁迫分子机制的研究。
1. 材料与方法
1.1 植物材料与胁迫处理
将野生东北白桦种子均匀撒播于泥炭土、蛭石和珍珠岩体积比为5∶4∶1的混合基质中,置于室温25 ℃、相对湿度65% ~ 70%、光照强度400 μmol/(m2·s)、光周期为14 h光照和10 h黑暗的温室中。选择长势良好且株高相对一致的3月龄的白桦植株,用200 mmol/L NaCl溶液分别胁迫处理3、6、12、24和48 h,用纯水正常浇灌的白桦作为对照(CK),所有胁迫处理均设3次重复。分别取不同胁迫时间点的根、茎及叶组织置于−80 ℃保存,用于后续试验。
白桦无菌组培苗置于温度25 ℃、相对湿度65% ~ 75%、光照强度400 μmol/(m2·s)、光周期为16 h光照和8 h黑暗的人工气候室培养,至株高7 ~ 10 cm用于试验研究。
1.2 质粒与菌株
大肠杆菌(Escherichia coli)Top10、根癌农杆菌(Agrobacterium tumefaciens)EHA105和植物表达载体pROKⅡ、pFGC5941均为本实验室保存。
1.3 白桦BpPAT1基因的克隆及生物信息学分析
在盐胁迫白桦转录组数据中筛选获得一条GRAS转录因子基因,将其命名为BpPAT1。根据基因序列设计引物BpPAT1-F、BpPAT1-R(表1),使用通用植物总RNA提取试剂盒(北京百泰克生物技术有限公司)提取白桦RNA,并使用反转录试剂盒(北京全式金生物技术有限公司)合成第1链cDNA,作为模板进行PCR扩增,将获得的片段连接到pMD18-T载体上,并转化大肠杆菌进行测序验证。
表 1 引物序列Table 1. Primers used in this study用途 Application 引物名称 Primer name 引物序列(5′—3′) Primer sequence (5′−3′) 实时荧光定量 PCR Quantitative real-time PCR q-BpPAT1-F TACTGCTGCATTCTATCCAC q-BpPAT1-R ACTTACCAAGAAGCTCATG q-BpPAT1-OE-F CCCCACATCCGCATAACA q-BpPAT1-OE-R CCCAGGTCGAATCCCAAG q-BpPAT1-IE-F ACGAACGGTGTTGCACTT q-BpPAT1-IE-R GAGCCATAGGTATTGTCAGG Actin-F TGAGAAGAGCTATGAGTTGC Actin-R GTAGATCCACCACTAAGCAC Tubulin-F TCAACCGCCTTGTCTCTCAGG Tubulin-R TGGCTCGAATGCACTGTTGG 基因克隆 Gene cloning BpPAT1-F GCTCTAGAATGTCCAACGGATTGTACTATC BpPAT1-R GGGTACCTCACTTCCATGCACAAGCAG 载体构建 Vector verification pROKⅡ-F AGACGTTCCAACCACGTCTT pROKⅡ-R CCAGTGAATTCCCGATCTAG pFGC5941-cisF CGCTCGAGTATAAGAGCT pFGC5941-cisR ACCTTCCCACAATTCGTCGG pFGC5941-antiF GCATGCTATGCATTCAAT pFGC5941-antiR CGTGCACAACAGAATTGAAAGC pFGC5941-BpPAT1-cisF CCCATGGCAGCTATGCTACAATGATAG pFGC5941-BpPAT1-cisR TTGGCGCGCCTCCACATATAGAAGAGCCAT pFGC5941-BpPAT1-antiF CTCTAGACAGCTATGCTACAATGATAG pFGC5941-BpPAT1-antiR CGGATCCTCCACATATAGAAGAGCCAT 从NCBI(www.ncbi.nlm.nih.gov)数据库中下载8个不同物种的GRAS蛋白序列,利用DNAMAN8与BpPAT1进行多序列比对分析。在拟南芥TAIR(http://www.arabidopsis.org/)数据库获得拟南芥GRAS家族蛋白序列[15],与BpPAT1蛋白进行系统进化树分析。
1.4 BpPAT1基因的表达模式分析
分别提取不同胁迫时间点白桦根、茎及叶组织的总RNA,并反转录成cDNA。根据BpPAT1基因设计q-BpPAT1-F和q-BpPAT1-R引物(表1),选取白桦肌动蛋白基因(Actin,登录号:MK388227)和白桦微管蛋白基因(Tubulin,登录号:FG067376)为内参基因[16],引物序列见表1。利用SYBR Green Realtime PCR Master mix(Toyobo, Japan)试剂盒,在Qtower3G(Analytikjena, German)仪器上进行qRT-PCR分析。为了避免试验误差,确保数据准确性,上述试验设置3个生物学重复。反应体系为10 μL 2 × Power SYBR Green PCRmastermix,2 μL cDNA模板(100 ng)和0.5 μL上下游引物(10 μmol/L)。反应程序为:预变性94 ℃ 30 s,变性94 ℃ 12 s,退火58 ℃ 30 s,延伸72 ℃ 40 s,读板82 ℃ 1 s,45个循环。采用2−ΔΔCt法对基因的表达进行相对定量分析[17]。
1.5 pROKⅡ-BpPAT1植物过表达载体的构建
根据植物过表达载体pROKⅡ的多克隆位点,结合BpPAT1基因序列设计引物pROKⅡ-BpPAT1-F和pROKⅡ-BpPAT1-R(表1),引入XbaI和KpnI酶切位点,以白桦cDNA为模板进行PCR扩增BpPAT1基因。用XbaI和KpnI对纯化后pROKⅡ质粒与BpPAT1基因进行双酶切。用T4 DNA连接酶对线性化的载体和基因进行连接,转化大肠杆菌,并进行测序验证。对测序比对正确的重组载体pROKⅡ-BpPAT1提取质粒并转化到农杆菌EHA105感受态中,保存菌种备用。
1.6 pFGC5941-BpPAT1植物抑制表达载体的构建
根据植物抑制表达载体pFGC5941的多克隆位点以及BpPAT1基因序列设计引物pFGC5941-BpPAT1-cisF和pFGC5941-BpPAT1-cisR、pFGC5941-BpPAT1-antiF和pFGC5941-BpPAT1-antiR(表1),分别引入NcoI、AscI和XbaI、BamHI酶切位点,以pROKⅡ-BpPAT1质粒为模板进行PCR,对得到的产物进行双酶切。用T4DNA连接酶对线性化的载体和基因进行连接,转化大肠杆菌并测序。经测序比对正确后,提取pFGC5941-BpPAT1重组载体质粒,并转化EHA105农杆菌感受态,保存菌种备用。
1.7 BpPAT1基因瞬时表达白桦的获得
将载体pROKⅡ-BpPAT1-EHA105、pFGC5941-BpPAT1-EHA105和pROKⅡ-EHA105按照农杆菌介导的高效瞬时转化技术技术[18]瞬时侵染4周苗龄的白桦组培苗,获得BpPAT1基因瞬时过表达(OE)、抑制表达(IE)及对照(Control)植株。为了研究BpPAT1基因在瞬时表达白桦植株中的表达情况,将上述白桦分别移入含150 mmol/LNaCl的1/2MS培养基中胁迫24 h。分别提取盐胁迫及正常条件下(1/2MS培养基)OE、IE及Control植株的总RNA,并反转录合成cDNA,进行qRT-PCR分析。
1.8 BpPAT1基因瞬时表达白桦的抗逆生理指标分析
将BpPAT1基因瞬时过表达、抑制表达及对照白桦植株分别移入含150 mmol/L NaCl的1/2MS培养基中胁迫24 h,分别取瞬时表达和对照白桦植株叶片,置于2 mL离心管中,加入1.5 mL二氨基联苯胺(DAB)或氯化硝基四氮唑蓝(NBT)染色液,室温染色过夜。染色结束后,用75%乙醇和5%甘油沸水浴脱色,拍照。取各胁迫处理的瞬时表达和对照白桦植株,依次测定各植株内的过氧化物酶(POD)及超氧化物歧化酶(SOD)活性,电解质渗透率、丙二醛(MDA)及脯氨酸含量,方法参照王关林等[19]。为了避免试验误差,并确保数据的准确性,每次试验至少包含6株白桦幼苗,并进行3次重复。所有的数据分析全部应用SPSS21.0进行处理,并采用Duncan法来判断差异显著性,显著性水平设为P < 0.05。
2. 结果与分析
2.1 BpPAT1基因的克隆及生物信息学分析
BpPAT1基因的开放阅读框长度为1 608 bp,编码535个氨基酸。利用NCBI网站对BpPAT1编码的氨基酸序列进行同源性搜索BLAST,发现该序列与长穗鹅耳枥(Carpinus fangiana)和欧洲栓皮栎(Quercus suber)的GRAS家族基因相似性较高,分别为90.81%和85.85%。通过蛋白多序列比对及系统进化树来分析BpPAT1编码的氨基酸序列与其他9个物种的GRAS蛋白相似性,结果显示,BpPAT1蛋白具有GRAS家族的序列特征,其在C端的氨基酸序列相似度比较高(图1A)。与拟南芥33个GRAS蛋白进化树分析结果显示,BpPAT1与AtPAT1蛋白亲缘关系较近,同属于AtPAT1亚家族(图1B)。
![]() 图 1 白桦BpPAT1蛋白的多序列比对分析(A)及系统进化树分析(B)AtPAT1. 拟南芥(NP_001332482.1);QsGRAS. 欧洲栓皮栎(XP_023916980.1);MrGRAS. 杨梅(KAB1208676.1);JrGRAS. 胡桃(XP_018849898.1);VvGRAS. 葡萄(XP_002272334.1);JcGRAS20. 麻风树(XP_012081428.1);CfGRAS. 土瓶草(GAV74587.1);DlGRAS54. 龙眼(AGE44291.1);TcGRAS. 可可(EOX93442.1)。AtPAT1, Arabidopsis thaliana (NP_001332482.1); QsGRAS, Quercus suber (XP_023916980.1); MrGRAS, Morella rubra (KAB1208676.1); JrGRAS, Juglans regia (XP_018849898.1);VvGRAS, Vitis vinifera (XP_002272334.1); JcGRAS20, Jatropha curcas (XP_012081428.1); CfGRAS, Cephalotus follicularis (GAV74587.1); DlGRAS54, Dimocarpus longan (AGE44291.1); TcGRAS, Theobroma cacao (EOX93442.1).Figure 1. Multiple sequence alignment analysis of Betula platyphylla BpPAT1 protein (A) and phylogenetic tree analysis (B)
图 1 白桦BpPAT1蛋白的多序列比对分析(A)及系统进化树分析(B)AtPAT1. 拟南芥(NP_001332482.1);QsGRAS. 欧洲栓皮栎(XP_023916980.1);MrGRAS. 杨梅(KAB1208676.1);JrGRAS. 胡桃(XP_018849898.1);VvGRAS. 葡萄(XP_002272334.1);JcGRAS20. 麻风树(XP_012081428.1);CfGRAS. 土瓶草(GAV74587.1);DlGRAS54. 龙眼(AGE44291.1);TcGRAS. 可可(EOX93442.1)。AtPAT1, Arabidopsis thaliana (NP_001332482.1); QsGRAS, Quercus suber (XP_023916980.1); MrGRAS, Morella rubra (KAB1208676.1); JrGRAS, Juglans regia (XP_018849898.1);VvGRAS, Vitis vinifera (XP_002272334.1); JcGRAS20, Jatropha curcas (XP_012081428.1); CfGRAS, Cephalotus follicularis (GAV74587.1); DlGRAS54, Dimocarpus longan (AGE44291.1); TcGRAS, Theobroma cacao (EOX93442.1).Figure 1. Multiple sequence alignment analysis of Betula platyphylla BpPAT1 protein (A) and phylogenetic tree analysis (B)2.2 盐胁迫下BpPAT1基因的表达模式分析
为研究盐胁迫下BpPAT1基因的表达情况,分别提取胁迫0、3、6、12、24及48 h的白桦根、茎及叶组织的总RNA,进行qRT-PCR分析。结果显示:盐胁迫下,BpPAT1基因呈上调表达模式,在根组织中胁迫24 h表达量最高,茎组织中胁迫3 h表达量最高,而叶组织中胁迫6 h的表达量最高(图2)。上述结果表明:BpPAT1基因在白桦根、茎和叶组织中均有表达且响应盐胁迫。
2.3 盐胁迫下瞬时转化白桦中BpPAT1基因的表达分析
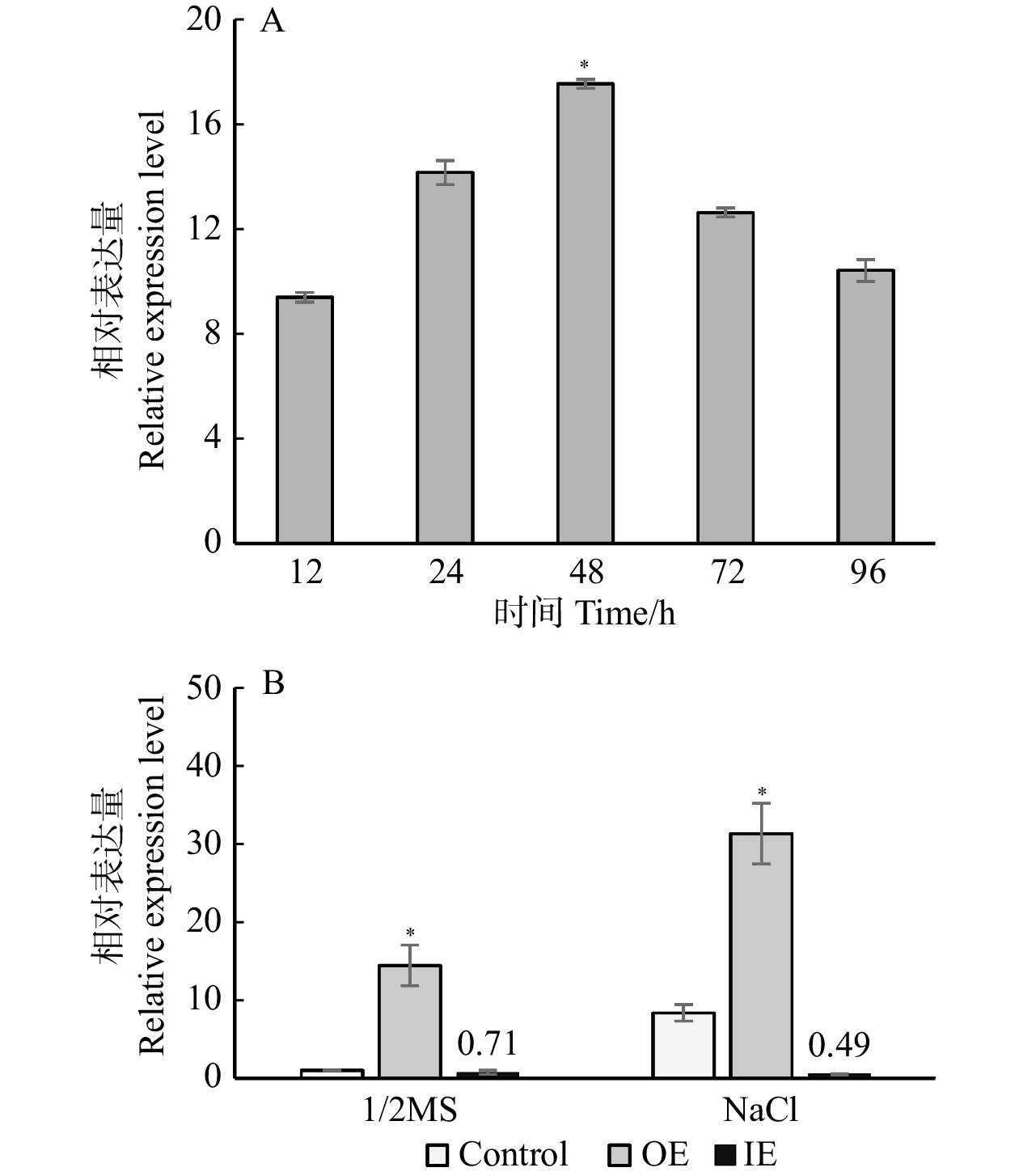
通过瞬时转化系统获得BpPAT1基因瞬时过表达白桦植株,利用qRT-PCR技术分别对不同转化后时间点(12、24、48、72及96 h)的过表达(OE)及对照(Control)白桦植株中BpPAT1基因的表达水平进行分析。结果表明,转化后48 h时,BpPAT1在OE植株中的表达量最高(图3A)。说明BpPAT1已经成功在瞬时转化的白桦植株中过表达,瞬时转化的白桦植株可用于后续的功能获得研究。通过qRT-PCR对OE、IE及Control白桦植株中BpPAT1基因的表达水平进行分析,以转化后48 h的对照白桦植株的表达量作为基准值(记作1)来均一化BpPAT1基因的表达。qRT-PCR结果表明,在正常条件下(1/2MS培养基),OE白桦植株中BpPAT1的表达量明显高于Control和IE植株;在盐胁迫条件下(含150 mmol/L NaCl的1/2MS培养基),OE白桦植株中BpPAT1的表达量相对于Control显著上升,表明该基因在瞬时转化白桦植株中已经成功过表达。在IE植株中,其表达量相对于Control显著降低,说明BpPAT1在瞬时转化白桦植株中已经成功被抑制表达(图3B)。上述结果表明已成功获得了BpPAT1基因瞬时过表达及抑制表达白桦植株,可用于后续研究。
![]() 图 3 盐胁迫下BpPAT1基因在瞬时表达白桦植株中的表达情况A. 盐胁迫下不同转化时间对照植株及瞬时过表达植株BpPAT1基因的表达水平;B. 正常条件下与盐胁迫后对照植株及瞬时转化植株BpPAT1基因的表达水平;Control. 对照植株;OE. 瞬时过表达植株;IE. 瞬时抑制表达植株;*. 显著性差异(P < 0.05)。下同。A, expression levels of BpPAT1 gene in control plants and transient overexpressed plants at different transformation time under salt stress; B, expression level of BpPAT1 gene in control plants and transient transformed plants under normal conditions and salt stress. Control, control plant; OE, transient overexpression plant; IE, transient inhibitory expression plant. Asterisks indicate significant difference (P < 0.05). The same below.Figure 3. Expression of BpPAT1 gene in transient expression plants of B. platyphylla under salt stress
图 3 盐胁迫下BpPAT1基因在瞬时表达白桦植株中的表达情况A. 盐胁迫下不同转化时间对照植株及瞬时过表达植株BpPAT1基因的表达水平;B. 正常条件下与盐胁迫后对照植株及瞬时转化植株BpPAT1基因的表达水平;Control. 对照植株;OE. 瞬时过表达植株;IE. 瞬时抑制表达植株;*. 显著性差异(P < 0.05)。下同。A, expression levels of BpPAT1 gene in control plants and transient overexpressed plants at different transformation time under salt stress; B, expression level of BpPAT1 gene in control plants and transient transformed plants under normal conditions and salt stress. Control, control plant; OE, transient overexpression plant; IE, transient inhibitory expression plant. Asterisks indicate significant difference (P < 0.05). The same below.Figure 3. Expression of BpPAT1 gene in transient expression plants of B. platyphylla under salt stress2.4 BpPAT1基因瞬时表达及对照白桦植株活性氧(ROS)水平分析
POD能够催化H2O2氧化其他底物产生H2O2,是清除植物体内氧自由基伤害的重要保护酶,与植物的抗逆能力密切相关。SOD能够通过催化O2−自由基的歧化反应,抵御活性氧或其他过氧化物自由基对细胞膜系统的损伤,有效地增强植物的抗逆功能[20]。对瞬时表达及对照白桦植株在盐胁迫条件下POD和SOD的活性进行研究。分别取各胁迫处理24 h的瞬时表达和对照白桦植株(3次重复)来测定POD和SOD活性。结果如图4A、C所示:在非胁迫条件下,BpPAT1基因的OE、IE及Control植株的POD、SOD活性无明显差异;盐胁迫条件下,BpPAT1基因OE植株的POD、SOD活性明显高于Control及IE植株;且与正常生长条件相比,BpPAT1基因OE植株的POD、SOD活性显著上升。结果表明:过表达BpPAT1基因增加了白桦植株POD和SOD活性。
通过DAB和NBT染色检测白桦体内H2O2和O2−的含量,以研究BpPAT1基因瞬时表达及对照白桦植株的ROS水平。DAB染色结果表明:在非胁迫条件下(0 h),OE、IE及Control植株棕色均较浅且无明显差异,说明各植株中H2O2含量基本相同;盐胁迫下,OE植株叶片上的棕色明显浅于IE及Control植株叶片,且与正常条件下相比,OE植株叶片上的颜色变化程度不大,说明OE植株叶片内的H2O2含量比IE及Control植株要少(图4B)。NBT染色结果表明:在非胁迫条件下(0 h),OE、IE及Control植株蓝色均较浅且无明显差异,说明各植株中O2−含量基本相同;盐胁迫下,OE植株叶片上的蓝色明显浅于IE及Control植株叶片,且与正常条件下相比,OE植株叶片上的颜色变化不大,说明OE叶片内的O2−的含量较IE及Control植株叶片要少(图4D)。上述结果说明,过表达BpPAT1基因能够降低白桦体内H2O2和O2−含量,从而增强白桦的ROS清除能力,提高了白桦的耐盐能力。
2.5 BpPAT1基因瞬时表达及对照白桦植株中细胞受损情况和MDA含量分析
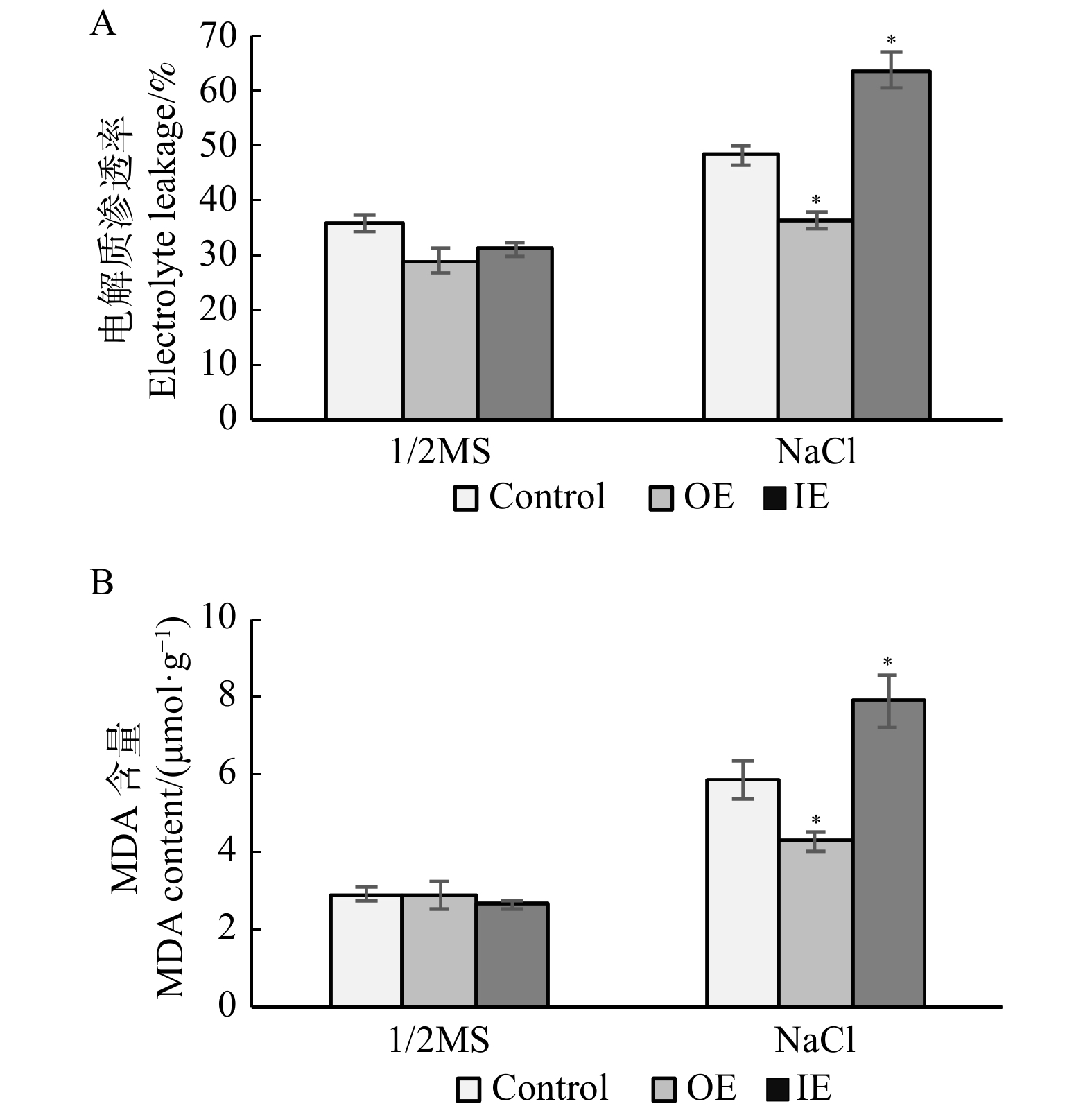
在逆境下,植物的细胞膜受损严重,导致植物细胞内的电解质外渗,破坏了植物细胞膜的通透性以及细胞膜的完整性,直接影响了植物的抗逆能力[21]。分别取盐胁迫处理24 h的瞬时表达和对照白桦植株叶片(3个重复)进行电解质渗透率测定,结果见图5A。正常生长状态下,瞬时表达和对照白桦植株的电解质渗透率基本一致;盐胁迫下,IE和Control植株的电解质渗透率明显高于OE植株,且IE植株的电解质渗透率最高,且与正常条件下相比,OE植株的电解质渗透率无明显提高,而IE植株电解质渗透率显著提高。该结果说明过表达BpPAT1基因通过降低白桦的电解质渗透率,保护了细胞膜的通透性及细胞膜结构的完整性。
丙二醛是植物器官在逆境条件下或衰老时发生膜脂过氧化作用而产生的一种有机化合物,它的产生加剧细胞膜的损伤。因此,MDA含量通常用于表示细胞膜脂过氧化程度和植物对逆境条件反应的强弱的指标[22]。对瞬时表达及对照白桦植株的MDA含量进行进一步研究,分别取瞬时表达和对照白桦植株叶片(3次重复)进行MDA测定,结果如图5B所示:在正常生长条件下,瞬时表达和对照白桦植株的MDA含量无明显差异;而在盐胁迫下,IE及Control植株的MDA含量明显高于OE植株,OE植株的MDA含量最低,而IE植株的值最高,与正常生长条件下相比,OE植株的MDA含量无明显提高,而IE植株的MDA含量显著提高。该结果说明BpPAT1基因的过表达能显著降低白桦在盐胁迫下MDA的积累,从而增强其耐盐能力。
2.6 BpPAT1基因瞬时表达及对照白桦植株中脯氨酸含量分析
脯氨酸是植物蛋白质的组分之一,可以游离状态广泛存在于植物体中,在植物体内起到渗透调节和渗透保护作用。在干旱、盐渍等胁迫条件下,许多植物体内脯氨酸大量积累。积累的脯氨酸除了作为植物细胞质内渗透调节物质外,还在稳定生物大分子结构、降低细胞酸性、解除氨毒以及作为能量库调节细胞氧化还原势等方面起重要作用[23]。对瞬时表达及对照白桦植株在盐胁迫条件下的脯氨酸含量进行研究。分别取各胁迫处理24 h的瞬时表达和对照白桦植株(3次重复)测定脯氨酸含量。结果如图6所示:在正常生长条件下,BpPAT1基因OE、IE及Control植株的脯氨酸含量无明显差异;在盐胁迫条件下,BpPAT1基因OE植株的脯氨酸含量明显高于Control及IE植株,OE植株的脯氨酸含量最高,而IE植株的脯氨酸含量最低,且与正常生长条件相比,BpPAT1基因OE植株的脯氨酸含量显著上升。上述结果表明过表达BpPAT1基因增加了植物体内的脯氨酸含量,进而提高了白桦的耐盐能力。
3. 讨 论
GRAS蛋白是植物特有的一类转录因子,其大多都有一个保守的C端DNA结合结构域,和一个多样化的N端结构域[24-25]。本研究将BpPAT1与9个物种的GRAS蛋白进行多序列比对,结果显示BpPAT1具有1个保守的C端DNA结合结构域,说明该蛋白具有GRAS家族的序列特征(图1A)。通过与拟南芥33个GRAS蛋白进行系统进化树分析,结果表明BpPAT1与AtPAT1蛋白亲缘关系较近(图1B)。对BpPAT1基因在不同盐胁迫时间点、不同组织中的表达模式进行分析,发现BpPAT1基因在白桦根、茎和叶组织中均有表达,且能响应盐胁迫。该结果与之前GRAS转录因子能够响应盐胁迫的研究一致,如:Ma等[26]在胡杨中(Populus euphratica)克隆出了PeSCL7基因,在盐胁迫下PeSCL7的表达量逐渐上调,且在3 h表达量最高,说明PeSCL7能够积极响应盐胁迫;周莲洁等[27]用高浓度NaCl处理盐穗木幼苗发现,GRAS家族基因HcSCL13表达量随着时间的延长逐渐上调,说明HcSCL13可以响应盐胁迫。
本研究采用的瞬时转化系统是一种快速、有效的遗传转化方法,该方法对木本植物基因抗逆功能的研究具有重要意义。目前,已在多个物种中利用瞬时转化系统对目的基因的抗逆功能进行了鉴定。Liu等[28]利用瞬时转化技术证明BpHSP9在白桦耐热性方面发挥重要的作用。Zang等[29]发现ThZFP1通过增加脯氨酸含量和SOD、POD活性来提高刚毛柽柳(Tamarix hispida)对盐和渗透胁迫的耐受性。通过过表达ThZFP1转基因拟南芥植株验证了瞬时表达柽柳植株的结果,Yang等[30]通过瞬时转化证明过表达ThDREB增强了刚毛柽柳的耐盐及耐干旱胁迫性。在本研究中,瞬时转化后qRT-PCR结果表明:在过表达和抑制表达植株中BpPAT1基因的表达量显著的上升或下降,且转化后48 h,BpPAT1在瞬时过表达植株中的表达量最高。这说明已经成功获得瞬时转化植株,上述瞬时表达白桦可用于后续的功能获得和缺失的研究。
为了进一步研究BpPAT1的耐盐功能,分别对BpPAT1基因过表达、抑制表达及对照白桦植株中的关键抗逆生理指标进行了测定。根据POD、SOD活性测定及DAB、NBT染色结果,判断白桦体内的ROS水平(图4)。结果显示:盐胁迫下,过表达BpPAT1能显著提高POD和SOD的活性,从而增强白桦植株的ROS清除能力,进而提高了白桦植株的耐盐性。Li等[31]在研究中发现,过表达BrLas通过增强ROS清除能力提高了转基因拟南芥的抗旱性。Guo等[32]通过研究发现BplMYB46通过影响SOD、POD和P5CS基因的表达,提高了活性氧清除能力和脯氨酸水平,从而提高了白桦对盐和渗透胁迫的耐受性。He等[33]研究发现ThNAC7诱导与胁迫耐受性相关的基因表达,通过增加渗透势和增强ROS清除能力来提高刚毛柽柳对盐和渗透胁迫的耐受性。因此,我们推断BpPAT1基因可能通过调控POD、SOD酶等相关基因的表达增加了POD和SOD的活性,从而降低ROS在逆境下的积累,进而增加白桦的耐盐能力。BpPAT1基因的过表达降低了白桦在逆境胁迫下的电解质渗透率及MDA含量(图5),增加了脯氨酸含量(图6),保证了细胞膜结构的完整性,通过提高渗透势从而提高了白桦的耐盐能力。同一个亚家族的成员具有相似的结构域特征,往往在植物生长代谢过程中具有相似的功能。Yuan等[34]在研究中发现,过表达VaPAT1导致转基因拟南芥脯氨酸和可溶性糖含量的增加,是转基因拟南芥抗寒、抗旱、耐盐能力增强的两个重要因素[34]。Zhang等[35]在研究中发现,在拟南芥中异源过表达HcSCL13能通过正向调控过氧化氢酶活性以及可溶性蛋白、可溶性糖的含量来降低植物体内ROS的累积,从而使其具有更好的耐盐胁迫能力。这些结果与本研究中过表达BpPAT1基因能够提高白桦耐盐能力的结果相一致。本研究发现过表达BpPAT1基因能显著提高白桦植株的耐盐性,结合植物GRAS转录因子的研究现状,后续将进一步对BpPAT1调控的靶基因及靶基因的抗逆生理学途径进行系统深入的研究,为阐明白桦GRAS转录因子的抗逆分子机制奠定理论基础。
4. 结 论
本研究从盐胁迫白桦转录组数据中获得一条GRAS家族基因BpPAT1。该基因在白桦根、茎和叶组织中均有表达,且能响应盐胁迫。过表达BpPAT1显著提高了盐胁迫下白桦植株中POD及SOD酶的活性,从而增强了白桦的ROS清除能力;同时显著提高了盐胁迫下白桦植株的脯氨酸含量,降低了电解质渗透率及丙二醛含量,从而有效减轻植物组织细胞的受损程度。综上所述,BpPAT1基因参与白桦对盐胁迫的响应,过表达BpPAT1基因显著提高了白桦植株的耐盐能力。
-
图 1 白桦BpPAT1蛋白的多序列比对分析(A)及系统进化树分析(B)
AtPAT1. 拟南芥(NP_001332482.1);QsGRAS. 欧洲栓皮栎(XP_023916980.1);MrGRAS. 杨梅(KAB1208676.1);JrGRAS. 胡桃(XP_018849898.1);VvGRAS. 葡萄(XP_002272334.1);JcGRAS20. 麻风树(XP_012081428.1);CfGRAS. 土瓶草(GAV74587.1);DlGRAS54. 龙眼(AGE44291.1);TcGRAS. 可可(EOX93442.1)。AtPAT1, Arabidopsis thaliana (NP_001332482.1); QsGRAS, Quercus suber (XP_023916980.1); MrGRAS, Morella rubra (KAB1208676.1); JrGRAS, Juglans regia (XP_018849898.1);VvGRAS, Vitis vinifera (XP_002272334.1); JcGRAS20, Jatropha curcas (XP_012081428.1); CfGRAS, Cephalotus follicularis (GAV74587.1); DlGRAS54, Dimocarpus longan (AGE44291.1); TcGRAS, Theobroma cacao (EOX93442.1).
Figure 1. Multiple sequence alignment analysis of Betula platyphylla BpPAT1 protein (A) and phylogenetic tree analysis (B)
图 3 盐胁迫下BpPAT1基因在瞬时表达白桦植株中的表达情况
A. 盐胁迫下不同转化时间对照植株及瞬时过表达植株BpPAT1基因的表达水平;B. 正常条件下与盐胁迫后对照植株及瞬时转化植株BpPAT1基因的表达水平;Control. 对照植株;OE. 瞬时过表达植株;IE. 瞬时抑制表达植株;*. 显著性差异(P < 0.05)。下同。A, expression levels of BpPAT1 gene in control plants and transient overexpressed plants at different transformation time under salt stress; B, expression level of BpPAT1 gene in control plants and transient transformed plants under normal conditions and salt stress. Control, control plant; OE, transient overexpression plant; IE, transient inhibitory expression plant. Asterisks indicate significant difference (P < 0.05). The same below.
Figure 3. Expression of BpPAT1 gene in transient expression plants of B. platyphylla under salt stress
表 1 引物序列
Table 1 Primers used in this study
用途 Application 引物名称 Primer name 引物序列(5′—3′) Primer sequence (5′−3′) 实时荧光定量 PCR Quantitative real-time PCR q-BpPAT1-F TACTGCTGCATTCTATCCAC q-BpPAT1-R ACTTACCAAGAAGCTCATG q-BpPAT1-OE-F CCCCACATCCGCATAACA q-BpPAT1-OE-R CCCAGGTCGAATCCCAAG q-BpPAT1-IE-F ACGAACGGTGTTGCACTT q-BpPAT1-IE-R GAGCCATAGGTATTGTCAGG Actin-F TGAGAAGAGCTATGAGTTGC Actin-R GTAGATCCACCACTAAGCAC Tubulin-F TCAACCGCCTTGTCTCTCAGG Tubulin-R TGGCTCGAATGCACTGTTGG 基因克隆 Gene cloning BpPAT1-F GCTCTAGAATGTCCAACGGATTGTACTATC BpPAT1-R GGGTACCTCACTTCCATGCACAAGCAG 载体构建 Vector verification pROKⅡ-F AGACGTTCCAACCACGTCTT pROKⅡ-R CCAGTGAATTCCCGATCTAG pFGC5941-cisF CGCTCGAGTATAAGAGCT pFGC5941-cisR ACCTTCCCACAATTCGTCGG pFGC5941-antiF GCATGCTATGCATTCAAT pFGC5941-antiR CGTGCACAACAGAATTGAAAGC pFGC5941-BpPAT1-cisF CCCATGGCAGCTATGCTACAATGATAG pFGC5941-BpPAT1-cisR TTGGCGCGCCTCCACATATAGAAGAGCCAT pFGC5941-BpPAT1-antiF CTCTAGACAGCTATGCTACAATGATAG pFGC5941-BpPAT1-antiR CGGATCCTCCACATATAGAAGAGCCAT -
[1] 刘中原, 刘峥, 徐颖, 等. 白桦HSFA4转录因子的克隆及耐盐功能分析[J]. 林业科学, 2020, 56(5):69−79. doi: 10.11707/j.1001-7488.20200508 Liu Z Y, Liu Z, Xu Y, et al. Cloning and salt tolerance analysis of transcription factor HSFA4 from Betula platyphylla[J]. Scientia Silvae Sinicae, 2020, 56(5): 69−79. doi: 10.11707/j.1001-7488.20200508
[2] 刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用[J]. 科学通报, 2000, 45(14):1465−1474. doi: 10.3321/j.issn:0023-074X.2000.14.002 Liu Q, Zhang G Y, Chen S Y. The structure and regulation of plant transcription factors[J]. Chinese Science Bulletin, 2000, 45(14): 1465−1474. doi: 10.3321/j.issn:0023-074X.2000.14.002
[3] 唐瑞, 韩妮, 虎亚静, 等. 黄瓜GRAS家族全基因组鉴定与表达分析[J/OL]. 分子植物育种, 2021, 19(13): 4242−4251 [2020−07−23]. http://kns.cnki.net/kcms/detail/46.1068.S.20200526.1639.012.html. Tang R, Han N, Hu Y J, et al. Genome-wide identification and expression analysis of GRAS genes in Cucumber[J/OL]. Molecular Plant Breeding, 2021, 19(13): 4242−4251 [2020−07−23]. http://kns.cnki.net/kcms/detail/46.1068.S.20200526.1639.012.html.
[4] Lee M H, Kim B, Song S K, et al. Large-scale analysis of the GRAS gene family in Arabidopsis thaliana[J]. Plant Molecular Biology, 2008, 67(6): 659−670. doi: 10.1007/s11103-008-9345-1
[5] Tian C, Wan P, Sun S, et al. Genome-wide analysis of the GRAS gene family in rice and Arabidopsis[J]. Plant Molecular Biology, 2004, 54(4): 519−532. doi: 10.1023/B:PLAN.0000038256.89809.57
[6] Song X M, Liu T K, Duan W K, et al. Genome-wide analysis of the GRAS gene family in Chinese cabbage (Brassica rapa ssp. pekinensis)[J]. Genomics, 2013, 103(1): 135−146.
[7] Cordelia B. The role of GRAS proteins in plant signal transduction and development[J]. Planta, 2004, 218(5): 683−692. doi: 10.1007/s00425-004-1203-z
[8] Liu Y D, Huang W, Xian Z Q, et al. Overexpression of SlGRAS40 in tomato enhances tolerance to abiotic stresses and influences auxin and gibberellin signaling[J/OL]. Frontiers in Plant Science, 2017, 8: 1659 [2020−09−23]. https://doi.org/10.3389/fpls.2017.01659.
[9] Kim Y J, Yang D H, Park M Y, et al. Overexpression of zoysia ZjCIGR1 gene confers cold stress resistance to zoysiagrass[J]. Plant Biotechnology Reports, 2020, 14(1): 21−31. doi: 10.1007/s11816-019-00570-z
[10] Laura D L, Joanna W D, Jocelyn E M, et al. The SCARECROW gene regulates an asymmetric cell division that is essential for generating the radial organization of the Arabidopsis root[J]. Cell, 1996, 86(3): 423. doi: 10.1016/S0092-8674(00)80115-4
[11] Helariutta Y, Fukaki H, Wysocka D J, et al. The SHORT-ROOT gene controls radial patterning of the Arabidopsis root through radial signaling[J]. Cell, 2000, 101(5): 555−567. doi: 10.1016/S0092-8674(00)80865-X
[12] Chen K M, Li H W, Chen Y F, et al. TaSCL14, a novel wheat (Triticum aestivum L.) GRAS gene, regulates plant growth, photosynthesis, tolerance to photooxidative stress, and senescence[J]. Journal of Genetics and Genomics, 2015, 42(1): 21−32. doi: 10.1016/j.jgg.2014.11.002
[13] Torres-Galea P, Huang L F, Chua N H, et al. The GRAS protein SCL13 is a positive regulator of phytochrome-dependent red light signaling, but can also modulate phytochrome a responses[J]. Molecular Genetics & Genomics, 2006, 276(1): 13−30.
[14] 吴捷, 兰士波, 宁晓光. 东北白桦种质资源生态耦合性分析及可持续利用策略[J]. 林业勘查设计, 2017(4):62−64. doi: 10.3969/j.issn.1673-4505.2017.04.029 Wu J, Lan S B, Ning X G. Ecology coupling analysis and sustainable utilization strategy of Betula platyphylla germplasm resource[J]. Forest Investigation Design, 2017(4): 62−64. doi: 10.3969/j.issn.1673-4505.2017.04.029
[15] Liu X, Widmer A. Genome-wide comparative analysis of the GRAS gene family in Populus, Arabidopsis and rice[J]. Plant Molecular Biology Reporter, 2014, 32(6): 1129−1145. doi: 10.1007/s11105-014-0721-5
[16] Li Z, Lu H, He Z, et al. Selection of appropriate reference genes for quantitative real-time reverse transcription PCR in Betula platyphylla under salt and osmotic stress conditions[J/OL]. PLoS One, 2019, 14(12): e0225926 [2020−08−03]. https://doi.org/10.1371/journal.pone.0225926.
[17] Livak K, Schmittgen T. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCt method[J]. Methods, 2001, 25(4): 402−408. doi: 10.1006/meth.2001.1262
[18] 郭勇, 王玉成, 王智博. 一种基于农杆菌介导的拟南芥瞬时转化技术优化[J]. 东北林业大学学报, 2016, 44(6):41−44, 83. doi: 10.3969/j.issn.1000-5382.2016.06.011 Guo Y, Wang Y C, Wang Z B. Optimizing transient genetic transformation method on Arabidopsis plants mediated by Agrobacterium tumefaciens[J]. Journal of Northeast Forestry University, 2016, 44(6): 41−44, 83. doi: 10.3969/j.issn.1000-5382.2016.06.011
[19] 王关林, 方宏筠. 植物基因工程实验技术指南[M]. 北京: 科学出版社, 2016. Wang G L, Fang H J. Laboratory guide for plant genetic engineering[M]. Beijing: Science Press, 2016.
[20] 聂显光. 柽柳ThbHLH1基因调控抗逆响应的分子机理研究[D]. 哈尔滨: 东北林业大学, 2014. Nie X G. Functional characterization of the abiotic stress response mechanisms of ThbHLH1 transcript factor from Tamarix hispida[D]. Harbin: Northeast Forestry University, 2014.
[21] 卢惠君, 李子义, 梁瀚予, 等. 刚毛柽柳NAC24基因的表达及抗逆功能分析[J]. 林业科学, 2019, 55(3): 54−63. Lu H J, Li Z Y, Liang H Y, et al. Expression and stress tolerance analysis of NAC24 from Tamarix hispida[J]. Scientia Silvae Sinicae, 2019, 55(3): 54−63.
[22] 刘羽佳. 拟南芥AtbHLH112基因调控植物抗逆机制的研究[D]. 哈尔滨: 东北林业大学, 2013. Liu Y J. Study on stress tolerance mechanism mediated by AtbHLH112 from Arabidopsis thaliana[D]. Harbin: Northeast Forestry University, 2013.
[23] 国会艳. 白桦BplMYB46基因调控抗旱耐盐和次生壁形成的分子机理[D]. 哈尔滨: 东北林业大学, 2014. Guo H Y. The molecular mechanism of BplMYB46 from Betula platyphylla in mediating drought and salt tolerance and formation of secondary wall[D]. Harbin: Northeast Forestry University, 2014.
[24] Lu X, Liu W, Xiang C, et al. Genome-wide characterization of GRAS family and their potential roles in cold tolerance of Cucumber (Cucumis sativus L.)[J/OL]. International Journal of Molecular Sciences, 2020, 21(11): 3857 [2020−08−29]. https://doi.org/10.3390/ijms21113857.
[25] Sidhu N S, Pruthi G, Singh S, et al. Genome-wide identification and analysis of GRAS transcription factors in the bottle gourd genome[J]. Scientific Reports, 2020, 10(1): 2367−2372. doi: 10.1038/s41598-020-59417-1
[26] Ma H S, Liang D, Shuai P, et al. The salt- and drought-inducible poplar GRAS protein SCL7 confers salt and drought tolerance in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2010, 61(14): 4011−4019. doi: 10.1093/jxb/erq217
[27] 周莲洁, 杨中敏, 张富春, 等. 新疆盐穗木GRAS转录因子基因克隆及表达分析[J]. 西北植物学报, 2013, 33(6):1091−1097. doi: 10.3969/j.issn.1000-4025.2013.06.004 Zhou L J, Yang Z M, Zhang F C, et al. Expression analysis and cloning of GRAS transcription factor gene from Halostachys capsica[J]. Acta Botanica Boreali-Occidentalia Sinica, 2013, 33(6): 1091−1097. doi: 10.3969/j.issn.1000-4025.2013.06.004
[28] Liu Z Y, Wang P L, Zhang T Q, et al. Comprehensive analysis of BpHSP genes and their expression under heat stresses in Betula platyphylla[J]. Environmental and Experimental Botany, 2018, 152: 167−176. doi: 10.1016/j.envexpbot.2018.04.011
[29] Zang D D, Wang C, Ji X Y, et al. Tamarix hispida zinc finger protein ThZFP1 participates in salt and osmotic stress tolerance by increasing proline content and SOD and POD activities[J]. Plant Science, 2015, 235: 111−121. doi: 10.1016/j.plantsci.2015.02.016
[30] Yang G Y, Yu L L, Zhang K M, et al. A ThDREB gene from Tamarix hispida improved the salt and drought tolerance of transgenic tobacco and T. hispida[J]. Plant Physiology & Biochemistry, 2017, 113: 187−197.
[31] Li P, Zhang B, Su T B, et al. BrLAS, a GRAS transcription factor from Brassica rapa, is involved in drought stress tolerance in transgenic Arabidopsis[J/OL]. Frontiers in Plant Science, 2018(9): 1792 [2020−08−06]. https://doi.org/10.3389/fpls.2018.01792.
[32] Guo H Y, Wang Y C, Wang L Q, et al. Expression of the MYB transcription factor gene BplMYB46 affects abiotic stress tolerance and secondary cell wall deposition in Betula platyphylla[J]. Plant Biotechnology Journal, 2017, 15(1): 107−121. doi: 10.1111/pbi.12595
[33] He Z H, Li Z Y, Lu H J, et al. The NAC protein from Tamarix hispida, ThNAC7, confers salt and osmotic stress tolerance by increasing reactive oxygen species scavenging capability[J/OL]. Plants, 2019, 8(7): 221 [2020−07−12]. https://doi.org/10.3390/plants8070221.
[34] Yuan Y Y, Fang L C, Sospeter K K, et al. Overexpression of VaPAT1, a GRAS transcription factor from Vitis amurensis, confers abiotic stress tolerance in Arabidopsis[J]. Plant Cell Reports, 2016, 35(3): 655−666. doi: 10.1007/s00299-015-1910-x
[35] Zhang S, Li X, Fan S, et al. Overexpression of HcSCL13, a Halostachys caspica GRAS transcription factor, enhances plant growth and salt stress tolerance in transgenic Arabidopsis[J]. Plant Physiology and Biochemistry, 2020, 151: 243−254. doi: 10.1016/j.plaphy.2020.03.020
-
期刊类型引用(7)
1. 丁金华,许艳秋,钱晶. 苏南水网地区水域景观破碎化时空演变特征及驱动因子研究——以吴江区为例. 西北林学院学报. 2024(01): 247-255 .  百度学术
百度学术
2. 陈实,陈丽捷,洪宇,刘金福,阙翔,李意敏,何东进,赵婧雯. 泉州湾湿地碳库生态安全评价及其障碍因素研究. 福建农林大学学报(自然科学版). 2024(05): 686-695 .  百度学术
百度学术
3. 杨烜涵,付晖,秦煜姬,程恩起,陈圣天. 近20年来海口湿地景观格局演变及其驱动因子. 中国城市林业. 2023(03): 28-35 .  百度学术
百度学术
4. 王琦,刘子刚,周隽伊. 三江平原沼泽湿地变化的影响因素及其空间效应. 中国人口·资源与环境. 2023(07): 44-54 .  百度学术
百度学术
5. 赵红梅,毛欣,刘春雷,李亚松,刘林敬. 福建泉州湾海岸带MIS 3阶段以来的海侵—海退过程. 地质力学学报. 2023(04): 569-583 .  百度学术
百度学术
6. 朱映辰,谭芳林,阙翔,洪宇,潘爱芳,刘金福. 多时间尺度下森林公园负离子变化特征及与温湿度关系研究. 西北林学院学报. 2023(06): 211-218+227 .  百度学术
百度学术
7. 赵红梅,刘春雷,毛欣,毕志伟,刘哲,李亚松. 泉州湾海岸带全新世地层及沉积环境演化. 地层学杂志. 2022(04): 401-410 .  百度学术
百度学术
其他类型引用(5)



 下载:
下载: