Development and identification of SSR markers based on RAD-seq of Lonicera japonica
-
摘要:目的 通过开发特异性高的多态性分子标记位点,以弥补金银花在简单重复序列(SSR)标记方面的匮乏,推动金银花在遗传资源管理及良种鉴定等方向的研究,为后续全基因组测序及组装策略奠定基础。方法 运用RAD-seq技术对2份金银花样品进行简化基因组测序,利用MISA软件识别contig上的SSR序列并对各类型序列特征进行归纳分析,进而设计引物并利用生物信息学的方法进行引物筛选和有效性检测。结果 对‘九丰一号’和‘亚特’金银花进行简化基因组测序,分别获得27.805 Mbp和42.560 Mbp干净读长。在组装的45 850个基因序列中共检测到46 999个SSR位点,其中单核苷酸重复单元比例最高(49.15%),六核苷酸重复单元最少(0.20%)。SSR位点的重复基序以(A/T)n为主,具有偏向性。除单碱基和二碱基重复类型外,SSR位点基序的重复次数集中在5 ~ 6次,且随重复次数增加,SSR位点重复类型出现的频率呈下降趋势。金银花基因组SSR序列长度介于10 ~ 310 bp之间,随重复次数增加,各SSR序列出现的频率逐渐下降。利用Primer3.0成功设计出38 507对SSR引物,设计成功率为81.93%。对挑选的35对SSR引物进行多态性分析,等位基因数、观测杂合度和多态性信息含量(PIC)的平均值分别为5.057、0.363和0.568,其中高多态信息位点26个,中度多态信息位点9个,均未偏离哈迪–温伯格平衡。结论 利用RAD-seq技术可实现SSR标记的大量开发和多态性SSR引物的筛选,并为金银花在遗传多样性分析和种质鉴定等相关方面的研究提供数据支持。
-
关键词:
- 金银花 /
- 简单重复序列(SSR) /
- RAD-seq /
- 多态性
Abstract:Objective Through the development of polymorphic molecular markers to make up for the lack of Lonicera japonica in SSR markers, this paper aims to promote the research in genetic resource management and variety identification, and lay the foundation for future de novo assembling of Lonicera japonica genome.Method RAD-seq (restriction-site associated DNA sequencing) was used to conduct simplified genome sequencing for two Lonicera japonica samples. The SSR sequences were identified by MISA and the characterizations of SSR sequences and their polymorphisms were analyzed by bioinformatics methods.Result 27.805 Mbp and 42.560 Mbp clean reads were obtained in ‘Jiufengyihao’ and ‘Yate’ using restriction-site associated DNA (RAD). A total of 46 999 SSR loci were detected among the 45 850 gene sequences assembled, with the highest proportion of mono-nucleotide repeat SSRs (49.15%) and the lowest proportion of hexa-nucleotide repeat SSRs (0.20%). The repeated motifs at SSR loci were dominated by (A/T)n and showed a bias. Apart from mono-nucleotide and di-nucleotide repetition types, the repeat counts of SSR motifs mainly ranged from 5 to 6. The frequency of SSRs repetition types showed a downward trend with the increase of repeat number. The range of SSR length ranged from 10 to 310 bp, and the frequency of SSR sequence tended to decrease as the number of repetitions increasing. 38 507 pairs of SSR primers were successfully designed, with a design success rate of 81.93%. 35 pairs of SSR primers were analyzed for polymorphism, in which the average number of alleles, observed heterozygosity and PIC were 5.057, 0.363 and 0.568, among which there were 26 highly polymorphic loci and 9 moderately polymorphic loci, all of which did not deviate from the Hardy-Weinberg equilibrium.Conclusion The large-scale development of SSR markers and the screening of polymorphic SSR primers can be realized based on RAD-seq technology, and provide data support for the research of Lonicera japonica in genetic diversity analysis and germplasm identification.-
Keywords:
- Lonicera japonica /
- simple sequence repeat (SSR) /
- RAD-seq /
- polymorphism
-
工业革命以来,随着全球变化的加剧,氮沉降的增加和降水格局的改变对陆地生态系统的影响也日益突出。过量的氮沉降会对植物生长和土壤性质产生明显的负作用[1-3],例如促使NH4+硝化、NO3-淋失、土壤酸化[4-6]和系统养分平衡失调[2]等,最终扰乱森林的正常结构和功能,甚至减少生态系统的生产力[7-8]和生物多样性[3];降水格局的变化则会直接影响土壤含水量,进而改变土壤温度和养分等理化性质,最终影响植物的形态结构和生理特性[9]。这些都会对森林生态系统冠层生产产生影响。森林凋落物是指在森林生态系统内,由地上植被生产又归还到地面并最终输入到土壤中的、能够维持森林生态系统正常功能的有机物的总称[10-11]。凋落物作为森林碳库的重要组成部分,含有大量的营养元素和有机物质,其分解和释放直接关系到土壤养分含量;地上部分的生物量主要通过凋落物的形式返还到地面,这部分生产量(凋落量)最高可占森林生态系统净积累量的30%[12],而且在调节森林生态系统能量流动和碳循环过程中扮演着重要角色[13-17]。由全球变化引发的氮沉降和降水减少对森林凋落物及其组分的影响越来越受到学者们的关注。目前,国内外对森林凋落物的研究有年凋落量、凋落物的组成、季节动态[18]及分解等[19-21]。其中,国内在森林凋落物总量及其各个组分变化对氮沉降[22-24]和降水格局变化[25-26]的响应方面有一定的研究,但是,与国外相比还存在很大差距,多数研究的施氮和降水控制时间不长,难以对长期的影响进行预测,主要问题在于缺乏凋落物量对长期氮-水控制响应的机理方面的研究;另外,多数研究主要集中在凋落物中枝和叶的沉积量(现存量)[17, 27-28],而将凋落物组分中的木质碎屑(花、果、皮)作为凋落物主要成分的研究相对较少,这些组分都可能对凋落物总量有较大的贡献度[29-30];而且,多数研究都只围绕着凋落物产量进行展开,对于如何消除林分组成等影响没有太深入的研究。因此开展长期氮添加和降水格局变化对森林凋落物量影响机制的研究十分必要。
阔叶红松(Pinus koraiensis)林是我国北方典型的地带性植被类型,是东北地区结构最复杂、物种最丰富的顶级森林群落[22],其凋落物动态与阔叶落叶林和针叶林相比,具备二者的共同特点,即落叶阔叶林的凋落期主要集中在秋季,针叶林则是全年都在生产凋落物[31]。本研究主要通过长期施氮和减少降水两种控制条件,结合胸高断面积对凋落物量进行单位化,以期消除林分自身差异产生的影响,来探求阔叶红松林凋落物总量及其各组分对长期氮水变化的响应机理,为了解全球变化条件下氮沉降和降水减少对森林凋落物量的影响提供参考。
1. 研究方法
1.1 研究地概况
长白山自然保护区位于吉林省东南部(41°41′49″~42°25′18″N,127°42′55″~128°16′48″E),属于典型的温带大陆性季风气候,具有显著的中纬度山地气候特征,春季干旱多风,夏季炎热多雨,秋季晴朗少雨,冬季干燥寒冷,年平均气温3.3~6.1 ℃,8月气温最高(平均20.6 ℃),1月气温最低(平均-16.5 ℃),年平均降水量600~900 mm,主要集中在6—8月,全年日照时数为2 271~2 503 h,无霜期109~141 d。土壤为山地暗棕色森林土。主要乔木树种有红松、蒙古栎(Quercus mongolica)、水曲柳(Fraxinus mandschurica)、紫椴(Tilia amurensis)、胡桃楸(Juglans mandshurica)、色木槭(Acer mono)等;灌木有毛榛(Corylus mandshurica)、东北山梅花(Philadelphus schrenkii)、东北茶藨子(Ribes mandshuricum),长白茶藨子(Ribes komarovii)等;草本有龙牙草(Agrimonia pilosa)、水金凤(Impatiens noli-tangere)、山茄子(Lonicera caerules)。生长季为4—9月份。
1.2 实验设计
在中国科学院长白山森林生态系统野外定位站的径流场(42°24′18″N,128°6′19″E)样地内,选取施氮(NA)样地3块,减水施氮(RN)样地3块,对照(CK)样地2块,在附近额外设置1块对照样地,样地大小均为25 m×50 m,样地之间间隔10 m以上。施氮样地年施氮量为50 kg/hm2,在每年5—10月份的月初(2009年开始),将氮肥(主要成分为NH4NO3)溶解在40 L水中后,以肩背式喷雾器在样地内来回均匀喷洒,以避免因水分不同而导致的差异,在对照样地喷洒等量的水;减水样地为减少30%穿透雨,选用透光性良好的聚碳酸酯材料(透光性95%)制作成V字型截雨板,用钢管架支撑,距离地面高约1 m,截雨板的有效截雨面积占样地的30%[32]。
在2015年秋季生长季结束之后,对不同处理的9块样地内的林分状况进行本底调查(每木检尺)和土壤指标测定,如表 1。
表 1 各样地基本概况Table 1. Basic situation of sample sites处理
Treatment林分特征Stand characteristics 土壤氮素含量Soil nitrogen (N) content 每公顷株数
Tree number per hectare胸高断面积
Basal area at breast height/m2全氮
Total N/%铵态氮
Ammonium N/(mg·kg-1)硝态氮
Nitrate N/(mg·kg-1)对照Control(CK) 312±105.44 10.80±0.80 1.18±0.08 17.95±1.82 19.02±1.07 施氮N addition(NA) 373±63.79 11.15±1.12 1.25±0.10 17.77±0.94 20.85±1.05 减水施氮Reduced precipitation and N addition(RN) 285±84.51 8.91±1.33 1.14±0.09 19.29±1.42 21.13±1.52 凋落物的收集采用直接收集法。在每种不同处理的样地中随机布置10个1 m×1 m的收集器,每个收集器距离地面20 cm。从2015年6—10月,每个月收集1次。收集后将凋落物分成红松、蒙古栎、其他树种、枝和花果皮屑等5个组分,于65 ℃条件下烘干24 h至恒质量后称质量。由于各样地树种组成的差异,在计算每公顷各组分凋落物量及其总凋落量时,结合每个样地内各树种的胸高断面积,计算出单位胸高断面下各组分凋落物的量[33-34]。当凋落物量用胸高断面积单位化之后,其代表的生物学含义就发生了变化,此时不再代表凋落物量的多少,而是代表单位生物量生产凋落物能力的大小,在本研究中称之为凋落系数(T),计算公式如下:
林分凋落系数:
T=∑M/∑S 式中:∑M为总凋落物质量;∑S为总胸高断面积。
各组分凋落系数由各自凋落物量除以相应组分的胸高断面积,其中,枝和花果皮屑未区分树种,以总胸高断面积计算。
1.3 数据处理
统计分析采用Excel 2007和SPSS 21.0软件进行,作图采用SigmaPlot 10.0。对各组分凋落物量和相应胸高断面积进行Pearson相关性分析(P<0.05),并采用双因素方差分析检验不同处理各组分凋落物量、凋落系数的显著性(P<0.05)。
2. 结果与分析
2.1 不同处理样地凋落物的组成
本研究将森林凋落物分为红松叶、蒙古栎叶、其他树叶、花果皮屑、枝5部分,并对3种不同处理的凋落物各组分的量进行了统计,得到了各组分生长季内的凋落量的平均值,如表 2。
表 2 不同处理各组分凋落量及占总量的百分比Table 2. Litter mass of components(±STD)in different treatments and percentage in total处理
Treatment凋落物组分/(t·hm-2) Litter component/(t·ha-1) 总量Total 红松叶
Pinus koraiensis leaf(PK)蒙古栎叶
Quercus mongolica leaf(QM)其他叶
Other broadleaf(OT)枝
Twig(TW)花果皮屑
Reproductive organics and bark(ROB)CK 0.75±0.25(27%) 0.32±0.12(12%) 1.12±0.12(41%) 0.21±0.05(8%) 0.34±0.07(12%) 2.74±0.38(100%) NA 0.43±0.05(16%) 0.18±0.05(6%) 1.56±0.13(57%) 0.24±0.02(9%) 0.35±0.06(13%) 2.75±0.21(100%) RN 0.47±0.21(18%) 0.30±0.12(12%) 1.30±0.03(51%) 0.25±0.07(10%) 0.23±0.01(9%) 2.55±0.38(100%) 注:括号内为该组分占总量的百分比。Note: data in brackets represent the component percentage in total. 各样地凋落物总量平均值在2.55~2.75 t/hm2之间。叶凋落物量所占比重较大,可达到79%~81%,而枝和花果皮屑凋落物量所占比重相对较小,占到总量19%~21%;施氮样地红松、蒙古栎和其他树种所占凋落物量(16%,6%和57%)比较对照样地(红松27%,蒙古栎占12%,其他占41%)有较大差异,减水施氮地中分别占18%,12%和51%;在对照地和施氮地中,花果皮屑所占凋落物量的比例(分别为12%和13%)都高于枝的(分别为8%和9%),而减水施氮地中花果皮屑所占比例(9%)低于枝的(10%)。
方差分析显示:各组分凋落物量中,仅其他叶凋落物量在不同处理中差异显著(P=0.016),其余均未达到显著水平。
2.2 不同处理凋落物月动态
由图 1可以看出,各处理月凋落动态为单峰型,各组分凋落量的峰值主要集中在9—10月份,最小值主要集中在7—8月份,其中红松和枝两部分6月份的量略高于7—8月份,但是相对于9—10月的凋落物量较小。
图 2可看出,不同处理各组分具有不同的月凋落节律。月变化最大的是叶凋落物量,枝和花果皮屑的变化平缓,每个月差异不大。
在对照处理中,6月份各组分凋落物量以红松叶为最多,其次是花果皮屑、枝和其他叶,最少为蒙古栎叶;7—8月份变化规律不明显,但这两个月对应的组分之和基本呈花果皮屑>其他叶>红松叶>枝>蒙古栎叶;9月份的凋落物量以其他叶的为最大,其次是红松叶、花果皮和枝,蒙古栎叶最少;而10月份凋落物量最大值为其他叶,其次仍是红松叶、蒙古栎和枝,最少为花果皮。
在施氮处理中,6月份各组分凋落物量最多为花果皮屑,其次是红松叶,枝、其他叶和蒙古栎同对照处理;7—8月份的变化规律较明显,均为为花果皮屑>其他叶>枝>红松叶>蒙古栎叶;9月份凋落物量最多的为其他叶,其次为花果皮屑、红松叶和枝,以蒙古栎叶最少;10月份的变化规律与对照地的相同。
在减水施氮处理中,6月份各组分凋落物量最多为红松叶,其次是枝、花果皮屑和其他叶,蒙古栎叶最少;7—8月份变化规律与施氮处理相同;9月份凋落物量最多仍为为其他叶,其次是蒙古栎叶、红松叶、枝,最少为花果皮屑;10月份的变化规律与前两种处理的相同。
方差分析显示:各月凋落物组分中,6—7月各处理间,红松叶(分别为P=0.001和P=0.018)、蒙古栎叶(分别为P=0.010和P=0.030)、其他叶(分别为P=0.015和0.024)和花果皮屑(分别为P=0.020和P=0.020)差异显著;8月各处理间仅花果皮(P=0.004)差异显著;9月各处理间仅蒙古栎叶(P=0.040)差异显著;10月各处理间,红松叶(P=0.007)和其他叶(P=0.012)差异显著;各月份中,枝处理间差异均不显著。
2.3 不同处理各凋落物量与胸高断面积的相关性
对不同处理各凋落物组分与对应的胸高断面积进行相关性分析,结果如表 3。
表 3 各组分凋落物量与胸高断面积相关性分析Table 3. Correlation analysis of component litterfall mass and basal area at breast height参数
Parameter处理
Treatment凋落物组分Litterfall component 叶总量
Leaf litterfall amount总量
Total amountPK QM OT TW ROB 胸高断面积
Basal area at breast heightCK 0.759 0.994 0.996* 0.778 0.797 0.790 0.654 NA 0.978 0.999* 0.990 0.872 0.932 0.863 0.921 RN 0.869 0.742 0.988 0.987 0.963 0.953 0.959 注:*表示在P<0.05上显著相关。Note: * represents significant correlation at P<0.05 level. 由表 3可知,各组分凋落物量与胸高断面积均呈正相关,总体呈现叶的相关性较好,但不同处理间变化规律不同。在对照处理中,其他叶相关系数最大(0.996),且相关性显著,其次为蒙古栎叶(0.994),但未达到显著水平,总凋落物量的相关性最小(0.654);在施氮处理中,蒙古栎叶的相关系数最大,且显著正相关,其次为其他叶(0.990)、红松叶(0.978)、花果皮屑(0.932)和总凋落量(0.921),但均未达到显著水平,最小值为叶总量(0.863);在减水施氮处理中,相关系数从大到小依次为其他叶(0.988)、枝(0.987)、花果皮屑(0.963)、总量(0.959)、叶总量(0.953)、红松叶(0.869)、蒙古栎叶(0.742),但均未达到显著水平。
2.4 不同处理各组分凋落系数
双因素方差分析显示(表 4),不同组分凋落物系数对长期氮水控制响应的差异各不相同。施氮显著降低了红松叶的凋落系数,而减水施氮也降低了红松叶的凋落系数,但不显著,其中,降减水少对凋落系数有一定的促进作用,但效果不显著;施氮和减水施氮都提高了蒙古栎和其他树种的凋落物系数,但减水施氮对其他叶凋落物系数的影响显著;施氮和降水减少对枝和花果皮的凋落系数均无显著影响;施氮会降低叶凋落系数和林分凋落系数,降水减少提高了叶凋落系数和林分凋落系数,其中对叶总量影响显著。总体表现为氮添加会抑制针叶树种的凋落物量,促进阔叶树种的凋落物量,降水减少会促进各组分凋落物量。
表 4 不同处理各组分的凋落系数Table 4. Litterfall coefficient of components in different treatments处理
Treatment凋落物组分Litter component/(t·m-2) 叶凋落物系数
Leaf litterfall coefficient (LC)林分凋落系数
Forest litterfall coefficient (FC)PK QM OT TW ROB CK 0.200 a(0.049) 0.124 a(0.040) 0.212 b(0.013) 0.019 a(0.004) 0.031 a(0.005) 0.230 ab(0.020) 0.253 a(0.027) NA 0.115 b(0.014) 0.172 ab(0.024) 0.261 ab(0.003) 0.022 a(0.004) 0.031 a(0.005) 0.194 b(0.018) 0.247 a(0.007) RN 0.149 ab(0.018) 0.251 a(0.008) 0.305 a(0.005) 0.027 a(0.000) 0.026 a(0.000) 0.233 a(0.018) 0.286 a(0.011) 注:括号内为标准差,a、b代表各组分不同处理中差异显著(P<0.05)。Notes: Standard deviations are shown in brackets, a, b represent significant difference in each treatment among these components (P<0.05). 同一处理下各组分凋落系数也不相同,但是,在各处理中其他的凋落系数都高于其余各组分的。在对照地和施氮地中凋落系数最小的是枝(分别为0.019和0.022 t/m2),在减水施氮地中最小值为花果皮屑(0.026 t/m2)。
3. 讨论
3.1 凋落总量与同地区类似研究比较
森林凋落物量是衡量生态系统初级生产力的重要指标,是地上有机产物向地下转移、输入的重要环节。森林凋落量主要受气候、林龄和森林类型的影响,并且在年际间的差异很大[35-39]。Bray等[40]人发现凉温带、暖温带的年凋落物量分别为3.1 t/a和4.9 t/a。韩士杰等[41]发现小兴安岭中南部凉水国家级自然保护区阔叶红松林的年凋落量为3.49 t/(hm2·a);原作强等[33]研究表明,长白山阔叶红松林年凋落量为3.92 t/(hm2·a);Li等[42]对长白山阔叶红松林过去19年的凋落量进行了研究,发现其年均凋落量为4.02 t/(hm2·a);陈金玲[22]在研究阔叶红松林对对模拟氮沉降响应中得出,不同施氮处理下凋落物总量范围在3.69~4.36 t/(hm2·a)。本研究通过3种不同处理(CK、NA、RN)样地得到,生长季内凋落物总量的平均值分别为对照地(2.74 t/hm2),施氮地(2.75 t/hm2),减水施氮地(2.55 t/hm2)。另外,与上述研究结果相比,本研究的年凋落量低了1/3左右,刘颖等[43]研究表明,长白山阔叶红松林在非生长季内(10月至翌年5月)凋落物中以枝最多,占凋落总量的1/3之多,这使得此差异得到了解释。
3.2 凋落物凋落节律比较
森林凋落量具有明显的季节变化规律,其季节动态模式主要分为单峰型,双峰型或不规则型[10]。影响森林凋落物凋落节律的主要因素为纬度、海拔差异下的降水和温度变化,土壤肥力,养分输入等[40, 43-48]。在本研究中,不同处理总凋落量和各组分(不包括花果皮屑)凋落量在生长季内凋落动态为单峰型,峰值都出现在9—10月份,最小值出现在夏季(7—8月份),这是因为生长季时树木需要吸收大量的养分、水分来维持自身的生长,其主要动力是冠层的蒸腾作用,故落叶较少,而生长季结束后,随着气温降低、降水减少,为降低养分、水分的消耗,形成大量生理性的落叶[33]。在减水施氮处理中,9月凋落量明显高于其他两种处理的,而到了10月又明显低于其他两种处理的,结合该处理各组分凋落物动态,可以得出降水减少会使其他树种的凋落期提前。有研究发现季节性的积水会造成凋落量发生改变,干旱导致森林凋落量显著增加[37],这说明凋落节律不仅受树种自身生物学和生态学特性的调控[43],还与土壤湿度降低密切相关[49]。在各组分凋落物中,红松在6月份的凋落量虽然相对于9—10月份的较小,但是明显高于7—8月份的,有研究表明针叶林的凋落量在一年之中也表现出明显的峰值[17, 50-51],苏格兰松的凋落期主要在8—10月,而花旗松有两个峰值,分别在春季和秋季[52],侯玲玲等[53]研究表明云冷杉红松林的凋落模式为双峰型,主要集中在9月,但是在4—5月份也出现一个小峰值。
3.3 不同处理对凋落量与胸高断面积相关性的影响
森林凋落量的多少受林分密度和林龄的影响,林分总胸高断面积不仅可以反应林分中树木的多少,还可以反映林分中树木的大小。李雪峰[34]在研究中发现阔叶凋落量不受其胸高断面积的影响,而红松叶凋落量随其树种的胸高断面积的增加而增加;原作强等[33]在研究中发现红松、紫椴、蒙古栎和色木槭的叶凋落量与样地内母树的胸高断面积呈显著正相关;而本研究发现,不同处理各组分凋落物量与胸高断面积均呈正相关,这与原作强等的研究结果一致,与李雪峰等的结论有一定出入。但各组分相关性变化对氮水控制响应不同,总体来看,施氮提高了各组分凋落物与其胸高断面积的相关性,降水减少降低了红松叶和蒙古栎叶与其胸高断面积的相关性,提高了枝、花果皮及总量的相关性。究其原因可能有以下两点:1)氮水处理改变了光合产物的分配模式,使树木原来的胸径发生了变化,赵亮等[54]在研究中发现,氮沉降显著提高了阔叶树种地上部分生物量的分配,其分配大小依次为径>枝>根>叶;Magill等[55]在对持续了15年氮添加实验进行总结时发现,针叶林和阔叶林生长对氮沉降的响应不相同,以美国赤松林为例,对照样地(空白处理)的平均直径增加量最高,低氮和高氮处理样地的直径增加量相对于对照样地分别下降了31%和54%,而期间阔叶林的响应则相反,低氮和高氮处理相对于对照样地使地上部分的NPP分别增加了11%和39%。2)氮水处理对胸径没有产生明显的影响,树种的凋落量主要还由自身生物特性调控,王樟华[56]认为,阔叶树种凋落量与其胸高断面积相关不显著的主要原因是不同树种自身的落叶量各不相同。
3.4 各组分凋落系数对氮水控制的响应
凋落物产量主要取决于土壤肥力、土壤水分和养分的供应[57],但在区域尺度上受海拔、纬度[58]、降雨量等影响较大[59]。本研究发现,氮添加(50 kg/(hm2·a))降低了林分凋落量,而降水减少会提高林分凋落物的产量,但未达到显著水平;氮添加显著降低了红松叶凋落量,降水减少显著提高了其他叶凋落量,对其他组分凋落量影响均不显著。有研究表明,30、60 kg/(hm2·a)的氮添加处理对凋落物量产生抑制作用[22];施氮会抑制美国赤松林叶片的产量[60];凋落物产量与降雨量呈负相关[59],在旱季阔叶凋落量比一般年份的要高[25],干旱会提高总凋落物产量[26, 61],这与本研究的结果相一致。花果皮对水氮控制的响应不明显,可能是因为森林的组织器官的凋落情况在一定程度上受气候因子(降雨和风)的直接调节,但其凋落物产量主要还受自身生物学特性的支配[62]。
另外,本研究还发现针叶树种和阔叶树种凋落量对氮水控制的响应各不相同,降雨减少会提高所有树种叶凋落物量,氮添加会降低针叶树种的凋落物量而提高阔叶树种的凋落物量。降水减少显著增加了阔叶树种的凋落量,对红松叶凋落量增加不显著,可能是因为阔叶树种对干旱的敏感性高于红松的,针叶树属于耐旱树种[63-64],对水分减少有一定的耐受性,有研究表明,氮沉降会降低植物获取水分的能力,从而增加了其对干旱的敏感性[3, 65],例如,荷兰高氮沉降地区的森林树种在干旱年份活力会明显降低,而到了正常年份又会恢复正常[66]。施氮显著降低了红松叶凋量,而林分的叶凋落量和总凋落量减少不明显,可能与施氮地内的土壤氮饱和状态有关,有研究表明,森林生态系统对氮素的饱和程度决定着植物生产力对氮沉降的响应,一般氮沉降的临界点在10~25 kg/(hm2·a)范围内,当氮素受限时,一定量的氮沉降量可以通过提高有效氮的供应来增加植物的生产力,对植物产生一定程度的“施肥效应”,但仅在短期内对植物的生长有促进作用[3],当达到饱和后,过量的氮素就会使土壤酸化、系统养分平衡失调[2],削弱树木对不良环境的抗性[67],扰乱森林的正常结构和功能[7-8],从而减少生态系统的生产力[68]。
-
表 1 RAD-seq基因组测序数据统计
Table 1 Data statistics of genomic sequencing generated by RAD-seq
样本
Sample过滤前 Before filter 过滤后 After filter 原始读长
Raw read/Mbp原始碱基总数
Total number of
original bases/GbpQ20/Gbp Q30/Gbp 干净读长
Clean read/Mbp有效碱基总数
Total number of
bpeffective base/GbpQ20/Gbp Q30/Gbp 平均长度
Average
length/bp‘九丰一号’
‘Jiufengyihao’31.929 4.789 4.478
(93.5%)4.147
(86.6%)27.805 4.034 3.935
(97.5%)3.758
(93.2%)145.1 ‘亚特’
‘Yate’45.994 6.899 6.652
(96.4%)6.367
(92.3%)42.560 6.304 6.211
(98.5%)6.047
(95.9%)148.1 注:Q20. 质量得分不低于20的碱基数及比例;Q30. 质量得分不低于30的碱基数及比例。
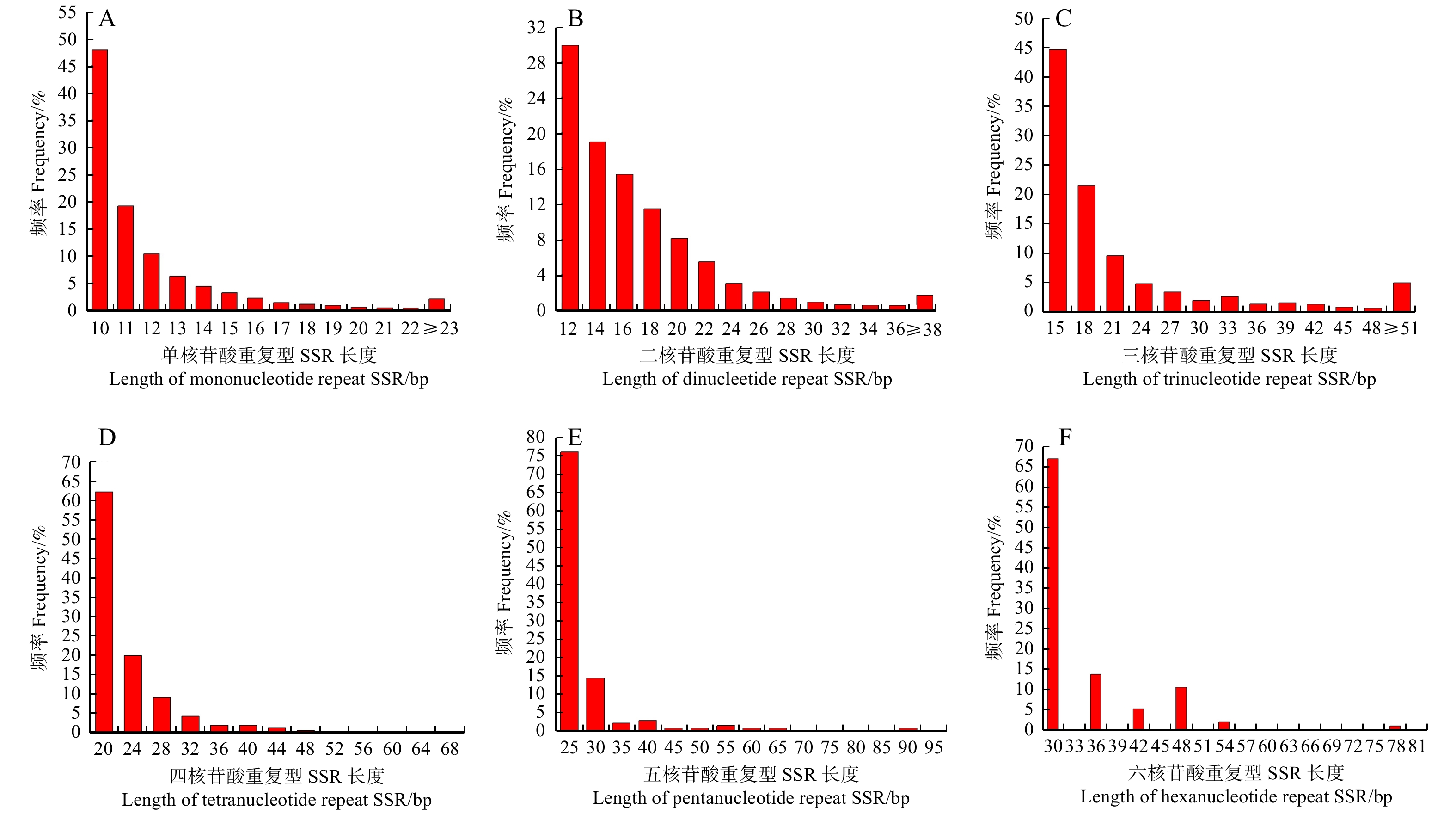
Notes:Q20, the number and proportion of bases with a quality score of not less than 20; Q30, the number and proportion of bases with a quality score of not less than 30.表 2 金银花基因组中SSR位点信息
Table 2 Information of SSR loci in Lonicera japonica
SSR类型
SSR type数量
Number重复次数
Repeat time平均长度
Average
length/bp百分比
Percentage/%重复单元类型 Motif type 数量 Number 主要基序 Main motif 单核苷酸 Mono-nucleotide 23 101 10 ~ 82 11.83 49.15 2 A/T,C/G 二核苷酸 Di-nucleotide 13 424 6 ~ 45 16.89 28.56 6 AT/TA,AG/CT,AC/GT 三核苷酸 Tri-nucleotide 3 883 5 ~ 49 22.04 8.26 59 AAT/ATT,GAA/TTC,TAA/TAA,AAG/CTT,TCA/TGA 四核苷酸 Tetra-nucleotide 540 5 ~ 14 23.03 1.15 86 AAAT/ATTT,TAAT/ATTA,TAAA/TTTA,CTTT/AAAG,TTTC/GAAA,ATAC/GTAT,TATC/GATA 五核苷酸 Penta-nucleotide 147 5 ~ 18 27.99 0.31 92 TTTTC/GAAAA,TTTCT/AGAAA,AAAAT/ATTTT,GGGTT/AACCC,ATCCA/TGGAT 六核苷酸 Hexa-nucleotide 94 5 ~ 13 34.40 0.20 74 GGCAAT/ATTGCC,GGAGAT/ATCTCC,GGTGGA/TCCACC,GATATA/TATATG,CAATCA/TGATTG,GCATTT/AAATGC 复合型 Compound 5 810 70.01 12.36 总计 Total 46 999 21.54 100.00 319 表 3 35个位点SSR引物多态性分析
Table 3 Polymorphism analysis of SSR primers at 35 loci
引物
Primer引物序列(5′—3′)
Primer sequence (5′−3′)SSR类型
Type of SSR重复基序
Repeat motifNa Ne He Ho H I PIC jap2 F:AGATGGATAGTGGGCCATTGT
R:TAGAGTCACCGGGAGCTACCP3 (TGA)5 6.000 2.342 0.598 0.402 0.573 1.211 0.544 jap5 F:GCACGTGCACTCAGAAACAG
R:GCAACCGTTGAGCTCCTTTGP3 (GTA)7 8.000 6.000 0.870 0.130 0.833 1.932 0.814 jap6 F:GCATAGACTCTGCTGGCCAA
R:AGCAGTGGCAGATATGGCTGP3 (TCA)6 4.000 3.236 0.721 0.279 0.691 1.258 0.635 jap15 F:TGAAAAGAAGGGAATAGGATTAGGGT
R:CCTCTTGGTGAGACCGCTTTP4 (GAAT)5 5.000 2.220 0.576 0.424 0.550 1.097 0.512 jap17 F:GAAGAAGAGCGGCTACTGCA
R:AAGAGTGAAGCAGCAGCAGTP3 (CAC)5 5.000 3.723 0.766 0.234 0.731 1.417 0.685 jap18 F:AGGCTACATGATGATTGAGACGA
R:TTGGCCCCACCTGATCAAAAP3 (ACA)6 4.000 2.504 0.627 0.373 0.601 1.054 0.524 jap21 F:ACCCTCACCTTCACCCTCAT
R:TGGGTCATCGCATCGTCAAAP3 (CAT)5 4.000 1.419 0.308 0.692 0.295 0.624 0.281 jap44 F:ATGGAGGGCTAGTGAATGCG
R:TCCACACTCGGTACACCAAAP3 (GAT)5 4.000 1.959 0.511 0.489 0.490 0.882 0.431 jap51 F:ACGTCGAATGTTAATGGCACG
R:TGCAGCACATTTGGCATTCTP3 (ATT)6 4.000 2.575 0.641 0.359 0.612 1.121 0.555 jap53 F:GTCTGGGGAATGAGTTGCCA
R:AGCCTTTGATCGAGTCAGGTP3 (TTG)14 7.000 3.556 0.750 0.250 0.719 1.549 0.686 jap73 F:TCAAACTCCTACCTGGCTGC
R:CTGTATGTGCCGGACATGGTP3 (GCT)6 6.000 2.642 0.649 0.351 0.622 1.328 0.594 yjap4 F:CAACTTCACCGGTGGATCCA
R:TCCTTCCAATTCGAAACTAAACCTP3 (TTG)5 4.000 2.483 0.623 0.377 0.597 1.119 0.552 yjap5 F:AAAGGCGTGGTGAAAGGTCT
R:CACATATGGTTGCTGCAGCCP3 (GAA)5 10.000 6.698 0.888 0.112 0.851 2.084 0.835 yjap6 F:TTCTAGGGCTTCGGCAACTG
R:CCTTCCCCTCGAAAACTGCAP3 (CTG)5 4.000 1.714 0.435 0.565 0.417 0.837 0.393 yjap9 F:TGGCAACTTTCCCCCAAATTC
R:TGAGCGAGTCATATAGGCGAGP3 (TTC)6 4.000 2.361 0.601 0.399 0.576 0.980 0.485 yjap10 F:AACGGGAAAGAGTGAGCGAG
R:GTGTGTTTCCCTTTTCCGGCP3 (ATT)5 3.000 1.834 0.475 0.525 0.455 0.803 0.410 yjap12 F:GCATAGACTCTGCTGGCCAA
R:AGCAGTGGCAGATATGGCTGP3 (TCA)5 5.000 2.852 0.678 0.323 0.649 1.305 0.614 yjap19 F:GGTGGATGAGCTTCTGAAGAGA
R:CCAATGGCTTCTTCCCCTGTP3 (TTG)6 5.000 2.028 0.529 0.471 0.507 0.994 0.463 yjap24 F:GCTTTTGCAGCTCCTACTTTGT
R:TTTGCTGTTAAGACACGCGCP3 (CAC)5 4.000 3.429 0.739 0.261 0.708 1.309 0.659 yjap36 F:GACACACACGGTTGCTTGAC
R:AGAGTTTCTGCTGTTGGTGGAP3 (GCA)6 4.000 2.969 0.692 0.308 0.663 1.200 0.600 yjap39 F:TCACTACCTGTATCCGCTCT
R:TGGAAGCAGAAAGGGCATCTP3 (ATC)7 4.000 2.743 0.663 0.337 0.635 1.140 0.566 yjap47 F:GCTTCGCCTTTGGTTCGGTA
R:CAACTCGCTCTCTCCCTTCCP3 (ATG)7 4.000 1.419 0.308 0.692 0.295 0.624 0.281 yjap48 F:GGGCATTTTCTGGTGGAGGA
R:AGCATCCAAACCTGATTTGTTTP3 (GTT)5 4.000 2.642 0.649 0.351 0.622 1.105 0.553 yjap51 F:CGGAGAGAGATACTAGGCGC
R:CGCGGCTGAATTTTAGAGCCP2 (TA)6 7.000 3.692 0.761 0.239 0.729 1.595 0.701 yjap53 F:CATTCATTCCCCCACCAGGT
R:GGGACCGGTCGAAACCAATAP2 (TC)10 5.000 2.034 0.533 0.468 0.508 1.056 0.483 yjap59 F:GGATCCAGCATGTCTCGAACT
R:TGCTTTGGCCCTTCCTCATTP2 (AT)8 4.000 2,215 0.573 0.428 0.549 1.013 0.500 yjap89 F:GAGTAAAGGTGGAGAGCGTGT
R:AGTTTCCGAGAAGTGCGTGTP4 (CTAT)5 4.000 3.310 0.728 0.272 0.698 1.283 0.645 yjap105 F:TGTTTAGCAACAAGGGATGGT
R:GCTGGCAGGGACAAAAAGTGP2 (GA)7 6.000 4.033 0.788 0.212 0.752 1.547 0.714 yjap109 F:AACTTCTGTGACAGGGCAGG
R:GCATATGCGGCAATGGGAAGP2 (GT)6 5.000 2.504 0.627 0.373 0.601 1.188 0.559 yjap120 F:GCCAGAGCTCCGATAAGGTC
R:TGCATCGCGTGTGTATGTGAP2 (AT)6 8.000 4.966 0.833 0.167 0.799 1.848 0.779 yjap153 F:ACATGAGGGTTTGCCGATCC
R:GGAGTATGGCCAGGTTCGTCP3 (GTG)6 5.000 2.852 0.678 0.323 0.649 1.253 0.596 yjap169 F:AGTTCGAGGGATTGTGCGTT
R:TGTGGAATTCGTCAATGGCGP3 (TTC)7 3.000 1.674 0.420 0.580 0.403 0.722 0.363 yjap171 F:TCAGAGAGGGGGTAACAGCA
R:TCGGCTTATACGACTGCGAGP3 (ACC)5 4.000 2.483 0.623 0.377 0.597 1.119 0.552 yjap175 F:GTTTCAATGCGAGCTCGACG
R:TCTCATTCTTCCCAAAATTCATCAGAP3 (TTA)6 9.000 3.388 0.736 0.265 0.705 1.647 0.681 yjap183 F:GGGGCCTCATTTTCCCTCAA
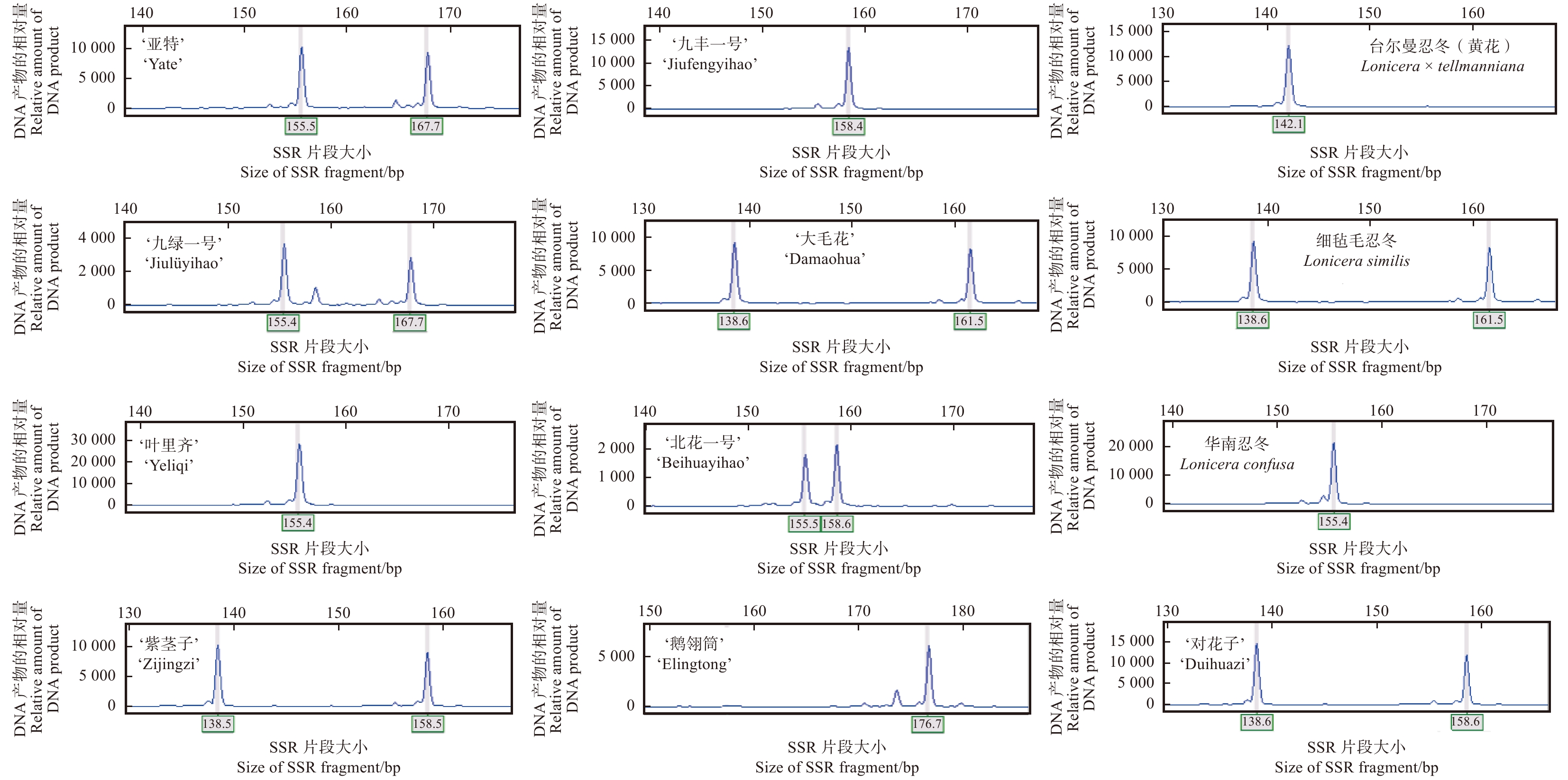
R:TTTTCCGGTGGTGGAGCAATP3 (CAC)5 5.000 3.236 0.721 0.279 0.691 1.342 0.644 均值 Mean 5.057 2.907 0.638 0.363 0.611 1.217 0.568 注:Ho. 观测杂合度;Na. 观测等位基因数;Ne. 有效等位基因数;He. 期望杂合度;I. Shannon信息指数;H. Nei’s遗传多样性指数;PIC. 多态性信息含量。Notes: Ho, observation of heterozygosity; Na, observed number of alleles; Ne, effective number of alleles; He, expected heterozygosity; I, Shannon information index; H, Nei ’s genetic diversity index; PIC, polymorphism information content. -
[1] Cai A C, Liao H Y, Wang C C, et al. A comprehensive study of the aerial parts of Lonicera japonica Thunb. based on metabolite profiling coupled with PLS-DA[J]. Phytochemical Analysis, 2020, 31(6): 786−800. doi: 10.1002/pca.2943
[2] 朱凤洁, 杨健, 袁媛, 等. 金银花种质资源化学指纹图谱及代谢物相似性分析[J]. 中国中药杂志, 2018, 43(12):2575−2579. Zhu F J, Yang J, Yuan Y, et al. Chemical fingerprinting and similarity analysis of Lonicera japonica resources[J]. China Journal of Chinese Materia Medica, 2018, 43(12): 2575−2579.
[3] 蔡芷辰, 刘训红, 王程成, 等. 金银花分子生物学研究进展[J]. 中国中药杂志, 2020, 45(6):1272−1278. Cai Z C, Liu X H, Wang C C, et al. Research progress in molecular biology of Lonicerae Japonicae Flos[J]. China Journal of Chinese Materia Medica, 2020, 45(6): 1272−1278.
[4] 张贵生, 王林泠, 方元平, 等. 基于SSR标记的大别山区金银花遗传多样性分析[J]. 分子植物育种, 2019, 17(19):6543−6548. Zhang G S, Wang L L, Fang Y P, et al. Genetic diversity analysis of Lonicera japonica in dabie mountains based on SSR markers[J]. Molecular Plant Breeding, 2019, 17(19): 6543−6548.
[5] 徐石勇, 李欧静, 张舒玮, 等. 金银花SSR指纹图谱的构建及遗传多样性分析[J]. 天津农业科学, 2015, 21(5):11−14,18. doi: 10.3969/j.issn.1006-6500.2015.05.003 Xu S Y, Li O J, Zhang S W, et al. Construction of SSR-based molecular finger printing and analysis of genetic diversity for Lonicera japonica[J]. Tianjin Agricultural Sciences, 2015, 21(5): 11−14,18. doi: 10.3969/j.issn.1006-6500.2015.05.003
[6] 孙稚颖, 姚辉, 王振中, 等. 金银花种质资源遗传多样性的ISSR分析[J]. 世界科学技术-中医药现代化, 2013, 15(9):1890−1895. Sun Z Y, Yao H, Wang Z Z, et al. ISSR analysis on genetic diversity of germplasm resource of Lonicerae Japonicae Flos[J]. Modernization of Traditional Chinese Medicine and Materia Medica-World Science and Technology, 2013, 15(9): 1890−1895.
[7] Heckenberger M, Vort J, Peleman J, et al. Variation of DNA fingerprints among accessions within maize inbred lines and implications for identification of essentially derived varieties( II): genetic and technical sources of variation in AFLP data and comparison with SSR data[J]. Molecular Breeding, 2003, 12: 97−106. doi: 10.1023/A:1026040007166
[8] 王久利, 朱明星, 徐明行, 等. 基于RAD-seq技术的异型花SSR信息分析[J]. 植物研究, 2017, 37(3):447−452. Wang J L, Zhu M X, Xu M X, et al. Analysis on SSR in Sinoswertia tetraptera base on RAD-seq[J]. Bulletin of Botanical Research, 2017, 37(3): 447−452.
[9] 黄兴发, 尹跃, 赵建华, 等. 黑果枸杞基因组SSR标记开发及遗传多样性分析[J]. 西北农林科技大学学报(自然科学版), 2021,49(1):126−135. Huang X F, Yin Y, Zhao J H, et al. Development of genomic SSR markers and genetic diversity analysis of Lycium ruthenicum Murr.[J]. Journal of Northwest A&F university (Natural Science Edition), 2021,49(1): 126−135.
[10] Kumar A A, Deepak S, Archana V, et al. MSDB: a comprehensive, annotated database of microsatellites[J]. Nucleic Acids Research, 2020, 48(D1): D155−D159. doi: 10.1093/nar/gkz886
[11] Andrews K R, Good J M, Miller M R, et al. Harnessing the power of RAD-seq for ecological and evolutionary genomics[J]. Nature Review Genetics, 2016, 17(2): 81−92. doi: 10.1038/nrg.2015.28
[12] Hou Y, Nowak M D, Mirré V, et al. RAD-seq data point to a northern origin of the arctic-alpine genus Cassiope (Eri-caceae)[J]. Molecular Phylogenetics and Evolution, 2016, 95: 152−160. doi: 10.1016/j.ympev.2015.11.009
[13] Yuan Z, Fang Y, Zhang T, et al. The pomegranate (Punica granatum L.) genome provides insights into fruit quality and ovule developmental biology[J]. Plant Biotechnology Journal, 2018, 6(7): 1363−1374.
[14] Miller M R, Dunham J P, Amores A, et al. Rapid and cost-effective polymorphism identification and genotyping using restriction site associated DNA (RAD) markers[J]. Genome Research, 2007, 17(2): 240−248. doi: 10.1101/gr.5681207
[15] Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads[J]. Embnet Journal, 2011, 17(1): 10−12.
[16] Andreas U, Ioana C, Triinu K, et al. Primer3–new capabilities and interfaces[J]. Nucleic Acids Research, 2012, 40(15): e115. doi: 10.1093/nar/gks596
[17] Schuelke M. An economic method for the fluorescent labeling of PCR fragments[J]. Nature Biotechnology, 2000, 18: 233−234. doi: 10.1038/72708
[18] Hulce D, Li X, Snyder-Leiby T, et al. GeneMarker genotyping software: tools to increase the statistical power of DNA fragment analysis[J]. Journal of Biomolecular Techniques, 2011, 22(Suppl.): S35−S36.
[19] Yeh F C, Yang R C, Boyle T, et al. POPGENE Version 1.32: Microsoft Windows-based freeware for populations genetic analysis[Z]. Edmonton: University of Alberta, 1999.
[20] Marshall T C, Slate J, Kruuk L, et al. 1998. Statistical confidence for likelihood-based paternity inference in natural populations[J]. Molecular Ecology, 1998, 7(5): 639−655. doi: 10.1046/j.1365-294x.1998.00374.x
[21] 洪文娟, 郝兆祥, 刘康佳, 等. 基于石榴全基因组序列的SSR标记开发及鉴定[J]. 北京林业大学学报, 2019, 41(8):38−47. Hong W J, Hao Z X, Liu K J, et al. Development and identification of SSR molecular markers based on whole genomic sequences of Punica granatum[J]. Journal of Beijing Forestry University, 2019, 41(8): 38−47.
[22] Feng J Y, Zhao S, Li M, et al. Genome-wide genetic diversity detection and population structure analysis in sweet potato (Ipomoea batatas) using RAD-seq[J]. Genomics, 2020, 112(2): 1978−1987. doi: 10.1016/j.ygeno.2019.11.010
[23] 刘瑞娟, 路兴旺, 窦全文. 基于简化基因组测序开发垂穗披碱草(Elymus nutans)SSR标记[J]. 分子植物育种, 2018, 16(6):1888−1894. Liu R J, Lu X W, Dou Q W. Development of SSR markers in Elymus nutans based on reduced-representation genome sequencing[J]. Molecular Plant Breeding, 2018, 16(6): 1888−1894.
[24] 宋立肖, 李国旗, 靳长青, 等. 大麻状罗布麻的全基因组分析和SSR标记开发[J]. 植物遗传资源学报, 2019, 20(5):1309−1316. Song L X, Li G Q, Jin C Q, et al. Whole genome sequencing and development of SSR markers in Apocynum cannabinum[J]. Journal of Plant Genetic Resources, 2019, 20(5): 1309−1316.
[25] Morgante M, Hanafey M, Powell W, et al. Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes[J]. Nature Genetics, 2002, 30(2): 194−200. doi: 10.1038/ng822
[26] Huo N, Lazo G, Vogel J P, et al. The nuclear genome of Brachypodium distachyon: analysis of BAC end sequences[J]. Functional Integrative Genomics, 2008, 8(2): 135−147. doi: 10.1007/s10142-007-0062-7
[27] Tóth G, Gáspári Z, Jurka J, et al. Microsatellites in different eukaryotic genomes: survey and analysis[J]. Genome Research, 2000, 10(7): 967. doi: 10.1101/gr.10.7.967
[28] Yang Y Y, He R Q, Zheng J, et al. Development of EST-SSR markers and association mapping with floral traits in Syringa oblata[J]. BMC Plant Biology, 2020, 20(1): 436. doi: 10.1186/s12870-020-02652-5
[29] Li D Y, Long C, Pang X M, et al. The newly developed genomic-SSR markers uncover the genetic characteristics and relationships of olive accessions[J]. PeerJ, 2020, 8: 341−345.
[30] Haydar K, Ying L, Wieland M. Survey of simple sequence repeats in completed fungal genomes[J]. Molecular Biology & Evolution, 2005, 22: 639−649.
[31] Tuskan G A, Gunter L E, Yang Z K, et al. Characterization of microsatellites revealed by genomic sequencing of Populus trichocarpa[J]. Canadian Journal of Forest Research, 2004, 34: 85−93. doi: 10.1139/x03-283
[32] 阎毛毛, 戴晓港, 李淑娴, 等. 松树、杨树及桉树表达基因序列微卫星比对分析[J]. 基因组学与应用生物学, 2011, 30(1):103−109. doi: 10.3969/gab.030.000103 Yan M M, Dai X G, Li S X, et al. Sequence analysis and comparison of EST-SSRs in pine, poplar and eucalyptus[J]. Genomics and Applied Biology, 2011, 30(1): 103−109. doi: 10.3969/gab.030.000103
[33] 周晓君, 王海亮, 李方玲, 等. 基于RAD-seq技术开发灵宝杜鹃多态性SSR标记[J]. 农业生物技术学报, 2019, 27(1):55−62. Zhou X J, Wang H L, Li F L, et al. Development of polymorphic SSR markers in Rhododendron henanense subsp. lingbaoense based on RAD-seq[J]. Journal of Agricultural Biotechnology, 2019, 27(1): 55−62.
[34] Beghè D, Molano J F G, Fabbri A, et al. Olive biodiversity in Colombia, a molecular study of local germplasm[J]. Scientia Horticulturae, 2015, 189: 122−131. doi: 10.1016/j.scienta.2015.04.003
[35] Cregan P B, Jarvik T, Bush A L, et al. An integrated genetic linkage map of the soybean genome[J]. Crop Science, 1999, 39: 1464−1490. doi: 10.2135/cropsci1999.3951464x
[36] Song Q J, Fickus E W, Cregan P B, et al. Characterization of trinucleotide SSR motifs in wheat[J]. Theoretical and Applied Genetics, 2002, 104: 286−293. doi: 10.1007/s001220100698
[37] 金玲, 刘明国, 董胜君, 等. 97个山杏无性系的遗传多样性及SSR指纹图谱[J]. 林业科学, 2018, 54(7):51−61. doi: 10.11707/j.1001-7488.20180706 Jin L, Liu M G, Dong S J, et al. Genetic diversity and fingerprints of 97 Armeniaca sibirica clones based on SSR markers[J]. Scientia Silvae Sinicae, 2018, 54(7): 51−61. doi: 10.11707/j.1001-7488.20180706
-
期刊类型引用(13)
1. 夏甜甜,杜秋硕,刘鹏. 基于数字赋能的泰山古柏生态修复技术应用探究. 山东林业科技. 2024(02): 77-83 .  百度学术
百度学术
2. 叶程浩,杜晓晨,鲍宇. 一种基于YOLOv5和轮廓约束的木材空洞缺陷应力波层析成像算法. 木材科学与技术. 2023(05): 61-68 .  百度学术
百度学术
3. 侯艳芳,王鑫,侯婷婷,郭靖,张艳. 古建筑木结构损伤检测方法研究进展. 粘接. 2022(01): 123-126+130 .  百度学术
百度学术
4. 宋辉,姬晓文,高琦,缑鹏超,王志强. 基于数据驱动的不规则用电检测误差校正系统设计. 电子设计工程. 2022(06): 109-112+117 .  百度学术
百度学术
5. 杨露露,董喜斌,徐华东. 电阻法和应力波法在活立木内部腐朽缺陷检测中的对比. 森林工程. 2022(04): 82-88 .  百度学术
百度学术
6. 戴禄君,吴瑞瑞. 数据驱动的非线性光学显微成像误差校正研究. 激光杂志. 2022(07): 159-163 .  百度学术
百度学术
7. 路程琳,何宇航,谢雨宏,刘依萍,刘嘉仪,王正. 应力波断层扫描法在立木检测方面的研究现状. 林业机械与木工设备. 2022(10): 37-40 .  百度学术
百度学术
8. 刘涛,李光辉. 基于射线分割的林木应力波断层成像算法. 林业科学. 2021(09): 181-192 .  百度学术
百度学术
9. 赵松,王勐. 移动AR+VR支持下全景图像拼接均匀性校正方法. 计算机仿真. 2021(11): 189-192+398 .  百度学术
百度学术
10. 何宇航,陈偲,王弘历,黄席阳,王正. 断层扫描法在木材无损检测的研究进展. 木工机床. 2020(01): 13-16 .  百度学术
百度学术
11. 张慧娟. 木材应力波检测精度的影响因素研究. 中国林副特产. 2020(01): 27-30 .  百度学术
百度学术
12. 魏喜雯,孙丽萍,许述正,杨扬,杜春晓. 基于应力波传播速度模型的原木缺陷定量检测. 北京林业大学学报. 2020(05): 143-154 .  本站查看
本站查看
13. 刘嘉新,高景泉,李超. 应用兰德韦伯算法的木材缺陷图像重建. 东北林业大学学报. 2019(12): 125-128 .  百度学术
百度学术
其他类型引用(8)



 下载:
下载: