Analysis of mtDNA genetic diversity and genetic structure of captive forest musk deer population
-
摘要:目的 研究林麝饲养种群的遗传多样性现状及遗传结构,为指导种群遗传管理及其饲养种群种质保护与利用提供理论依据。方法 遴选陕西省宝鸡市凤县的片仔癀麝场和逢春麝场,汉中市留坝县的太子岭麝场,采用分子粪便学的方法,采集220份新鲜的粪便样品,成功获得112条mtDNA控制区序列,计算单倍型多样性和核苷酸多样性。结果 在3个林麝繁育基地共检测到96个变异位点,均为简约信息位点,突变形式为转换和颠换,未出现插入和缺失现象;共获得36个单倍型,单倍型多样性和核苷酸多样性分别为0.957 8和0.039 5,单倍型多样性以片仔癀麝场最高,为0.953 9,留坝群体最低,为0.842 8;饲养种群间出现显著的遗传分化,其中,太子岭麝场和逢春麝场的遗传分化系数FST(0.137 7)最高,而太子岭麝场和片仔癀麝场的FST(0.077 2)最低。结论 饲养林麝种群较之众多濒危有蹄类饲养种群的遗传多样性高,但林麝饲养种群之间基因交流较少,建议实施基于非损伤取样的粪便mtDNA分析技术对群体内和群体间进行遗传管理,保护饲养林麝种群现有的遗传多样性。Abstract:Objective The research aimed to study the genetic diversity and genetic structure of the captive forest musk deer population, and provide scientific basis for guiding the population genetic management and protection and utilization of breeding population germplasm.Method This study collected 220 fresh samples from Pianzaihuang Musk Deer Farm and Fengchun Musk Deer Farm in Feng County, Baoji City, and Taiziling Musk Deer Farm in Liuba County, Han Zhong City of Shaanxi Province, northwestern China and sequenced 112 mitochondrial DNA control region sequences successfully. Haplotype diversity and nucleotide diversity were calculated.Result The 96 mutation sites were detected in the three forest musk deer breeding farms, all of which were simple information sites, with the mutation forms of conversion and translocation, without insertion or deletion. A total of 36 haplotypes were obtained, and the haplotype diversity and nucleotide diversity were 0.957 8 and 0.039 5, respectively. The haplotype diversity was the highest in Pianzaihuang population (0.953 9) and the lowest in Liuba population (0.842 8). The three populations showed significant genetic differentiation, among which the genetic differentiation coefficient FST (0.137 7) of Taiziling musk deer population and Fengchun musk deer population was the highest, while the FST (0.077 2) between Pianzaihuang and Taiziling was the lowest.Conclusion The genetic diversity of the captive forest musk deer is higher than that of many endangered ungulate species in captive, but the gene exchange among their breeding population is limited, and genetic differentiation has appeared. It is suggested to strengthen the exchange of genetic sources and enhance the gene exchange in order to help protect the genetic diversity of forest musk deer populations in captive.
-
Keywords:
- forest musk deer /
- mtDNA control region /
- genetic diversity /
- genetic structure
-
古筝作为中国传统弹拨乐器的一种,不仅民族文化悠久,演奏的音域还很广泛,这与构成它的部件密不可分,尤其共鸣面板对古筝的发声起了至关重要的作用[1-2]。古筝共鸣面板的振动性能与众多因素有关,其中共鸣面板的结构是影响其振动性能的重要因素之一。目前很多古筝制造厂多采用拼板与整板两种结构的共鸣面板,拼板结构的面板制作采用折弯工艺,由多块直板胶黏拼接后压弯制成[3];整板结构的面板制作采用压弯工艺,将整板烘烤压弯制成。我国民族乐器的发展起源较早,但在对民族乐器共鸣面板及用材性能的评价中,多是通过演奏者或技师的主观评判,这已不适应当前乐器工业发展的需要[4-5]。随着社会经济的繁荣发展以及民族乐器发展的复杂化,人们对于民族乐器的要求逐渐提高,越来越多的学者对其振动特性进行了研究。
作为力学的一个分支,模态分析技术早已应用于乐器振动研究领域,为乐器声学研究提供了新的技术手段[6-7]。其主要方法有:(1)计算模态分析,利用有限元分析等现代方法进行力学建模,通过计算机仿真分析得出结果,是借助于计算机的理论分析方法;(2)实验模态分析,使用科学仪器对实物或者模型进行测试,验证理论分析结果,或者直接获得其经验式的力学规律来解决问题。
随着人们对乐器的认识和研究,这类技术被广泛应用。如克拉尼(Chladni)用琴弓摩擦引起薄板振动,从中发现的克拉尼图形成为检验和研究乐器声学效果的有效方法,后来被许多人应用到吉他等其他乐器研究中[8-10];通过ANSYS进行仿真,研究琴体的结构或材料对其振动特性的影响,为乐器的设计和制作、音质优化提供了客观依据[11-13];共鸣面板作为乐器的关键部件,也常被利用这项技术进行单独研究,对实现生产过程中指导音板结构设计修改及其质量控制具有一定意义[14-16];两种模态分析各有优缺点,许多研究将两者结合,通过计算模态分析得到乐器振动特性参数,分析这些参数得出其振动特性,再通过实验模态分析法验证其结果的正确性,为音板声学品质提供了客观评测方法,有利于科学客观的音板声学品质评测系统的研究和建立[17-19]。目前在乐器声学振动特性领域,大多数学者都是将研究重点放在共鸣面板用小试件上,对整块共鸣面板的分析和评价相对较少。而以整个共鸣面板为研究对象,相对于以梁为研究对象更加贴近古筝共鸣面板的振动形式,使得研究结果更具严谨性。
基于上述分析,本研究以整板结构共鸣面板为研究对象,利用实验模态分析、计算模态分析对共鸣面板的振动特性进行研究,并综合比较实验模态分析与计算模态分析结果,验证计算模态应用于乐器共鸣面板振动特性分析的可行性,以期为整板结构古筝共鸣面板振动性能的研究以及对今后进一步探究不同类型古筝共鸣面板的振动规律提供一定理论依据,对于制造环节与产品声学性能提高也将具有一定意义。
1. 材料与方法
1.1 材 料
本研究选用扬州某企业提供的泡桐制乐器共鸣面板。具体实验对象为泡桐(Paulownia fortunei)制整板结构共鸣面板。其含水率处于12% ~ 14%,密度约等于260 kg/m3,几何参数如表1所示。
表 1 共鸣面板的尺寸规格Table 1. Geometric parameters of soundboardmm 长度
Length厚度
Thickness首部宽度
Head width尾部宽度
Tail width宽度方向弧长半径
Arc length radius in width direction长度方向弧长半径
Arc length radius in length direction1 630 7 350 295 330 7 300 1.2 方 法
1.2.1 实验模态分析
模态是指动力结构的固有振动特性,用固有频率、阻尼系数和固有振型等模态参数来描述。模态分析即结构固有振动特性的分析,目的是为了获得模态参数。实验模态分析法是通过实验采集激励力信号及振动响应信号,经过数据转换,求得频响函数(频响函数是用模态参数来表示)。从求得的频响函数估计出研究对象的模态参数,再对其固有频率、阻尼系数、固有振型等模态参数进行分析得出乐器的固有振动特性。
1.2.2 计算模态分析
有限元法是求解各类工程问题近似解的一种数值计算方法。计算模态分析法是将乐器实体结构离散成互不重叠的有限个微小单元体,其各相邻单元由节点连接。用这些离散的单元集合体的振动特性代替整个音板的振动特性,借助有限元软件ANSYS计算出研究对象的振动特性参数,包括固有频率、阻尼系数、固有振型等,分析这些参数得出乐器的固有振动特性[18]。
2. 实验模态分析
2.1 实验设置
为了使得到的结果更加接近古筝共鸣面板在实际演奏中的振动,本研究将古筝共鸣面板固定在共鸣箱上,共鸣箱内部框架材料为云杉( Picea asperata),底板材料为泡桐。经多次预实验后,最终决定将古筝共鸣面板划分为横纹理方向(x方向)6等分,顺纹理方向(y方向)32等分,需要采集信号的总点数为185个。在ZSDASP信号采集分析软件中建立几何结构模拟古筝共鸣面板,并使实际激励点与几何结构上的点相对应。采用ZSL系列冲击力锤逐一敲击试件上的点,通过ZSDASP信号采集分析软件对经ZS7016动态信号采集仪及微型压电式PE加速度传感器采集到的数据进行分析,得到试件传递函数的幅频,最终通过信号处理并选取模态因子函数,得到各阶共振频率及其对应的模态振型[20-21],实验设置如图1所示。
2.2 实验模态结果与分析
对古筝共鸣面板进行模态分析后,最终得到各阶次频率及其对应的模态振型图。为便于分析,本研究通过得到的振型图绘制了实验对象振动模态的节线图,如图2所示。
从图2可以得出整板结构共鸣面板的模态振型有如下特点:能够识别到13阶的共振频率与振型,依次为(0,1)、(0,2)、(0,3)、(1,3)、(0,4)、(1,5)、(2,4)、(2,5)、(2,6)、(1,7)、(1,8)、(2,8)、(1,11)阶。其中只沿横纹理方向的弯曲振动阶次为(0,1)、(0,2)、(0,3)、(0,4),在振型上分别表现为有1 ~ 4条顺纹理方向的振动节点线,且振动位移呈顺纹理方向对称。沿顺纹理方向的弯曲振动阶次为(1,n)、(2,n)阶,在振型上分别表现为有1、2条横向的振动节点线。(1,3)、(1,5)、(2,4)、(2,5)、(2,6)、(1,7)、(1,8)、(2,8)、(1,11)阶均为沿顺纹理方向和横纹理方向弯曲振动的叠加,振动形式复杂。另外,从各阶模态振型和振动节线图可以看出,随着阶次的逐渐升高,整板结构共鸣面板的振型也越复杂。同时,得到的振型均匀程度和清晰度也不尽相同,(0,n)阶对应的模态振型相对清晰易识别,(1,n)、(2,n)中的较低阶次,即(1,3)、(1,5)、(2,4)、(2,5)、(2,6)对应的振型识别较为困难。进一步综合分析识别到的整板结构古筝共鸣面板各阶频率的变化趋势,其结果如图3所示。
从图3可以得出,各阶次的频率具有如下特点:随着n值的增大,各阶的共振频率逐渐增大。同时,(2,n)阶的共振频率整体高于(1,n)阶,而(1,n)阶的共振频率整体大于(0,n)阶对应的共振频率。
结合之前所得出的特点,随着整板结构共鸣面板沿顺纹理方向和横纹理方向的阶次逐渐升高,对应的共振频率增大,且对应的模态振型均趋于复杂;整板结构共鸣面板的振动阶次多集中在(1,n)和(2,n)阶;从振型上看,整板结构共鸣面板(0,n)阶对应的模态振型相对清晰易识别,(1,n)、(2,n)中的较低阶次对应的振型则不均匀且较难识别。这一方面是由于实验的过程虽尽力营造适合模态分析的最佳条件,但最终的结果仍会受实验环境噪声、人工激励产生误差、边界条件等因素的影响而无法达到完全理想状态,且实验材料本身也并非均质,使得最终得到的模态振型无法达到理想条件下的均匀整齐。另一方面,本研究的对象区别于梁、平板的振动,除了板振动时正常的横纹理方向振动、顺纹理方向振动外,其特定的曲面结构、特殊的边界条件等,都会使其振动更加复杂。这就导致模态分析过程中有的阶次易识别且振型较为明显均匀,有的阶次不易识别,相对应的振型不明显。鉴于本实验个别阶次没有识别到,本研究将从整体上把握整板结构共鸣面板的振动规律和特点,共鸣面板各阶频率之间的具体规律及其具体比例关系需进一步研究得到[15]。
3. 计算模态分析
借助软件Solidworks 2016和ANSYS14.5对试件进行模拟并对所建模型进行模态分析,并与实验模态分析结果进行对比,验证计算模态分析的可行性。
3.1 共鸣面板模型的建立
本研究省略了顺纹理方向(y方向)的面板弧度,只考虑横纹理方向(x方向)的弧度,同时将面板看成左右同宽的规则结构(俯视图为矩形)。借助SolidWorks软件和通过实际测量获得的几何参数建立了整板结构古筝共鸣面板的三维模型,将模型导入到ANSYS Workbench中进行模态分析,将定义的新材料应用到所建的模型上[22]。对整板结构共鸣面板模型进行网格划分,共划分了2 144个单元,节点数为15 761个。
3.2 计算模态分析结果及数据分析
根据实验模态分析结果,设定古筝模型的频率范围为0 ~ 1 500 Hz,对整板结构共鸣面板模型进行求解,得到各阶共振频率和模态振型。依据实验获得的阶次范围确定模型的振型阶次。
从图4中的模型结果可以得出,所得振型均随着频率和阶数的升高而变得复杂,这一特点与实验所得结果一致。计算模态分析所得的振型相对实验模态分析所得振型图更均匀且平整。这与诸多因素有关,一方面,计算模态分析时软件会将模型看作是结构均匀致密的材料,不存在孔隙、裂纹或其他缺陷;另一方面,计算模态分析不需要像实验一样从外部采集信号,这样会避免外部环境、人为激励所造成的影响,所得结果较为理想化。
3.3 ANSYS模态分析结果与实验所得结果对比分析
对计算模态和实验模态所得阶次对应的频率进行对比并分析,结果如表2、图5所示。
表 2 计算模态分析与实验所得各阶频率对比Table 2. Frequency comparison between calculated modal analysis and experimental results阶数
Order频率 Frequency/Hz 误差
Error/%阶数
Order频率 Frequency/Hz 误差
Error/%实验结果
Experimental result计算结果
Calculated result实验结果
Experimental result计算结果
Calculated result(0,0) (1,5) 608.59 608.59 1.17 (0,1) 238.28 (1,6) 764.21 764.21 (0,2) 347.66 (1,7) 910.29 910.29 2.21 (0,3) 425.78 (1,8) 921.97 921.97 1.73 (0,4) 492.19 (1,9) 1 037.00 1 037.00 (0,5) (1,10) 1 117.20 1 117.20 (0,6) (1,11) 1 281.00 1 281.00 −0.63 (0,7) (2,4) 653.58 653.58 −0.41 (0,8) (2,5) 712.17 712.17 3.00 (0,9) (2,6) 758.96 758.96 1.72 (0,10) (2,7) 941.58 941.58 (1,3) 460.94 462.45 0.33 (2,8) 1 032.50 1 032.50 0.50 (1,4) 537.56 从表2可以得出,计算模态分析得到的结果更具连续性,能够识别到选定阶数范围的所有阶次,而实验模态分析时,个别阶数不够明显而识别不到。计算结果中,整板结构共鸣面板模型能够识别到的阶次为(1,n)阶和(2,n)阶,与实验所得结果相比,缺少(0,n)阶,这是由于计算模态分析与实验模态相比存在一定误差。实验模态分析时,共鸣面板被安置在共鸣箱上再进行四周固定;而计算模态分析时为了针对性地分析共鸣面板的各阶次频率及振型,直接对共鸣面板模型进行了四周固定[15]。
从图5中可以看出,计算模态分析所得各阶频率与实验结果具有相同的变化趋势,各阶频率均随着阶次的升高而逐渐增大,且计算结果与实验结果得到的各阶次对应频率很接近。具体从误差值来看,计算模态分析所得结果除个别阶次外,整体略高于实验模态所得结果。这一方面是由于软件在计算时会将所建模型默认为是材质均匀致密的无缺陷的试件;另一方面,对模型进行前处理时所设置的四周固定边界条件为理想状态,而在实际的实验过程中很难达到理想中的四周完全固定[23]。但实验对象计算模态分析所得各阶振型对应的频率误差均在5%以内,基于上述分析,可得出该计算模态所得振型及对应频率较为合理,证明了ANSYS模态分析对本研究具有一定可行性。
我国森林资源总量相对不足、质量不高、分布不均,木材市场“需大于供”的状况尚未得到根本改变[24]。整板结构共鸣面板的原材料相对较少,成本较高,而拼板结构共鸣面板对于木材资源的利用率更高。现如今制筝厂通常采用多种面板拼合的方式制作古筝,不仅提高了制作效率,还能通过无缝拼接使木质更加均匀,统一不同音区的音色[25]。综上,今后可在本研究基础上进一步探究不同结构共鸣面板的振动特点,为厂家在制造共鸣面板过程中所遇到的问题提供一定的帮助和参考。
4. 结 论
本研究以整板结构古筝共鸣面板为研究对象,利用ZS7016动态信号采集仪对整板结构共鸣面板进行实验模态分析,得到各阶振动频率及模态振型,从而实现对整板结构共鸣面板声振动特点的分析;并借助软件SolidWorks和ANSYS对共鸣面板建立了三维模型进行计算模态分析,将求解结果与实验模态结果进行对比以探究该方法应用于本研究的可行性,最终得到以下结论:
(1)实验模态分析结果显示:整板结构共鸣面板的振动阶次多为(1,n)和(2,n)阶;(0,n)阶对应的模态振型清晰易识别,而(1,n)、(2,n)中的较低阶次对应的振型识别较为困难。对古筝共鸣面板上的激励点进行信号拾取时,发现拾取难度由筝首到筝尾逐渐降低。
(2)计算模态分析结果显示,整板结构共鸣面板模型能够识别到的阶次为(1,n)阶和(2,n)阶,与实验所得结果相比缺少(0,n)阶。同时,计算模态分析能够识别到选定阶数范围的所有阶,而实验模态分析时个别阶数较难识别。
(3)对比计算模态所得各阶频率,与实验模态结果一致。计算模态分析与实验模态所得各阶频率均随着阶次的升高而逐渐增大,同时共鸣面板所得模态振型均趋于复杂。但计算模态分析的振型更加均匀且理想化。
(4)共鸣面板计算模态分析所得各阶频率整体上比实验模态所得结果略高,但误差均在5%以内,处于合理范围。综合前面得出的结论,计算模态分析应用于本研究具有一定的可行性。
-
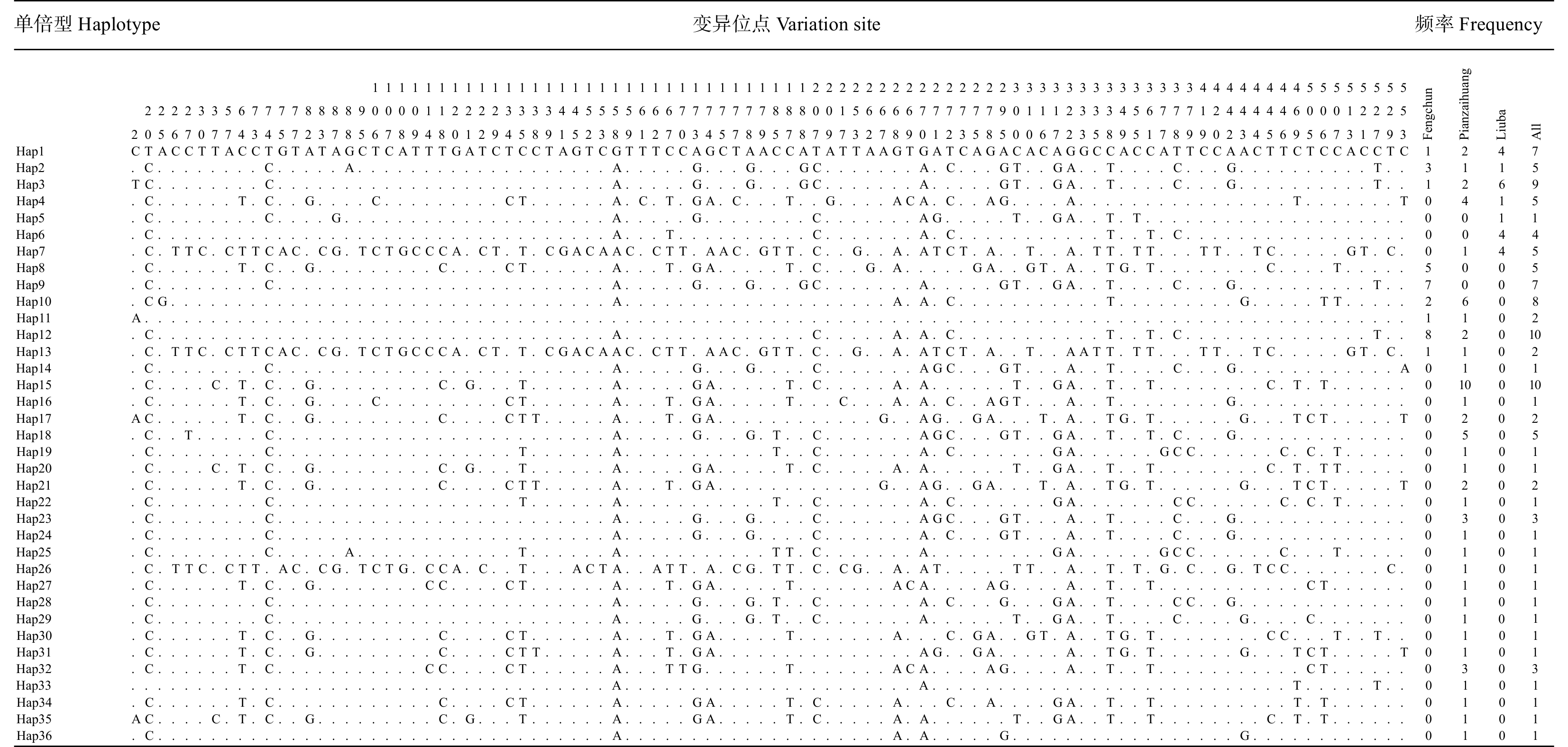
表 1 林麝的遗传多样性参数
Table 1 Genetic diversity parameters of forest musk deer
群体
Population样本量
Number of sample变异位点
Variation site单倍型数
Number of haplotype单倍型多样性
Haplotype diversity核苷酸多样性
Nucleotide diversity逢春群体
Fengchun population29 82 9 0.844 8 0.030 3 片仔癀群体
Pianzaihuang population62 95 32 0.953 9 0.038 2 留坝群体
Liuba population21 78 7 0.842 8 0.048 1 合计Total 112 96 36 0.957 8 0.039 5 表 2 3个林麝群体内(对角线)和群体间(对角线下)的平均遗传距离
Table 2 Mean genetic distances within (on the diagonal) and between (below the diagonal) three forest musk deer populations
群体
Population逢春群体
Fengchun population片仔癀群体
Pianzaihuang population留坝群体
Liuba population逢春群体
Fengchun population0.032 0 片仔癀群体
Pianzaihuang population0.038 8 0.040 3 留坝群体
Liuba population0.043 7 0.049 5 0.052 4 表 3 林麝群体分子方差分析
Table 3 Analysis of molecular variance of M. berezovskii populations
变异来源
Source of variation自由度
Degree of freedom方差平方和
Sum of squares of variance变异组分
Variance component变异百分比
Percentage of variation/%种群间 Inter population 2 3.808 0.043 82 8.82 种群内 Within population 109 49.353 0.452 78 91.18 总计
Total111 53.161 0.496 60 遗传分化系数 Genetic differentiation coefficient (FST) 0.088 2 表 4 群体间的遗传分化系数
Table 4 Genetic differentiation coefficient (FST) between populations
群体 Population 逢春群体
Fengchun population片仔癀群体
Pianzaihuang population留坝群体
Liuba population逢春群体
Fengchun population+ + 片仔癀群体
Pianzaihuang population0.079 1 + 留坝群体
Liuba population0.137 7 0.077 2 注:+ 表示遗传分化显著(P < 0.05)。Note: + indicates significant genetic differentiation (P < 0.05). -
[1] Pautasso M. Challenges in the conservation and sustainable use of genetic resources[J]. Biology Letters, 2012, 8(3): 321−323. doi: 10.1098/rsbl.2011.0984
[2] Pauls S U, Nowak C, Bálint M, et al. The impact of global climate change on genetic diversity within populations and species[J]. Molecular Ecology, 2013, 22(4): 925−946. doi: 10.1111/mec.12152
[3] Kolleck J, Yang M, Zinner D, et al. Genetic diversity in endangered Guizhou snub-nosed monkeys (Rhinopithecus brelichi): contrasting results from microsatellite and mitochondrial DNA data[J]. PLoS One, 2013, 8(8): 1−7.
[4] 沈富军, 张志和, 李光汉, 等. 圈养大熊猫的系谱分析[J]. 遗传学报, 2002, 29(4): 307−313. Shen F J, Zhang Z H, Li G H, et al. Pedigree analysis of captive giant panda[J]. Acta Genetica Sinica, 2002, 29(4): 307−313.
[5] 廖辉, 袁梨, 殷毓中, 等. 中国动物园大猩猩圈养种群现状分析[J]. 野生动物学报, 2015, 36(4): 378−381. doi: 10.3969/j.issn.1000-0127.2015.04.003 Liao H, Yuan L, Yin Y Z, et al. Genetic diversity and statistical feature of the captive gorilla population in China’s zoos[J]. Chinese Journal of Wildlife, 2015, 36(4): 378−381. doi: 10.3969/j.issn.1000-0127.2015.04.003
[6] Caballero A, Toro M A. Interrelations between effective population size and other pedigree tools for the management of conserved populations[J]. Genetics Research, 2000, 75(3): 331−343. doi: 10.1017/S0016672399004449
[7] Jimenez-Mena B, Schad K, Hanna N, et al. Pedigree analysis for the genetic management of group-living species[J]. Ecology and Evolution, 2016, 6(10): 3067−3078. doi: 10.1002/ece3.1831
[8] Liu G, Shafer A B A, Zimmermann W, et al. Evaluating the reintroduction project of Przewalski’s horse in China using genetic and pedigree data[J]. Biological Conservation, 2014, 171: 288−298. doi: 10.1016/j.biocon.2013.11.022
[9] 盛和林. 我国麝资源现状及救护措施[J]. 野生动物学报, 1996(3): 10−12. Sheng H L. Status and conservation measures of musk deer resources in China[J]. Chinese Journal of Wildlife, 1996(3): 10−12.
[10] 肖亮, 褚同邦. 全国野生动植物保护及自然保护区建设工程简介[J]. 生物学教学, 2003, 28(1): 53−54. doi: 10.3969/j.issn.1004-7549.2003.01.033 Xiao L, Chu T B. National wildlife protection and nature reserve construction project briefiIntroduction[J]. Biology Teaching, 2003, 28(1): 53−54. doi: 10.3969/j.issn.1004-7549.2003.01.033
[11] 蔡瑞波. 圈养林麝MHC基因和线粒体序列的遗传多样性[D]. 北京: 北京林业大学, 2016. Cai R B. Genetic diversity of MHC genes and mitochondrial sequences in captive forest musk deer (Moschus berezovskii)[D]. Beijing: Beijing Forestry University, 2016.
[12] 赵莎莎. 圈养林麝遗传多样性及泌香性能关联标记的分析研究[D]. 杭州: 浙江大学, 2009. Zhao S S. Assessment of genetic diversity in the captive forest musk deer (Moschus berezovskii) and linkage analysis between the performance of musk productivity and DNA molecular markers[D]. Hangzhou: Zhejiang University, 2009.
[13] 许冠. 林麝微卫星标记开发与圈养种群遗传多样性分析[D]. 杨凌: 西北农林科技大学, 2017. Xu G. Microsatellite development and genetic diversity analysis of Moschus berezovskii[D]. Yangling: Northwest A&F University, 2017.
[14] 刘刚, 徐超群, 何岚, 等. 圈养林麝线粒体D-Loop区序列多态性的粪便DNA分析[C]//中国遗传学会. 全国生物遗传多样性高峰论坛会刊. 昆明: 中国遗传学会, 2012. Liu G, Xu C Q, He L, et al. Fecal DNA analysis of mitochondrial D-Loop sequence polymorphism in captive forest musk deer[C]//Chinese Society of Genetics. Proceedings of the national biogenetic diversity summit forum. Kunming: Chinese Society of Genetics, 2012.
[15] Bhattacharyya S, Ishtiaq F. Noninvasive sampling reveals population genetic structure in the Royle’s pika, Ochotona roylei, in the western Himalaya[J]. Ecology and Evolution, 2019, 9(1): 180−191. doi: 10.1002/ece3.4707
[16] Bauer M L, Ferry B, Holman H, et al. Monitoring a new england cottontail reintroduction with noninvasive genetic sampling[J]. Wildlife Society Bulletin, 2020, 44(1): 110−121. doi: 10.1002/wsb.1069
[17] Oliveira M L D, Couto H T Z D, Duarte J M B. Distribution of the elusive and threatened Brazilian dwarf brocket deer refined by non-invasive genetic sampling and distribution modelling[J]. European Journal of Wildlife Research, 2019, 65(2): 1−9.
[18] Liu G, Zang S, Li L, et al. Evaluation of fecal DNA preservation and extraction methods in Przewalski’s horse[J]. Conservation Genetics Resources, 2014, 6(3): 511−513. doi: 10.1007/s12686-014-0162-3
[19] 夏霖, 杨奇森, 魏辅文, 等. 马麝诸种群地理分化初步探讨[J]. 兽类学报, 2004, 24(1): 1−5. doi: 10.3969/j.issn.1000-1050.2004.01.001 Xia L, Yang Q S, Wei F W, et al. Study on geographical division of Alpine musk deer (Moschus sifanicus)[J]. Acta Theriologica Sinica, 2004, 24(1): 1−5. doi: 10.3969/j.issn.1000-1050.2004.01.001
[20] Su Q, Yao Y, Li D, et al. The complete mitochondrial genome sequence of a wild forest musk deer (Moschus berezovskii)[J]. Conservation Genetics Resources, 2018, 10(3): 329−333. doi: 10.1007/s12686-017-0816-z
[21] Singh V K, Mangalam A K, Dwivedi S, et al. Primer premier: program for design of degenerate primers from a protein sequence[J]. Biotechniques, 1998, 2(24): 318−319.
[22] Hall T A. Bioedit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41: 95−98.
[23] Tamura K, Peterson D, Peterson N, et al. Mega5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731−2739. doi: 10.1093/molbev/msr121
[24] Rozas J, Sanchez-Delbarrio J C, Messeguer X, et al. Dnasp, DNA polymorphism analyses by the coalescent and other methods[J]. Bioinformatics, 2003, 19(18): 2496−2497. doi: 10.1093/bioinformatics/btg359
[25] Excoffier L, Lischer H E L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564−567. doi: 10.1111/j.1755-0998.2010.02847.x
[26] Bandelt H R, Forster P, Hl A R. Median-joining networks for inferring intraspecific phylogenies[J]. Molecular Biology and Evolution, 1999, 16(1): 37−48. doi: 10.1093/oxfordjournals.molbev.a026036
[27] 孙小雅, 张志和, 张文平, 等. 圈养大熊猫群体间的基因流状况分析[J]. 四川动物, 2010, 29(3): 333−339. Sun X Y, Zhang Z H, Zhang W P, et al. State of gene flow between captive giant pandas populations[J]. Sichuan Journal of Zoology, 2010, 29(3): 333−339.
[28] 何岚, 刘刚, 葛兴芳, 等. 圈养林麝应激反应的性别差异及其适应性[C]//中国动物学会. 第八届全国野生动物生态与资源保护学术研讨会论文集. 沈阳: 第八届全国野生动物生态与资源保护学术研讨会, 2012. He L, Liu G, Ge X F, et al. Sex difference and adaptability of stress response of captive forest musk deer[C]//China Zoological Society. Proceedings of the 8th National Symposium on Wildlife Ecology and Resource Conservation. Shenyang: 8th National Symposium on Wildlife Ecology and Resource Conservation, 2012.
[29] 李明, 魏辅文, 饶刚, 等. 非损伤性取样法在保护遗传学研究中的应用[J]. 动物学报, 2001, 47(3): 338−342. doi: 10.3321/j.issn:0001-7302.2001.03.016 Li M, Wei F W, Rao G, et al. Application of noninvasive sampling in conservation genetics[J]. Acta Zoologica Sinica, 2001, 47(3): 338−342. doi: 10.3321/j.issn:0001-7302.2001.03.016
[30] Peng H, Liu S, Zou F, et al. Genetic diversity of captive forest musk deer (Moschus berezovskii) inferred from the mitochondrial DNA control region[J]. Animal Genetics, 2009, 40(1): 65−72. doi: 10.1111/j.1365-2052.2008.01805.x
[31] 冯慧, 黄原, 任轶, 等. 陕西省林麝mtDNA D-loop区序列结构和种群遗传多样性[J]. 生态学报, 2014, 34(20): 5887−5895. Feng H, Huang Y, Ren Y, et al. Structure of the mitochondrial DNA D-Loop region and genetic diversity of Moschus berezovskii in Shaanxi Province[J]. Acta Ecologica Sinica, 2014, 34(20): 5887−5895.
[32] Neigel J E, Avise J C. Application of a random walk model to geographic distributions of animal mitochondrial DNA variation [J]. Genetics, 1993, 135(4): 1209−1220.
[33] Frankham R, Ballou J D, Briscoe D A. Introduction to conservation genetics[M]. Cambridge: Cambridge University Press, 2002.
[34] Wright S. Evolution and the genetics of populations [M]//Wright S. Variability within and among natural populations. Chicago: University of Chicago Press, 1978: 4.
[35] 李林海. 我国麝类动物养殖产业现状及对策研究[D]. 北京: 北京林业大学, 2012. Li L H. The status and developing strategies of musk deer farming industry in China[D]. Beijing: Beijing Forestry University, 2012.
-
期刊类型引用(3)
1. 毛林海,孔祥涛,梁璞,傅金和,许佳诺. 竹材物理力学性质影响因素研究进展. 世界竹藤通讯. 2024(02): 91-97 .  百度学术
百度学术
2. 贾舒予,王游,韦鹏练,马欣欣,吴谊民. 竹节结构及力学性能研究现状. 世界竹藤通讯. 2024(03): 90-99 .  百度学术
百度学术
3. 曹释予,张翔,季加贵,江甜,周雨砚,王雪花. 截面形态对竹条弯曲性能的影响. 家具. 2023(05): 33-37+116 .  百度学术
百度学术
其他类型引用(1)



 下载:
下载: