Construction of a yeast cDNA library using Centaurea cyanus petals of different colors
-
摘要:目的
矢车菊花瓣的蓝色呈色和品种间花色变异的分子调控机制尚不明晰。本研究采用Gateway技术构建了矢车菊6个不同花色品种花瓣的酵母cDNA文库,以期进一步通过酵母单杂交或双杂交技术筛选参与调控花瓣呈色的关键互作蛋白。
方法本研究以白色、粉色、红色、蓝色、紫色和墨色矢车菊花瓣为材料,提取总RNA后分离和纯化mRNA,合成双链cDNA后依次进行BP重组反应和LR重组反应,分别获得初级和次级文库。最后将次级文库质粒转化酵母Y187,获得矢车菊不同颜色花瓣的酵母cDNA文库。
结果质量鉴定结果显示:初级文库的库容量为1.3 × 107 CFU,重组率为100%,且插入片段长度均在1 000 bp以上;次级文库的库容量为1.6 × 107 CFU,重组率为100%,且插入片段长度均在1 000 bp以上。酵母文库的滴度为3.5 × 107 CFU/mL,随机挑选的24个单克隆经PCR检测后均扩增出明亮条带,重组率为100%,插入片段长度均大于1 000 bp。
结论本研究构建的矢车菊不同颜色花瓣酵母cDNA文库的质量较高,能满足酵母文库筛选的试验要求,为后续探究矢车菊花瓣的蓝色呈色和品种间花色变异的分子调控机制提供了材料基础。
Abstract:ObjectiveThe molecular regulation mechanism of both blue petal coloration and petal color variation among cornflower cultivars remains unclear. In order to further screen the key interaction proteins involved in regulating petal coloration by Y1H or Y2H method in the near future, the yeast cDNA library from cornflower petals of six cultivars with different colors was established by Gateway technology in the present study.
MethodThe white, pink, red, blue, mauve and black cornflower petals were used to extract total RNA, followed by mRNA isolation and purification. After the synthesis of double-strand cDNA, the BP recombination and LR recombination were performed to obtain the primary and secondary library, respectively. Finally, the plasmids from secondary library were transformed into yeast Y187 competent cells to build the yeast cDNA library from cornflower petals of different colors.
ResultThe quality identification of both the primary and the secondary library revealed that the library capacities were 1.3 × 107 CFU and 1.6 × 107 CFU, respectively, the recombination rate was both 100%, and the average length of insert fragment was both more than 1000 bp. After transforming into yeast, the obtained cDNA library titer was 3.5 × 107 CFU/mL. A total of 24 yeast clones were chosen randomly for PCR detection and showed bright bands, i.e., the recombinant rate was 100%. Moreover, the length of inserted cDNAs was longer than1000 bp.
ConclusionA high quality of yeast cDNA library using cornflower petals of different colors is established, satisfying the standard of yeast library screen, which will provide material basis for research the molecular mechanism of both the blue petal coloration and the petal color variation among cultivars in the near future.
-
Keywords:
- cornflower /
- flower color /
- yeast /
- cDNA library /
- Gateway technology
-
矢车菊(Centaurea cyanus)为菊科(Asteraceae)矢车菊属的一年生草本植物,兼具观赏、药用和食用价值。其头状花序小巧、繁密,是制作干燥花、花境和食品装饰的优选材料。此外,矢车菊花瓣提取物具有抗氧化、抗炎症和抗菌等特性,被广泛用作传统药材和茶类饮品[1]。
目前关于矢车菊的研究多聚焦于花瓣呈色的相关机制上。自然界多数积累矢车菊素的物种仅呈现不同程度的红色花,但蓝色矢车菊中的矢车菊素却能形成鲜艳亮丽的蓝色,这使其成为研究蓝色呈色机制的好材料。已有研究表明,矢车菊的蓝色花源于6分子矢车菊素、6分子芹菜素、1分子Fe3+、1分子Mg2+和2分子Ca2+组成的蓝色超分子色素[2]。前人在蓝色矢车菊中分离得到了液泡铁转运基因CcVIT,其异源表达后能使△CCC1酿酒酵母在高浓度Fe2+溶液中正常生长,而紫色矢车菊突变体的CcVIT蛋白发生了一处氨基酸的替换,其在酿酒酵母中异源表达后,铁转运活性降低,据此推测CcVIT可能在矢车菊素蓝色呈色过程中发挥了重要作用[3]。除珍贵的蓝色系外,矢车菊在不同花色品种间表现出丰富的花色变异,其中白色系不积累花青素,粉色和红色系只积累天竺葵素衍生物,而紫色和墨色系只积累矢车菊素衍生物[4]。进一步借助转录组测序技术在矢车菊中挖掘了一系列参与花青素生物合成的结构基因和调节基因,并通过烟草叶片瞬时表达、双荧光素酶、双分子荧光互补、酵母双杂交等技术鉴定了正向调控花青素生物合成的转录因子CcMYB6-1和CcbHLH1[5]。虽然目前已在矢车菊中挖掘了参与色素合成、铁离子转运等的相关基因,但其花瓣呈色的遗传调控机制仍不完全明晰。因此,系统地挖掘矢车菊花瓣呈色的相关基因仍是关键所在。
酵母单杂交和双杂交系统是筛选靶蛋白与诱饵分子之间相互作用的常用方法,其中酵母单杂交系统用于分析DNA与蛋白质间的相互作用,而酵母双杂交系统用于分析蛋白质与蛋白质之间的相互作用[6−7]。酵母cDNA文库是挖掘功能基因的有效方法,在该文库的基础上进一步运用酵母双杂交或单杂交文库系统可筛选出关键候选基因。目前已在金柑(Fortunella crassifolia)、月季(Rosa hybrida)、山葡萄(Vitis amurensis)、香蕉(Musa acuminata)、大花蕙兰(Cymbidium faberi)、百合(Lilium spp.)、芍药(Paeonia lactiflora)、烟草(Nicotiana tabacum)和牡丹(Paeonia suffruticosa)等多种园艺植物中构建了酵母cDNA文库,协助解析花发育、逆境胁迫、果实成熟和色素合成等生命过程的分子调控机制[7−15]。然而,矢车菊的酵母cDNA文库尚未见报道。因此,构建高容量、高质量的酵母cDNA文库对矢车菊的相关研究具有重要的实际意义。
Clontech公司的SMART技术和Invitrogen公司的Gateway技术是在构建酵母cDNA文库时常用的2种方法。其中SMART技术使用0.025 ~ 1.000 µg mRNA,即可稳定获得全长cDNA[16−17];而Gateway技术的RNA起始用量最低为200 µg,在构建过程中cDNA不受限制性酶的酶切影响,可较好地保证cDNA的完整性[13]。本研究以矢车菊6个不同花色品种的花瓣为试验材料,在mRNA充足的条件下利用Gateway技术构建了酵母cDNA文库,以期在后续研究中通过酵母单杂交或双杂交筛库技术筛选参与花色形成的关键候选基因,从而为研究矢车菊花瓣呈色的遗传调控机制提供材料基础。
1. 材料与方法
1.1 试验材料
以矢车菊(Centaurea cyanus)6个不同花色品种的花瓣为试验材料,包括矮汤姆系列的白色(‘Dwarf Tom Pouce White’,DTPW)、粉色(‘Dwarf Tom Pouce Pink’,DTPP)、红色(‘Dwarf Tom Pouce Red’,DTPR)和蓝色(‘Dwarf Tom Pouce Blue’,DTPB)品种;高双球系列的紫色(‘Tall Double Ball Mauve’,TDBM)和墨色(‘Tall Double Ball Black’,TDBB)品种。将其播种于V草炭:V蛭石 = 1∶1的无土栽培基质,置于16 h光照和8 h黑暗的光照培养箱中,待开花后,按照前期对矢车菊不同品种不同发育阶段的划分标准进行取样[4],其中花瓣发育阶段的具体划分标准如下:S1,花瓣未着色;S2,花瓣开始着色,且着色区占整个花瓣的1/3;S3,花瓣完全着色,但未展开;S4,花瓣完全着色,且呈开展状态。将每个花色品种4个不同发育阶段的花瓣进行混合取样,置于液氮中预冷,随后保存于−80 ℃冰箱中备用。
华越洋植物RNA提取试剂盒购自于北京华越洋生物科技有限公司,CloneMiner Ⅱ cDNA文库构建试剂盒、UltraPureTM琼脂糖、PlatinumTM Taq DNA Plolymearse、dNTP(100 mmol/L)、PureLinkTM快速凝胶提取和PCR纯化试剂盒、PureLinkTM HQ Mini质粒DNA纯化试剂盒、UltraPureTM苯酚∶氯仿∶异戊醇(体积比25∶24∶1)均购自于Invitrogen公司,分子生物学试验专用醋酸铵溶液(5 mol/L)购自于Sigma公司。
1.2 研究方法
1.2.1 花瓣总RNA提取与mRNA分离
使用华越洋植物RNA提取试剂盒提取矢车菊6个不同花色品种不同发育阶段的花瓣中的总RNA,具体方法参照说明书。随后使用琼脂糖凝胶电泳和NanoDrop(Agilent)检测RNA提取质量。在此基础上使用Oligotex mRNA Midi Kit试剂盒分离纯化mRNA,并经1%琼脂糖凝胶电泳检测纯化质量。
1.2.2 初级cDNA文库的构建与质量鉴定
以1.2.1中分离纯化的mRNA为模板,参考CloneMiner说明书依次进行cDNA第一、二条链的合成。获得双链cDNA后,将其连接于3种三框attB1重组接头,分级分离后在Gateway® BP Clonase® Ⅱ酶促反应液的催化下将其连接至pDONRTM222载体,随后将重组质粒转入大肠杆菌DH10B,加入4 mL SOC培养基后于37 ℃摇床中230 r/min振荡培养,获得初级文库菌液。为鉴定文库容量,吸取10 μL培养物稀释100倍后涂布于50 mg/L卡那霉素的LB固体平板上,37 ℃过夜培养后,统计平板上的克隆数。为鉴定重组率和插入片段的长度,随机挑取24个单克隆进行菌液PCR,反应体系为:于每个克隆中加入2.0 μL 10 × PCR Buffer,0.5 μL dNTP (10 mmol/L),0.5 μL pDONR222F(5′-GTAAAACGACGGCCAG-3′, 20 μmol/L),0.5 μL pDONR222R(5′-CAGGAAACAGCTATGAC-3′,20 μmol/L),0.3 μL DNA Polymerase(5 U/μL),16.2 μL ddH2O。反应程序为:94 ℃ 5 min;94 ℃ 30 s,60 ℃ 30 s,72 ℃ 2 min,循环25次;72 ℃ 5 min。随后经1%琼脂糖凝胶电泳鉴定PCR产物。
1.2.3 次级文库的构建与质量鉴定
抽提1.2.2中验证合格的初级文库的质粒,将其稀释至300 ng/μL后,在Gateway® LR Clonase® Ⅱ酶促反应液的催化下进行LR重组反应,随后将LR重组质粒转化大肠杆菌DH10B,37 ℃、230 r/min振荡培养后获得次级文库菌液。为鉴定次级文库的质量,取10 μL菌液并稀释100倍,涂布于含100 mg/L氨苄霉素的LB固体平板,37 ℃过夜培养后统计克隆数。进一步使用菌液PCR技术鉴定重组率和插入片段长度,反应体系和反应程序同1.2.2,其中所用的引物为pGADT7-DEST-F(5′-TAATACGACTCACTATAGGGCGAGCGCCGCCATG-3′)和pGADT7-DEST-R(5′-GTGAACTTGCGGGGTTTTTCAGTATCTACGATT-3′)。随后经1%琼脂糖凝胶电泳鉴定PCR产物。
1.2.4 酵母文库的构建
取5 μg次级文库质粒、20 μL预变性的Carrier DNA、600 μL酵母(Y187)感受态细胞,涡旋振荡混合后加入2.5 mL 1 × PEG/LiAc,混合后于30 ℃水浴锅中孵育45 min(每15 min混匀一次),加入160 μL DMSO,42 ℃热激20 min(每10 min上下颠倒混匀一次),700 g离心5 min,随后去上清液,加入1 mL YPD Plus液体培养基重新悬浮,于30 ℃振荡复苏培养1.5 h。离心后弃上清液,加入30 mL 0.9% NaCl溶液重新悬浮细胞,取100 μL悬浮液分别稀释10、100、1 000、10 000倍后涂布于SD/-Leu固体平板,30 ℃培养3 ~ 5 d后收集转化子,并使用菌液PCR鉴定阳性克隆,反应体系为:1.0 μL菌液,2.5 μL 10 × Advantage 2 PCR Buffer,0.5 μL 50 × dNTP Mix,0.5 μL T7SP Primer(5′-TAATACGACTCACTATAGGGC-3′),0.5 μL 3′AD Primer(5′-AGATGGTGCACGATGCACAG-3′),0.5 μL 50 × Advantage 2 Polymerase Mix,19.5 μL ddH2O。反应程序为:94 ℃ 3 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 2 min,循环35次;72 ℃ 5 min。随后经1%琼脂糖凝胶电泳鉴定PCR产物。取阳性克隆进行测序,并使用NCBI在线网站(https://blast.ncbi.nlm.nih.gov/Blast.cgi?PROGRAM=blastx&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome)对测序结果进行比对和分析。
1.3 数据统计
在构建酵母文库过程中涉及的文库容量、重组率、文库滴度的计算公式如下。
C=(N1/V1)AV2 (1) 式中:C表示文库库容量,CFU;N1表示平板上的克隆数;V1表示涂布平板时的菌液体积,本研究中为50 μL;A表示稀释倍数,100倍;V2表示文库菌液总体积,本研究中为4 000 μL。
R=y/Y×100% (2) 式中:R为重组率,%;y为重组成功的克隆数;Y为检测的总克隆数。
T=(N2/V3)B (3) 式中:T表示文库滴度,CFU/mL;N2表示平板上的克隆数;V3表示涂布平板时的菌液体积,本研究中为0.1 mL;B表示稀释倍数,10 000倍。
2. 结果与分析
2.1 矢车菊不同花色品种花瓣RNA的质量鉴定
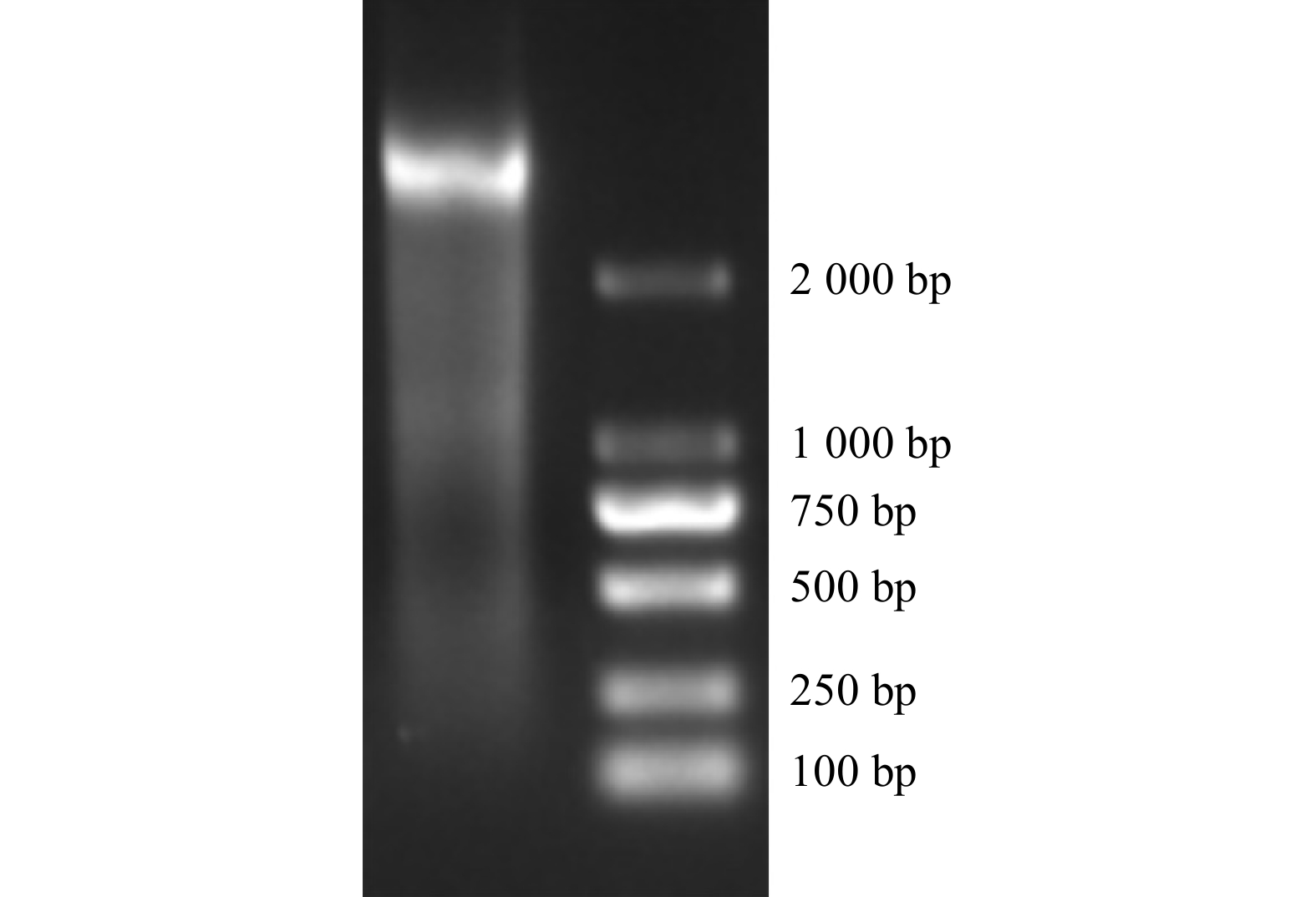
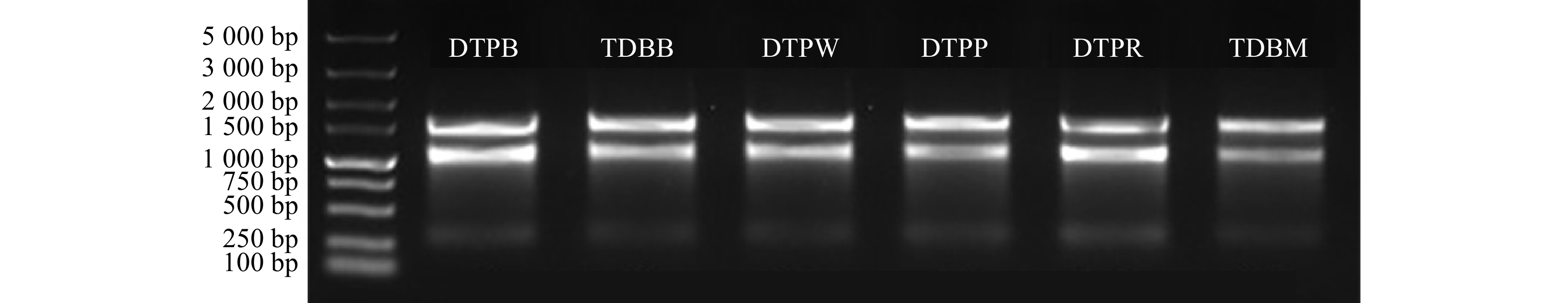
以蓝色、墨色、白色、粉色、红色和紫色矢车菊品种不同发育阶段的花瓣为试验材料进行总RNA提取,1%琼脂糖凝胶电泳结果显示,各花色品种的RNA主带清晰,无明显降解(图1)。Nanodrop检测结果显示,RNA质量浓度区间为525.30 ~ 1167.50 ng/μL(表1),说明RNA提取质量较好,可以满足后续试验需求。本研究构建混合文库,将DTPB、TDBB、DTPW、DTPP、DTPR和TDBM的初始总RNA进行混合,并分离和纯化mRNA。经1%琼脂糖凝胶电泳后,在2 000 bp以上处获得明亮条带,说明分离的mRNA质量较好(图2)。
表 1 矢车菊6个不同花色品种花瓣中的总RNA质量Table 1. Total RNA quality of cornflower petals from six cultivars of different colors品种
Cultivar质量浓度
Mass concentration/(ng·μL−1)A260/A280 DTPB 547.60 2.15 TDBB 715.70 2.13 DTPW 525.30 2.13 DTPP 527.80 2.20 DTPR 1151.60 2.14 TDBM 1167.50 2.09 2.2 cDNA第二链合成结果
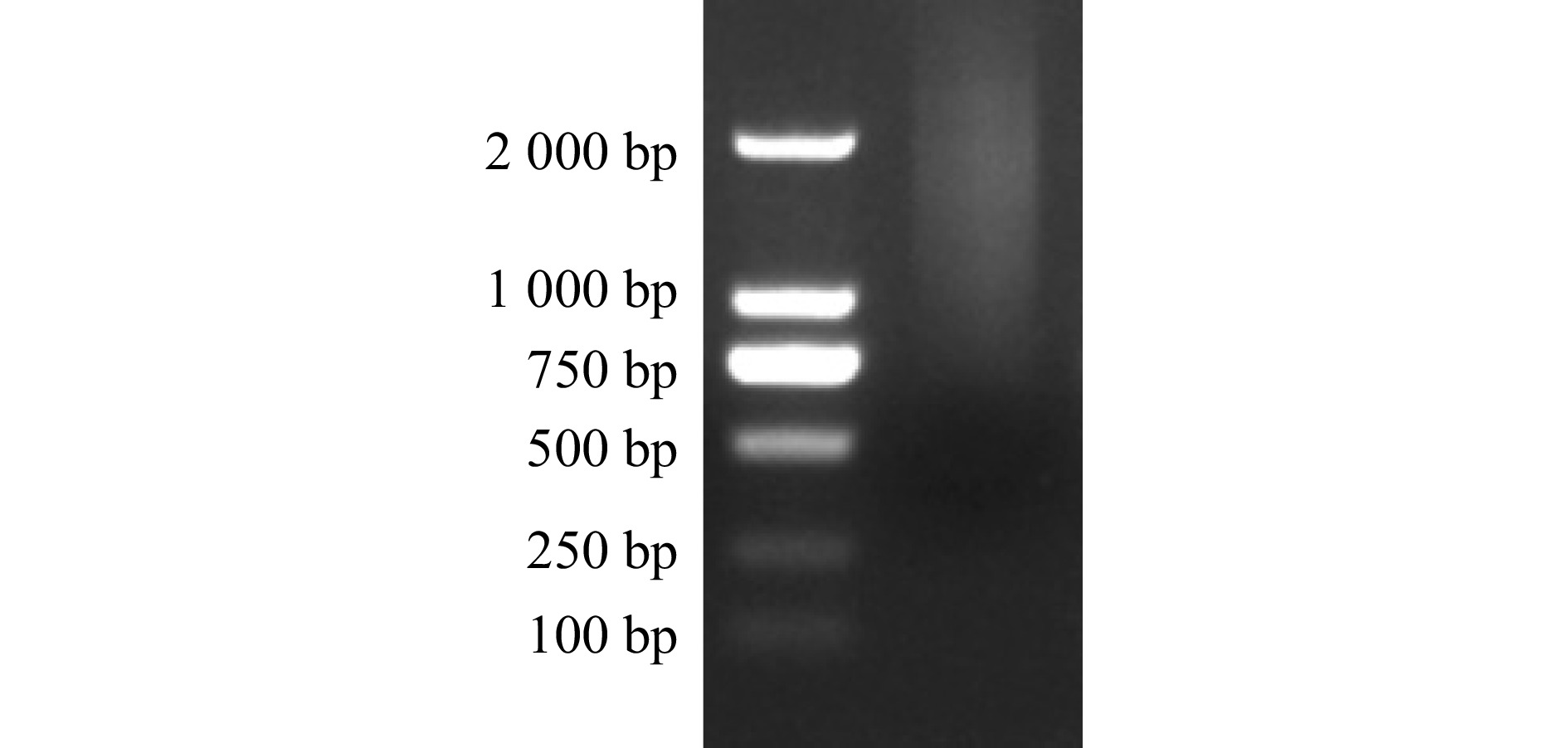
以分离纯化的mRNA为模板依次合成cDNA第一和第二条链,并使用琼脂糖凝胶电泳检测双链cDNA的质量。图3显示:双链cDNA的条带呈弥散状,cDNA长度在750 bp以上。这表明cDNA第二链的合成效果较好,可进一步构建cDNA文库质粒。
2.3 初级文库的构建质量
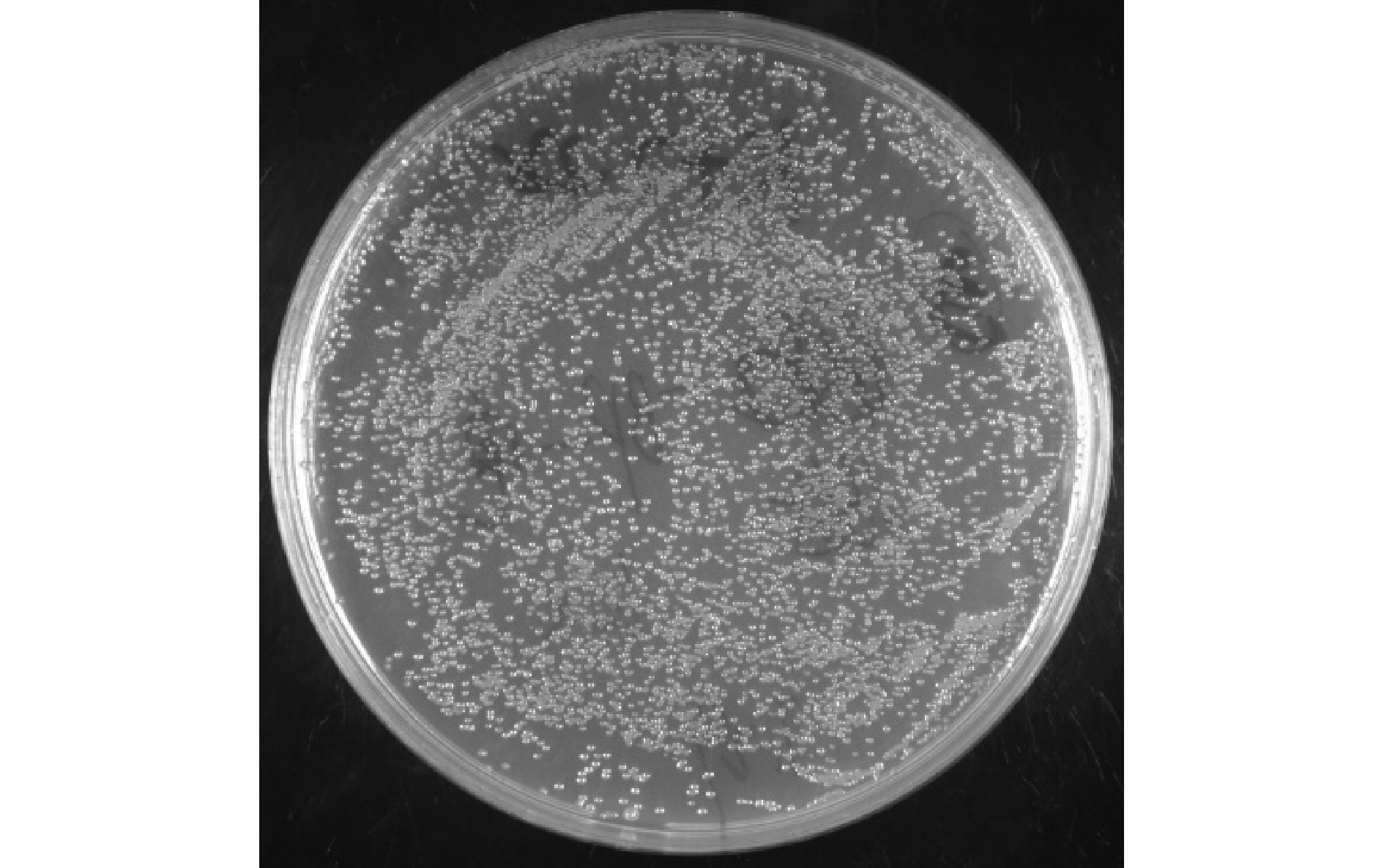
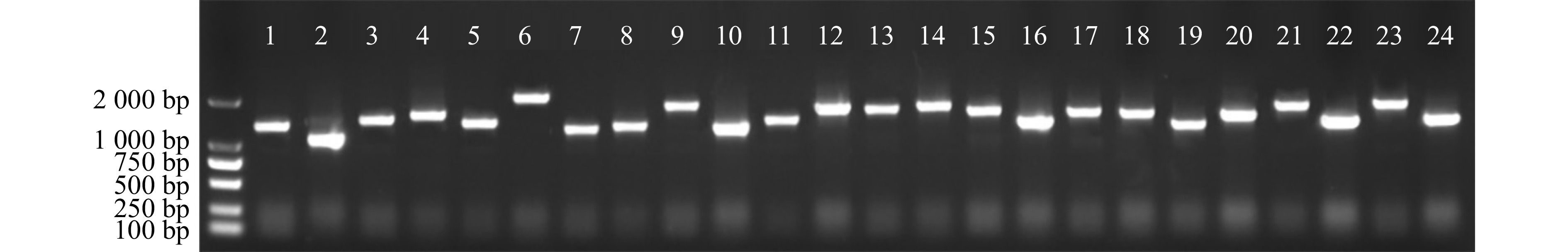
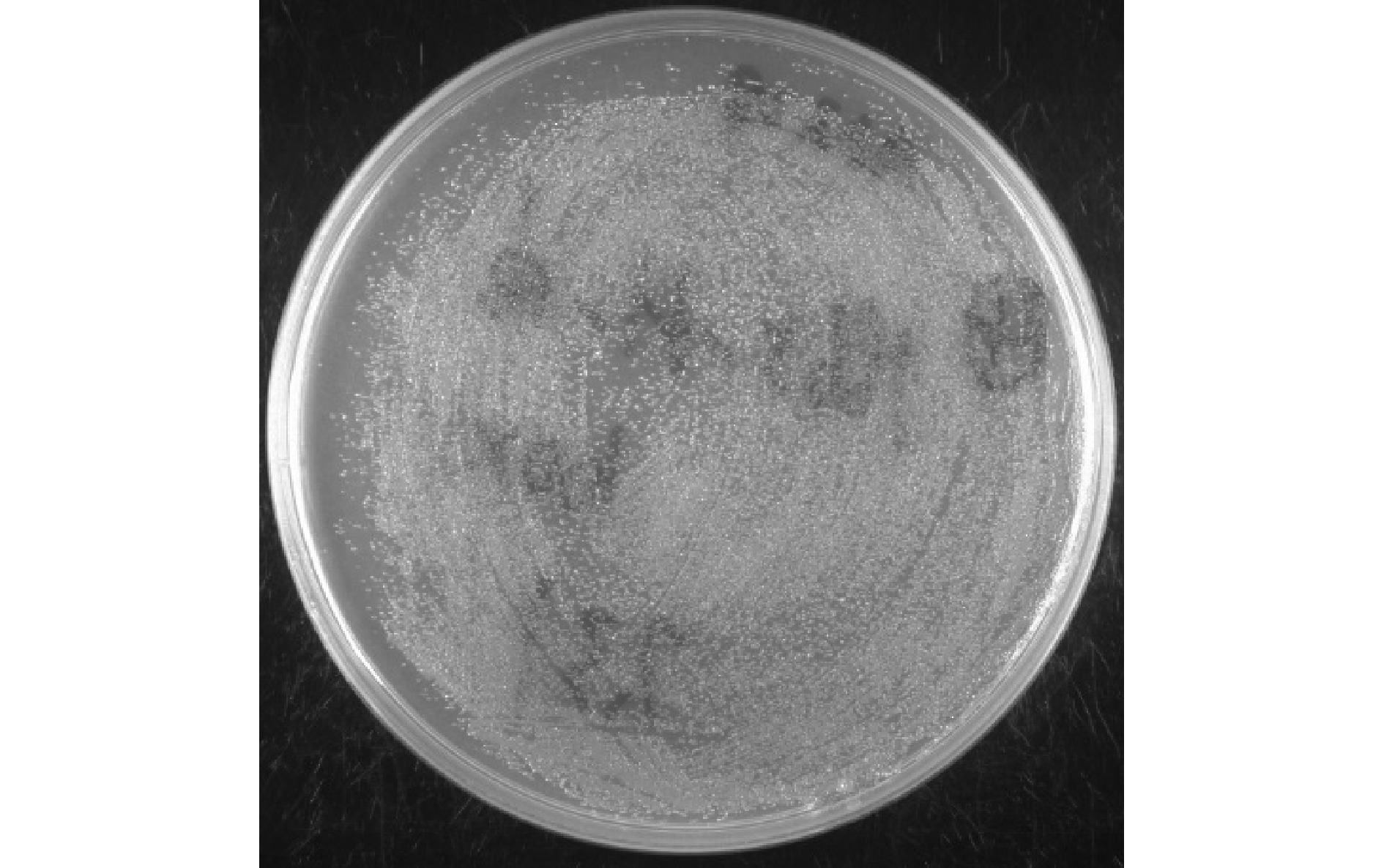
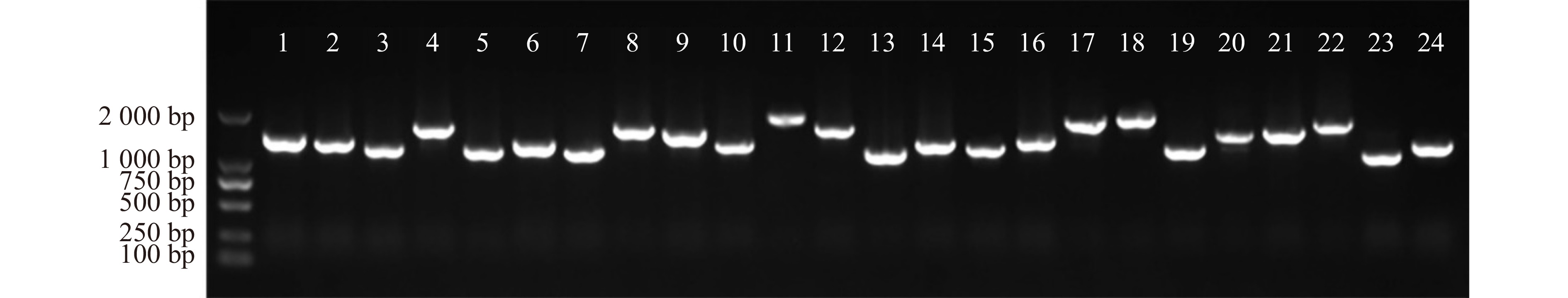
在合成cDNA第二链后,与3种三框attB1重组接头相连接,进行BP重组反应,并将重组质粒转入大肠杆菌DH10B,获得初级文库。将初级文库菌液稀释后涂布,37 ℃过夜培养后,统计固体平板上的克隆数为1 600个(图4),根据公式(1)得到,矢车菊初级文库的库容量为1.3 × 107 CFU。进一步随机挑取24个单克隆进行菌液PCR,以鉴定重组率和插入片段长度。所挑取的24个单克隆均扩增出亮条带,根据公式(2)得到重组率为100%,且插入片段长度均在1 000 bp以上(图5),符合初级文库的质量要求。
2.4 次级文库的构建质量
抽提初级文库的质粒进行LR重组反应,并将LR重组质粒转化大肠杆菌DH10B后获得次级文库。统计LB固体平板上的克隆数为2 000个(图6),根据公式(1)得到,矢车菊次级文库的库容量为1.6 × 107 CFU。随后,随机挑取24个单克隆进行菌液PCR,琼脂糖凝胶电泳结果显示,24个单克隆均扩增出目标条带,根据公式(2)得到重组率为100%,且插入片段长度均在1 000 bp以上(图7)。由此可见,次级文库的库容量大,连接效率高,能满足后续试验需求。
2.5 酵母cDNA文库的构建与鉴定
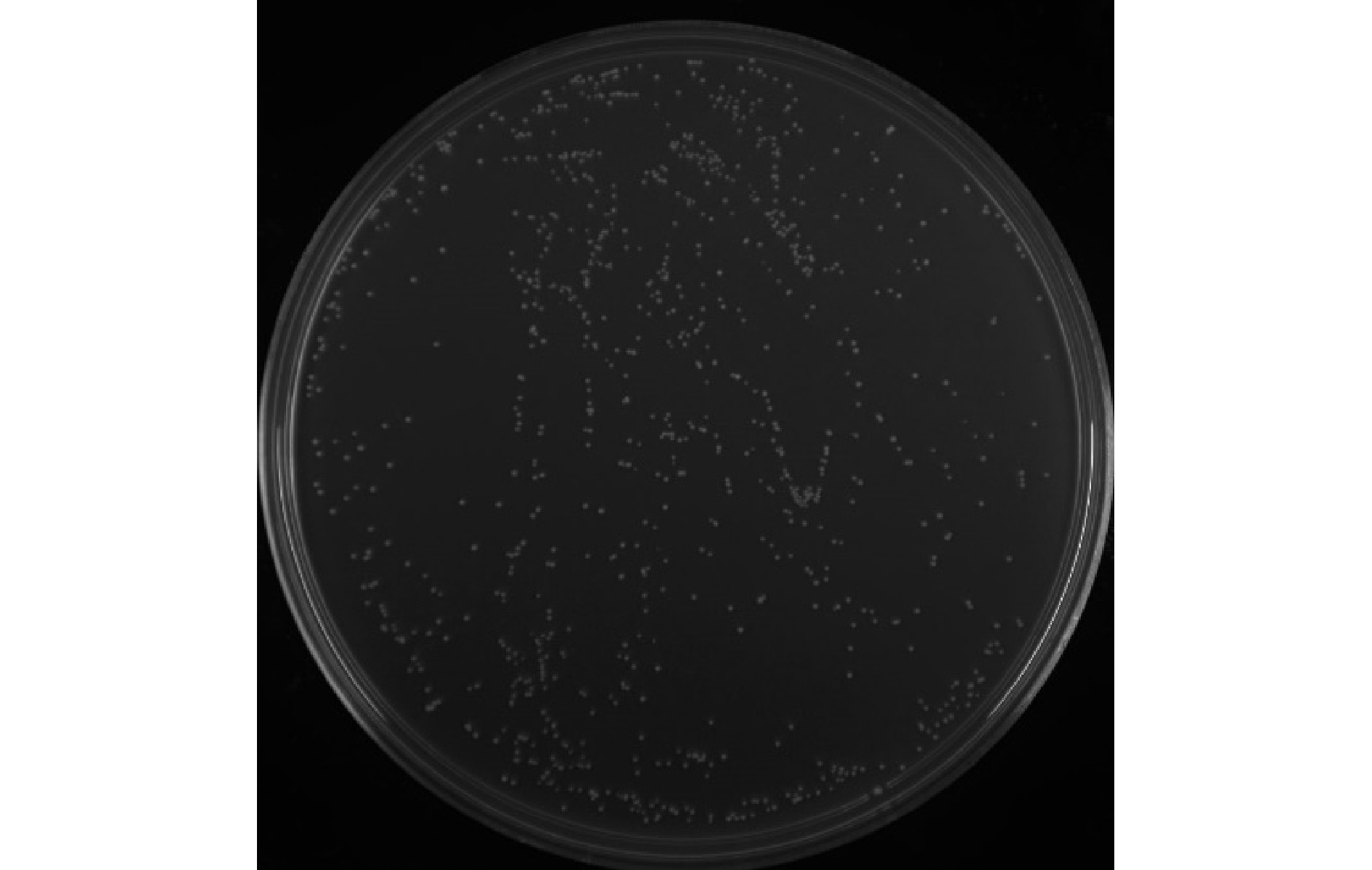
抽提次级文库质粒并转化酵母Y187感受态细胞,涂布于SD/-Leu平板上,2 ~ 3 d后观察,发现涂布10 000倍稀释液的平板上的克隆数为350个(图8),代入公式(3)计算,酵母cDNA文库的滴度为3.5 × 107 CFU/mL。随机挑选24个单克隆进行菌液PCR鉴定,均扩增出条带,根据公式(2)得到文库重组率为100%,插入片段长度均在1 000 bp以上(图9)。
随机选取24个克隆进行测序分析,并使用在线网站NCBI进行序列比对,从而获得序列的功能注释信息(表2)。除9号和10号为假定蛋白外,其余22个阳性单克隆均具有明确的基因功能,包括2个转录因子(12号和18号)和20个结构蛋白。值得注意的是,获得了4个可能参与类黄酮生物合成的结构蛋白,包括2个查尔酮合成酶(14号和20号)与2个糖基转移酶(5号和15号)。
表 2 酵母文库cDNA插入序列的功能注释Table 2. Functional annotation of inserted cDNA sequence in the yeast library阳性克隆
Positive clone同源基因登录号
Accession No. of homologous gene功能注释
Functional annotation1 XP_022002415.1 非特异性转脂蛋白 Non-specific lipid-transfer protein-like 2 PWA83678.1 生长素诱导蛋白 Auxin-induced protein AUX28 3 XP_024984456.1 光合系统 Ⅰ叶绿素 a/b结合蛋白 Photosystem Ⅰ chlorophyll a/b-binding protein 4 XP_024968924.1 突触融合蛋白 Syntaxin 5 XP_024966033.1 糖基转移酶 UDP-glycosyltransferase 6 XP_024984844.1 超长链烯酰辅酶 A还原酶 Very-long-chain enoyl-CoA reductase 7 XP_024973168.1 丝氨酸羟甲基转移酶 Serine hydroxymethyltransferase 8 XP_024994031.1 水通道蛋白 Aquaporin 9 KAI3748566.1 假定蛋白 Hypothetical protein 10 KAJ9551764.1 假定蛋白 Hypothetical protein 11 XP_043607664.1 糖激酶 Sugar kinase 12 XP_043613764.1 细胞数目调节因子 Cell number regulator 13 XP_043617839.1 醇酰基转移酶 Alcohol acyltransferase 14 UYR41662.1 查尔酮合成酶 Chalcone synthase 15 KVI07143.1 葡萄糖基转移酶 UDP-glucosyltransferase 16 XP_024987875.1 山梨醇脱氢酶 Sorbitol dehydrogenase 17 UQU55953.1 脂肪酸去饱和酶 Fatty acid desaturase 18 KAJ0622779.1 转录因子 C2H2家族 Transcription factor C2H2 family 19 XP_024967747.1 伴侣蛋白 Chaperone protein 20 ALG75882.1 查尔酮合成酶 Chalcone synthase 21 XP_043617710.1 营养细胞壁蛋白 Vegetative cell wall protein 22 KVI02816.1 丙二烯氧化物环氧化酶 Allene oxide cyclase 23 XP_052603629.1 调节突触膜胞吐蛋白 Regulating synaptic membrane exocytosis protein 24 KAG5222336.1 液泡蛋白分选相关蛋白 Vacuolar protein sorting-associated protein 3. 结论与讨论
花色是园林花卉的重要观赏性状之一,新奇、鲜丽的花色也是重要的育种目标。矢车菊不仅具有珍贵的蓝色系,还具有白色、粉色、红色、紫色和墨色等丰富的花色变异,这使其成为研究蓝色呈色和品种间花色变异机制的极佳材料。高等植物的器官呈色受细胞pH值、金属离子和色素合成等多种因素的影响,每一环节都涉及复杂的基因调控过程[18−20]。一个基因的编码产物常与其他蛋白或DNA发生互作后形成复合体,从而精密调控另一个基因的表达。利用酵母单杂交或双杂交系统高通量地筛选靶蛋白与诱饵分子间的相互作用,是快速鉴定分子调控网络的有效方法。而构建高质量的酵母cDNA文库是进行酵母单杂交或酵母双杂交文库筛选的前提,是挖掘功能基因的常用方法[21]。
构建高质量文库首先要获取高纯度、高完整度和高浓度的mRNA。本研究以矢车菊6个不同花色品种4个不同发育阶段的花瓣为试验材料提取总RNA,最大范围地涵盖了参与花色发育的全部基因资源。此外,酵母cDNA文库的重组率、插入片段长度和滴度等均会影响文库质量。通常cDNA文库的库容量应大于1 × 106 CFU、文库滴度大于1.7 × 106 CFU/mL时才能满足后续使用要求[13, 22]。以大花蕙兰(Cymbidium faberi)不同组织、不同花发育阶段为试验材料构建的酵母文库中,重组率为95%,插入片段长度为750 ~ 3 000 bp,库容量为6 × 109 CFU/µg[7]。而以百合(Lilium spp.)花瓣为试验材料构建的酵母文库的重组率为98%,插入片段长度为400 ~ 2 000 bp,库容量为1.6 × 106 CFU[12]。本研究构建的矢车菊不同颜色花瓣酵母文库的重组率为100%,插入片段长度均大于1 000 bp,滴度为3.5 × 107 CFU/mL,可以高质量地保证后期酵母单杂交或双杂交试验的正常开展,高效筛选参与花色形成的关键候选基因。
近年来,酵母单杂交或双杂交筛库技术已成功应用于高效解析高等植物器官呈色的分子调控网络上。一方面,在已构建的酵母文库的基础上,以目的基因的启动子序列为诱饵进行酵母单杂交筛库,可有效挖掘其上游调控基因。研究者以菊花(Chrysanthemum × morifolium)类胡萝卜素合成基因CmCCD4a-2的启动子序列为诱饵,经酵母单杂交筛库后成功挖掘到了上游转录因子CmAP3和CmUIF1,进一步结合酵母双杂交、双分子荧光互补、酵母三杂交等技术鉴定了调控菊花类胡萝卜素代谢的CmAP3-CmPI-CmUIF1蛋白复合体[23]。在百合中,以花青素生物合成激活子LhMYBSPLATTER基因的启动子为诱饵,经酵母单杂交筛库后获得与之互作的7个靶蛋白,包括BTF3、MYB4、IAA6-like、ERF4、ARR1、ERF WIN1-like、ERF061[12]。在苹果(Malus × domestica)中采用相同的策略获得了能够直接结合MdGSTF6启动子序列,并激活其表达的MdMYB1,参与苹果花青素的转运过程[24]。除转录激活子以外,也可以通过酵母单杂交筛库获得转录抑制子。例如:在菊花中以CmFNS启动子序列为诱饵进行筛库后,获得了抑制该基因表达的CmMYB012,进一步研究发现,该抑制子还能通过抑制花青素生物合成结构基因CmCHS、CmDFR、CmANS和CmUFGT的表达,降低花瓣中花青素的积累量[25]。另一方面,以目的基因编码的蛋白为诱饵进行酵母双杂交筛库,可有效获得与之互作的协同调控蛋白。例如:在苹果中以茉莉酮酸酯ZIM结构域蛋白MdJAZ2为诱饵,经酵母双杂交筛库后成功挖掘了互作蛋白MdHIR2和MdHIR4,其共同抑制苹果果皮中花青素的生物合成[26]。研究者利用酵母双杂交筛库技术发现:与光形态建成相关的转录因子SlBBX20可与COP9信号转导体亚基SlCSN5-2互作以激活SlDFR的表达,从而调控花青素生物合成过程[27]。在红薯(Ipomoea batatas)中以IbNAC29蛋白为诱饵,经酵母双杂交筛库后获得与之互作的R1型MYB蛋白IbMYB1R1,并进一步以其为诱饵获得与之互作的IbAITR5蛋白,经酵母三杂交鉴定发现,IbNAC29-IbMYB1R1-IbAITR5可形成调控模块,参与类胡萝卜素的生物合成[28]。
本课题组在前期研究中使用一系列分子生物学技术挖掘了可能参与矢车菊花瓣呈色的候选基因,包括参与色素生物合成的结构基因及其调节基因、金属离子转运和储藏的相关基因等[4−5],但其具体的分子调控机制尚不完全明晰。在后续研究中将以本研究构建的酵母文库为材料基础,进一步通过酵母单杂交或双杂交筛库技术充分挖掘互作蛋白,构建调控矢车菊花瓣呈色的分子网络。综上所述,本研究以矢车菊6个不同花色品种不同发育阶段的花瓣为材料,构建出了高质量的酵母cDNA文库,这为后续筛选参与蓝色呈色和品种间花色变异的互作蛋白提供了材料基础。
-
表 1 矢车菊6个不同花色品种花瓣中的总RNA质量
Table 1 Total RNA quality of cornflower petals from six cultivars of different colors
品种
Cultivar质量浓度
Mass concentration/(ng·μL−1)A260/A280 DTPB 547.60 2.15 TDBB 715.70 2.13 DTPW 525.30 2.13 DTPP 527.80 2.20 DTPR 1151.60 2.14 TDBM 1167.50 2.09 表 2 酵母文库cDNA插入序列的功能注释
Table 2 Functional annotation of inserted cDNA sequence in the yeast library
阳性克隆
Positive clone同源基因登录号
Accession No. of homologous gene功能注释
Functional annotation1 XP_022002415.1 非特异性转脂蛋白 Non-specific lipid-transfer protein-like 2 PWA83678.1 生长素诱导蛋白 Auxin-induced protein AUX28 3 XP_024984456.1 光合系统 Ⅰ叶绿素 a/b结合蛋白 Photosystem Ⅰ chlorophyll a/b-binding protein 4 XP_024968924.1 突触融合蛋白 Syntaxin 5 XP_024966033.1 糖基转移酶 UDP-glycosyltransferase 6 XP_024984844.1 超长链烯酰辅酶 A还原酶 Very-long-chain enoyl-CoA reductase 7 XP_024973168.1 丝氨酸羟甲基转移酶 Serine hydroxymethyltransferase 8 XP_024994031.1 水通道蛋白 Aquaporin 9 KAI3748566.1 假定蛋白 Hypothetical protein 10 KAJ9551764.1 假定蛋白 Hypothetical protein 11 XP_043607664.1 糖激酶 Sugar kinase 12 XP_043613764.1 细胞数目调节因子 Cell number regulator 13 XP_043617839.1 醇酰基转移酶 Alcohol acyltransferase 14 UYR41662.1 查尔酮合成酶 Chalcone synthase 15 KVI07143.1 葡萄糖基转移酶 UDP-glucosyltransferase 16 XP_024987875.1 山梨醇脱氢酶 Sorbitol dehydrogenase 17 UQU55953.1 脂肪酸去饱和酶 Fatty acid desaturase 18 KAJ0622779.1 转录因子 C2H2家族 Transcription factor C2H2 family 19 XP_024967747.1 伴侣蛋白 Chaperone protein 20 ALG75882.1 查尔酮合成酶 Chalcone synthase 21 XP_043617710.1 营养细胞壁蛋白 Vegetative cell wall protein 22 KVI02816.1 丙二烯氧化物环氧化酶 Allene oxide cyclase 23 XP_052603629.1 调节突触膜胞吐蛋白 Regulating synaptic membrane exocytosis protein 24 KAG5222336.1 液泡蛋白分选相关蛋白 Vacuolar protein sorting-associated protein -
[1] Fernandes L, Pereira J A, Saraiva J A, et al. Phytochemical characterization of Borago officinalis L. and Centaurea cyanus L. during flower development[J]. Food Research International, 2019, 123: 771−778. doi: 10.1016/j.foodres.2019.05.014
[2] Shiono M, Matsugaki N, Takeda K. Structure of the blue cornflower pigment[J]. Nature, 2005, 436: 791. doi: 10.1038/436791a
[3] Yoshida K, Negishi T. The identification of a vacuolar iron transporter involved in the blue coloration of cornflower petals[J]. Phytochemistry, 2013, 94: 60−67. doi: 10.1016/j.phytochem.2013.04.017
[4] Deng C Y, Li S S, Feng C Y, et al. Metabolite and gene expression analysis reveal the molecular mechanism for petal colour variation in six Centaurea cyanus cultivars[J]. Plant Physiology and Biochemistry, 2019, 142: 22−33. doi: 10.1016/j.plaphy.2019.06.018
[5] Deng C Y, Wang J Y, Lu C F, et al. CcMYB6-1 and CcbHLH1, two novel transcription factors synergistically involved in regulating anthocyanin biosynthesis in cornflower[J]. Plant Physiology and Biochemistry, 2020, 151: 271−283. doi: 10.1016/j.plaphy.2020.03.024
[6] Sun Y, Li Y, Huang G, et al. Application of the yeast one-hybrid technique to plant functional genomics studies[J]. Biotechnology and Biotechnological Equipment, 2017, 31(6): 1087−1092. doi: 10.1080/13102818.2017.1378595
[7] Xu Y, Zhou J, Liu Q, et al. Construction and characterization of a high-quality cDNA library of Cymbidium faberi suitable for yeast one- and two-hybrid assays[J]. BMC Biotechnology, 2020, 20: 4. doi: 10.1186/s12896-020-0599-2
[8] 苏玲, 李彬, 王青, 等. 金柑花蕾酵母双杂交cDNA文库构建及评价[J]. 基因组学与应用生物学, 2019, 38(7): 3169−3173. Su L, Li B, Wang Q, et al. Construction and evaluation of yeast two hybrid cDNA library of floral buds from Rongan Kumquat[J]. Genomics and Applied Biology, 2019, 38(7): 3169−3173.
[9] 张竹君, 任文娟, 耿子雯, 等. 月季酵母双杂交cDNA文库的构建及RhRD22互作蛋白的筛选[J]. 分子植物育种, 2021, 19(7): 2243−2255. Zhang Z J, Ren W J, Geng Z W, et al. Construction of a high-quality yeast two-hybrid cDNA library and screening of the proteins interacting with RhRD22 of rose[J]. Molecular Plant Breeding, 2021, 19(7): 2243−2255.
[10] 马登辉, 马红义, 李承男, 等. 低温胁迫下山葡萄叶片酵母cDNA文库构建[J]. 福建农业学报, 2022, 37(9): 1176−1181. Ma D H, Ma H Y, Li C N, et al. Construction of yeast cDNA library using Vitis amurensis leaves under low-temperature stress[J]. Fujian Journal of Agricultural Sciences, 2022, 37(9): 1176−1181.
[11] 王川, 盛鸥, 窦同心, 等. 香蕉果实酵母双杂交文库的构建及后熟转录因子MaMADS2互作蛋白筛选[J]. 南京农业大学学报, 2023, 46(2): 237−246. Wang C, Sheng O, Dou T X, et al. Construction of yeast two-hybrid library and screening of interacting proteins of ripening regulator MaMADS2 in banana fruit[J]. Journal of Nanjing Agricultural University, 2023, 46(2): 237−246.
[12] Cao Y, Bi M, Yang P, et al. Construction of yeast one-hybrid library and screening of transcription factors regulating LhMYBSPLATTER expression in Asiatic hybrid lilies ( Lilium spp. )[J]. BMC Plant Biology, 2021, 21: 563. doi: 10.1186/s12870-021-03347-1
[13] 费日雯, 樊富伟, 孙天祎, 等. 芍药种子休眠解除过程的酵母杂交cDNA文库构建[J]. 北京林业大学学报, 2023, 45(7): 120−129. doi: 10.12171/j.1000-1522.20210368 Fei R W, Fan F W, Sun T Y, et al. The construction of a yeast hybrid cDNA library during seed dormancy release of the Paeonia lactiflora[J]. Journal of Beijing Forestry University, 2023, 45(7): 120−129. doi: 10.12171/j.1000-1522.20210368
[14] Yang H, Zhou Y, Zhang Y, et al. Identification of transcription factors of nitrate reductase gene promoters and NRE2 cis-element through yeast one-hybrid screening in Nicotiana tabacum[J]. BMC Plant Biology, 2019, 19: 145. doi: 10.1186/s12870-019-1724-z
[15] Wang H, Wang Z, Lou X, et al. Construction of a membrane yeast two-hybrid library and its application in identification of PsARRO-1-interacting proteins in tree peony[J]. The Journal of Horticultural Science and Biotechnology, 2023, 98(1): 72−86. doi: 10.1080/14620316.2022.2092561
[16] Wellenreuther R, Schupp I, the German cDNA Consortium, et al. SMART amplification combined with cDNA size fractionation in order to obtain large full-length clones[J]. BMC Genomics, 2004, 5: 36. doi: 10.1186/1471-2164-5-36
[17] 张琨, 思彬彬, 周军, 等. 苹果砧木‘青砧1号’叶片cDNA文库构建及 MdMLO上游调控因子的筛选[J]. 园艺学报, 2023, 50(5): 933−946. Zhang K, Si B B, Zhou J, et al. Construction of cDNA library of apple rootstock ‘Qingzhen 1’ leaf and screen of MdMLO genes’ upstream regulator[J]. Acta Horticulturae Sinica, 2023, 50(5): 933−946.
[18] Tanaka Y, Sasaki N, Ohmiya A. Biosynthesis of plant pigments: anthocyanins, betalains and carotenoids[J]. The Plant Journal, 2008, 54: 733−749. doi: 10.1111/j.1365-313X.2008.03447.x
[19] Kellenberger R T, Glover B J. The evolution of flower colour[J]. Current Biology, 2023, 33(11): R484−R488. doi: 10.1016/j.cub.2023.01.055
[20] Sunil L, Shetty N P. Biosynthesis and regulation of anthocyanin pathway genes[J]. Applied Microbiology and Biotechnology, 2022, 106: 1783−1798. doi: 10.1007/s00253-022-11835-z
[21] Bürkle L, Meyer S, Dortay H, et al. In vitro recombination cloning of entire cDNA libraries in Arabidopsis thaliana and its application to the yeast two-hybrid system[J]. Functional and Integrative Genomics, 2005, 5: 175−183. doi: 10.1007/s10142-005-0134-5
[22] Ohara O, Temple G. Directional cDNA library construction assisted by the in vitro recombination reaction[J]. Nucleic Acids Research, 2001, 29(4): E22. doi: 10.1093/nar/29.4.e22
[23] Lu C F, Qu J P, Deng C Y, et al. The transcription factor complex CmAP3-CmPI-CmUIF1 modulates carotenoid metabolism by directly regulating the carotenogenic gene CmCCD4 a-2 in chrysanthemum[J]. Horticulture Research, 2022, 9: uhac020. doi: 10.1093/hr/uhac020
[24] Jiang S, Chen M, He N, et al. MdGSTF6, activated by MdMYB1, plays an essential role in anthocyanin accumulation in apple[J]. Horticulture Research, 2019, 6: 40. doi: 10.1038/s41438-019-0118-6
[25] Zhou L, Geng Z, Wang Y, et al. A novel transcription factor CmMYB012 inhibits flavone and anthocyanin biosynthesis in response to high temperatures in chrysanthemum[J]. Horticulture Research, 2021, 8: 248. doi: 10.1038/s41438-021-00675-z
[26] Chen K Q, Zhao X Y, An X H, et al. MdHIR proteins repress anthocyanin accumulation by interacting with the MdJAZ2 protein to inhibit its degradation in apples[J]. Scientific Reports, 2017, 7: 44484. doi: 10.1038/srep44484
[27] Luo D, Xiong C, Lin A, et al. SlBBX20 interacts with the COP9 signalosome subunit SlCSN5-2 to regulate anthocyanin biosynthesis by activating SlDFR expression in tomato[J]. Horticulture Research, 2021, 8: 163. doi: 10.1038/s41438-021-00595-y
[28] Xing S, Li R, Zhao H, et al. The transcription factor IbNAC29 positively regulates the carotenoid accumulation in sweet potato[J]. Horticulture Research, 2023, 10: uhad010. doi: 10.1093/hr/uhad010



 下载:
下载: