Phylogenetic relationships of the genus Rosa based on SSR markers and single copy nuclear gene
-
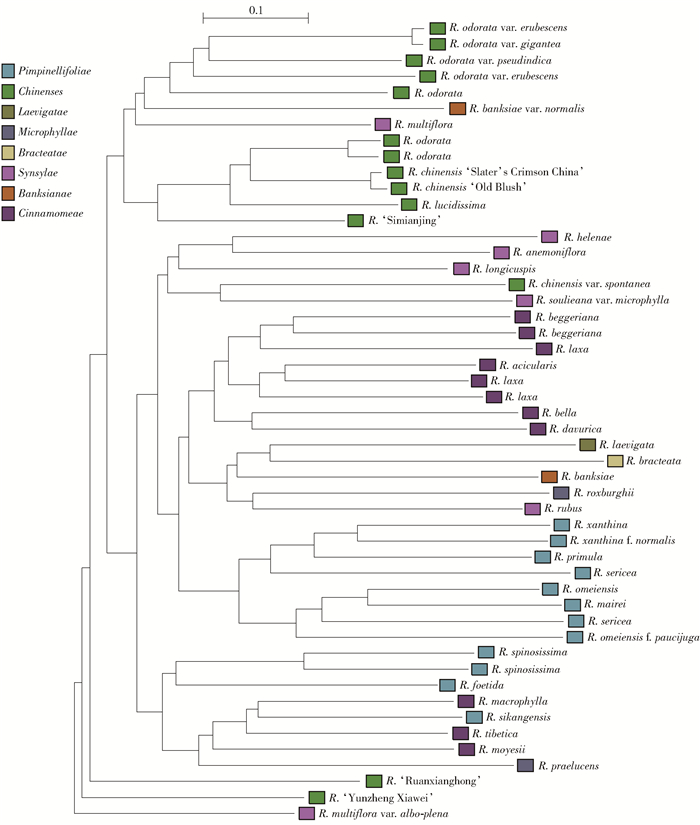
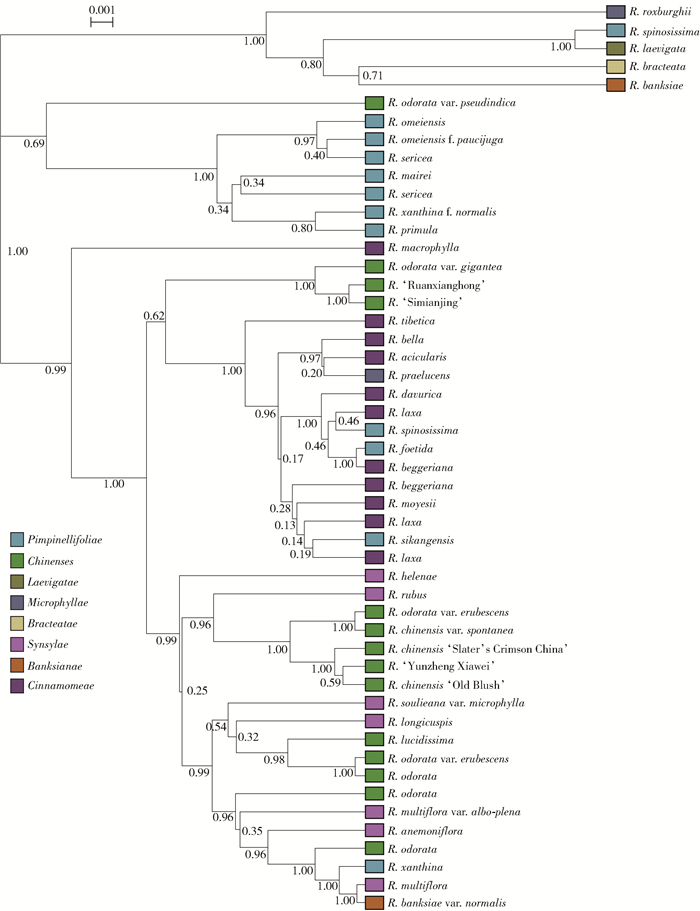
摘要:目的通过深入分析中国产野生蔷薇属植物的遗传背景,为其品种演化、系统分类提供分子学依据,也为种间杂交亲本的选择提供一定的指导,从而为进一步开发我国丰富的野生蔷薇属植物资源提供理论基础。方法本研究以50份蔷薇属植物样本、42个种或品种为研究对象,运用SSR标记及单拷贝核基因GAPDH对其遗传多样性进行分析。利用MAC-PR理论预测不同倍性蔷薇属植物的SSR基因型。结果在29个SSR位点上共计检测出382个等位基因变异,多态性信息含量介于0.413 9至0.934 0之间,平均值为0.798 9。计算Bruvo遗传距离并构建了邻接树,解决了SSR标记在不同倍性样本之间应用困难的问题。同时基于GAPDH基因序列片段构建了50个样本的贝叶斯树。基于SSR标记构建的系统发生树显示,50个样本聚成了6个分类群,月季组、桂味组样本聚类效果较好,而其他种类与现有分类系统差异较大。通过测序及克隆成功获得了所有样本的GAPDH基因序列片段,其中,比对后的序列长度为841 bp,变异位点数164个;基于GAPDH基因的聚类结果与现有的分类系统也有较大的差异。结论蔷薇属植物基于遗传关系的分类体系与现有的植物学分类系统有较大的差别。月季组、合柱组间遗传关系十分紧密;芹叶组、桂味组没有形成单系类群,这两组间可能存在着基因交流事件;小叶组中两个种没有很近的亲缘关系。Abstract:ObjectiveThis research provides some information for the controversy over some taxonomy and phylogenetic problems on Rosa species, and provides basis for the selection of parents of interspecific hybrids. This will provide a theoretical basis for further development of abundant wild rose resources in China.MethodIn this study, 50 samples with 42 species and varieties of genus Rosa were used to analyze genetic relationships and construct phylogenetic trees based on SSR markers and single copy nuclear gene (GAPDH). In order to predict the genotype of polyploidies, MAC-PR method was used.ResultA total of 382 alleles were detected at 29 loci. The polymorphism information content ranged from 0.413 9 to 0.934 0 with an average value of 0.798 9. Then we calculated the Bruvo distance and constructed NJ tree (Neighbour-joining tree). The problem to apply SSR markers in polyploidies was solved in the above-mentioned territory. The phylogenetic tree based on GAPDH fragment was also constructed by Bayesian inference. The phylogenetic tree showed that 50 accessions could be clustered into 6 taxa. The traditional classification system of Chinenses and Synstylae is mostly supported by molecular data while others not. GAPDH sequence fragments from all accessions were successfully obtained after cloning and sequencing. The sequence length was 841 bp with 164 variable loci. The phylogenetic relationship based on GAPDH did not correspond to traditional classification system.ConclusionThe result showed that the genus Rosa in its traditional infrageneric circumscription is not supported by molecular data. Sect. Chinenses and Sect. Synstylae are closed in genetic relationship. Sect. Pimpinellifoliae and Sect. Cinnamomeae are not monophyletic and a predicted gene flow exists between these sections. Two species in Sect. Microphyllae are not related based on molecular data.
-
Keywords:
- Rosa /
- SSR markers /
- single copy nuclear gene /
- genetic diversity
-
坡面薄层流是降雨在扣除截留、填洼、下渗等损失后沿坡面形成的浅层明流[1],是一种特殊而复杂的水流形态。研究表明,坡面薄层流不同于一般的明渠水流,水深一般只有几毫米甚至零点几毫米,薄层水流流向不稳定,沿程有质量源和动量源汇入,产生能量紊动,且受地表状况、雨强等诸多因素的影响,所以对坡面薄层水流的研究难度较大[2-4]。而坡面流是径流初始阶段和侵蚀演变的初始动力,其水动学特性对阐明土壤侵蚀和坡面产沙机理具有重要理论意义[5-8]。
目前,对于薄层水流特性的研究热点聚焦于坡面流流态及坡面流阻力特性,关于其流态归属问题,Horton等[9]认为坡面薄层水流是紊流中点缀层流的一种混合流区;Emmett[10]认为坡面流虽有紊流特性,但仍展现出较多层流性质,故定义为“扰动流”;Selby[11]认为坡面流是紊流和层流的混合流区。但仍然缺少公认的薄层流流态的界定方法。关于薄层水流阻力规律的研究,已有的研究成果多应用二元流雷诺数判别准则进行流区划分[4-5, 12]。关于降雨对坡面流阻力的影响,大部分的研究都表明,降雨对水流阻力的影响程度与水流流态有关,雨滴的打击作用和动能输入使其阻力增大,降雨在伪层流[4]情况下对水流阻力的影响最为显著,而有的学者认为降雨影响可以忽略[13]。而对于薄层水流特性的研究方法聚焦于人工模拟降雨或者水槽放水冲刷,两者共同作用下的水动力学特性研究十分少见。潘成忠等[13]通过上方来水和模拟降雨试验,研究了不同流量和坡度条件下坡面薄层水流水力学参数和滚波特性,初步探明了降雨和坡度对它们的影响。目前,对于坡面水流特性的探讨虽有不少研究成果,但由于坡度较小、床面光滑、缺少试验资料等因素,其结论的实际应用性也受到很大程度的限制,尤其在山地陡坡、雨量急、大的情况下。坡面流其底坡较天然明渠陡峭得多,重力作用更为突出。自然中,在山地陡坡、大雨条件下会发生超渗产流现象,坡面流冲刷和降雨同时作用于坡面,二者综合作用力对坡面的影响目前尚未明确。在国外,坡面流水动力学特性研究多侧重于缓坡,而国内多侧重于细沟水流,有研究表明,20°~ 25°是坡地土壤侵蚀的临界坡度[14]。目前针对陡坡和降雨对坡面流水流特性的研究尚显不足。与缓坡相比,陡坡条件下的水流动力特性和侵蚀特征具有其特殊性[15-16]。所以研究坡面流冲刷和降雨共同作用下的陡坡坡面流水动力学特性对防治水土流失有重要意义。
本文采用陡坡坡面定床阻力试验,通过人工模拟降雨和放水冲刷试验相结合的方法,定量研究5种流量和4个典型降雨强度(含无降雨)条件下,受4种不同粗糙度影响的水力要素关系及阻力的变化特征。研究陡坡降雨条件下的薄层水流水动力学特性对于揭示坡面薄层水流阻力的内在规律具有重要理论意义。
1. 研究区概况
缙云山位于三峡库区内,是国家级自然保护区,地理坐标为106°17′~106°24′ E、29°41′~29°52′ N,属于典型的亚热带季风湿润气候,植被资源丰富,森林覆盖率达96.6%。占地面积76 km2,海拔350.0~951.5 m,年平均降水量1 611.8 mm,最高年降水量1 683.8 mm。降雨主要发生在4—9月,降水量1 243.8 mm,占全年的77.2%。相对湿度年平均值为87%,年平均气温13.6 ℃。缙云山多雾,日照时数少,年平均雾日数高达89.8 d,年平均日照时数则低于1 293 h。缙云山地形平缓,土层深厚,土壤肥力高,以三叠纪须家河组厚层石英砂岩、灰质页岩和泥质页岩为木质风化而成的酸性黄壤土为主。试验研究林总面积约为33.5 hm2。保护区主要树种为四川大头茶(Gordonia acuminata)、杉木(Cunninghamia lanceolata)、马尾松(Pinus massoniana)、四川山矾(Symplocos setchuensis)、川杨桐(Adinandra bockiana)、广东山胡椒(Lindera kwangtungensis)、毛竹(Phyllostachys heterocycla)、细齿叶柃(Eurya nitida)等。
2. 试验方法
试验时间为2016年7—9月。具有固定的不透水下垫面和一定粗糙度的坡面称为定床阻力坡面,为了便于测量和控制浅层水流的边界条件,本试验采用坡面定床阻力试验,在模拟天然地表粗糙度的同时也消除了床面形态变化对水流紊动的影响[17-22]。试验对降雨和坡面流共同作用下的坡面薄层流水动力学特性进行研究。
试验水槽结构示意图如图 1所示,试验水槽结构尺度为长4.0 m,宽0.4 m,深0.1 m,在进水口管道安装精度为0.001 m3/h的流量计用于测量流量,每次试验开始前进行流量率定,保证在设计流量允许误差范围内。为模拟缙云山陡坡条件下的坡面流,坡度恒定设置为20°。为保证水流波动仅由水砂纸糙度引起,同时减小水槽边壁对水流的影响,故在水槽侧壁刷清漆,在水槽底部黏贴有机玻璃板,有机玻璃板与水槽侧壁的黏合使用玻璃胶,用刀片将边缘刮平,水砂布黏贴在有机玻璃板上。
通过黏贴水砂布设置4种不同下垫面,其中3种分别为40、60、80目水砂布床面,另外一种为光滑坡面。按照尼库拉兹提出的床面粗糙度(ks)表示方法,试验粗糙度(ks)分别为0.009(光滑坡面)、0.180、0.250、0.425 mm,分别对应缙云山不同土壤粒径的裸土表面。根据重庆缙云山的坡面径流小区监测的产流情况,同时也考虑到尽可能使水深取值范围较大,设计进口放水流量为0.486×10-3、0.694×10-3、1.042×10-3、1.389×10-3和1.736×10-3 m3/(s·m)共5个试验处理。根据重庆缙云山典型降雨强度,设计降雨强度分别为30、60和100 mm/h和无降雨。本试验采用侧喷式降雨机模拟天然降雨。天然降雨的主要特性包括降雨分布的均匀性、降雨强度、雨滴直径大小、雨滴的终点速度等。目前大多数科学试验都是以上述降雨特征作为人工模拟降雨的评价标准[23]。在试验区域内用烧杯收集降雨并采用体积法测量雨量,计算降雨均匀度在85%以上,一般要求0.8以上[24],部分能达到90%。实际降雨强度与设计降雨强度之差与设计降雨强度的比值为降雨强度误差,其值在5%以内。真实降雨雨滴直径通常为0.1~6.5 mm,本试验降雨强度为30~100 mm/h时,雨滴中数直径为1.32~2.05 mm[25]。天然降雨雨滴的终点速度为2.0~2.9 mm/s,研究表明降雨高度为7~8 m时,95%雨滴达到相应的终点速度[26];高度大于4.3 m时,大雨滴达到终点速度的80%[27];具有初速度的下喷式喷头,降雨高度达2 m时,不同直径的雨滴可以获得终点速度[25]。本装置采用喷嘴式喷头(具有初速度),有效降雨高度为6 m,可以满足2.0~2.9 mm/s的终点速度。降雨试验场次设计采用雨强、粗糙度与放水流量的完全组合试验并重复试验一次,共4×4×5×2=160场降雨。
沿水槽自上而下设纵向水深观测断面3个,分别距槽顶1.0、2.0和3.0 m,每个观测断面横向设3个间距等分观测点(图 1),即每个工况下测量9次水深,求平均值获得该工况下的平均水深。水深采用水位测针仪测定,精度为0.1 mm。断面表层流速采用KMnO4染色示踪法测定,选择水槽中部实验段3 m测量流速[28],在水流表面滴入染色剂并记录时间与试验段距离,以此反映坡面流的表层流速,6次重复,求平均值获得该工况下的平均表层流速。
水动力学参数计算公式如表 1。
表 1 水力参数计算Table 1. Calculation of hydraulic parameters公式序号
Formula order No.公式Formula 符号及其意义Symbol and its meaning (1) u=qh u为断面平均流速,m/s;q为单宽流量,m3/(m·s);h为实测断面平均水深,cm
u is mean velocity, m/s; q is unit discharge, m3/(m·s); h is measured flow depth, cm(2) α=uus α为流速修正系数;us为表层流速,m/s
α is velocity correction factor; us is velocity, m/s(3) u=ηq1−mJn m为流态指数; J为水流坡降,可近似取sin θ; θ为水槽坡度;η和n为拟合函数中的系数
m is flow-state indicator; J is hydraulic slope which closes to sin θ; θ is slope of flume; η and n are coefficient of the fitting function(4) Re=uRv Re为雷诺数;R为水力半径,m,薄层水流可视为二元流,水力半径近似等于断面平均水深h;υ为运动黏滞系数,cm2/s;υ=0.017 75/(1+0.033 7t+0.000 22t2),t为水温,℃
Re is Reynolds number; R is hydraulic radius, m, overland flow is regarded as binary stream so R closes to h; υ is coefficient of kinematic viscosity, cm2/s. υ=0.017 75/(1+0.033 7t+0.000 22t2). t is water temperature, ℃(5) Fr=u√gh Fr为弗劳德数;g为重力加速度Fr is froude number and g is gravitational acceleration (6) f=8gRJu2 f为阻力系数f is resistance coefficient 3. 结果与分析
3.1 平均流速及流速修正系数
由流量计测得的流量与水槽宽度之比为单宽流量,不同工况下坡面流的平均流速可以通过公式(1)u=qh计算得到,即单宽流量与实测断面平均水深之比,平均流速精度为0.01 m/s,误差为5%。图 2为不同降雨强度下平均流速随单宽流量的变化规律,由图可知降雨时,水流的平均流速随着单宽流量的增大呈幂函数增加趋势,随粗糙度的增加而减小。一般认为,由于雨滴击溅作用产生的附加阻力会使流速减小,而在本试验坡度条件下,降雨对平均流速有增加作用,但是不同降雨强度影响间差异不明显,这可能与陡坡条件下雨滴动量沿坡面分量较大有关[13]。粗糙度和降雨强度相同时,单宽流量增大一倍时平均流速增加68.7%,说明单宽流量对平均流速影响显著。在无降雨条件下,平均流速增幅较为稳定,水流的平均流速随单宽流量的增加呈幂函数增加,随粗糙度的增加而减小。无降雨时水流平均流速与单宽流量和与粗糙度的定性关系与以往结果一致[15, 19-20]。
在本试验给定的降雨和坡面流条件下,流量和粗糙度对平均流速影响显著,降雨主要起到扰动坡面流的作用,有增大坡面平均流速的趋势。
流速修正系数表示坡面流平均流速(u)与表层流速(us)的比值,坡面薄层流的水深较薄,难以直接观测水流垂线流速分布,只能通过探究流速修正系数的变化规律间接研究流速的垂线分布。图 3为不同降雨条件下流速修正系数随平均流速的变化规律,由图可知,无降雨时,流速系数随着平均流速的增加而增加,流速修正系数范围为0.04~0.37,数值偏小,且粗糙度间的流速系数差异较小。说明无降雨时坡面流的流速梯度较大,流速分布不均匀,粗糙坡面对底层流速的阻碍作用明显。降雨时,流速修正系数取值范围为0.42~0.98,随着粗糙度的增大而增大,随着平均流速的增加而增加。降雨条件下的流速修正系数数值相对较大且分布较为分散,最大值接近1。
表 2为收集和整理的以往试验数据,由表 2可知,降雨时流速修正系数数值偏大,最大值将近1。降雨对坡面流表层水流产生击溅作用,薄层水流内部产生扰动。当降雨强度增大时,薄层流内部扰动越来越大,水流上下层的流速差异越来越小,流速梯度越小。所以,降雨时流速修正系数整体比无降雨条件下的大。
表 2 收集数据及试验数据概况Table 2. Overview of literature datasets and experimental data资料来源
Source of date中值粒径
Median size
(d50)/mm坡度
Slope degree
(J)/(°)降雨强度
Rainfall intensity
P/(mm·h-1)单宽流量
Unit discharges×
10-3(q)/(m2·s-1)水深
Depth of water
(h)/mm雷诺数
Reynolds
number (Re)流速修正系数
Velocity correction
factor (α)文献[13] Literature [13] 1.5~15 0 0.08、0.25 0.27~1.37 320~998 0.40~0.70 文献[13] Literature [13] 1.5~15 30 0.08、0.25 0.31~1.48 409~1 097 0.41~0.67 文献[29] Literature [29] 0.4、0.67 1.2 0 0.01~0.06 0.84~1.33 26~102 0.56~0.61 文献[30] Literature [30] 0.74 3.5、5.5 0 0.13~1.46 2.37~5.79 295~3 188 0.33~0.86 本文This paper 0.009~0.425 20 0 0.49~1.74 0.7~5.9 142~842 0.04~0.37 本文This paper 0.009~0.425 20 30、60、100 0.49~1.74 2.0~6.0 514~1 862 0.42~0.98 从流速修正系数角度同样可以得到粗糙度对流速影响显著,降雨主要起到扰动坡面流的作用。
3.2 流态指数
张宽地等[31]提出流态指数概念,m被认为是与流态相关的指数,流态指数反应了单宽流量对坡面水流流速的影响程度,即水流耗能的主要形式。m值范围在0到1之间,m值越大,水流能留转化为动能较少,此时以阻力做功为主,反之,则以水流转化为动能为主。
由表 3可知,本实验条件下,流态多数在过渡流区,少数处于层流区,流态指数范围为0.291~0.538,平均值为0.418。由表 3和图 4可知,无降雨时,流态指数随着粗糙度的增加而明显减小,随着粗糙度的增大其减小程度分别为21%、28%和39%;中小雨强时,流态指数无明显规律,雨强为30 mm/h时,变化程度分别为18%、-29%和12%,雨强为60 mm/h时,变化程度分别为12%、-17%和-16%;大雨强时,流态指数呈现出较为明显下降的趋势,随着粗糙度的增大其变化程度分别为-6%、1%和-11%。降雨扰动造成了流态的复杂性。坡面水流流态指数m值的影响因素比较复杂[1],无降雨时,影响水流状态的主要因素为粗糙度,粗糙度较小时,水流水面失稳产生滚波,阻力作功耗能居主要地位,粗糙度较大时,坡面凹凸影响滚波发育,水流紊动强度较低,增加水流流速耗能居主要地位。中小雨强时,流态指数无明显规律,说明降雨是造成流态指数不稳定的关键因素(F=4.55>F0.05=3.86)(见表 4),降雨对水面的击溅作用扰动水面,增加水流的紊动强度。大雨强时,流态指数呈现出较为明显下降的趋势。说明随着粗糙度的增加,水流从阻力做功耗能居主要地位发展到能量转化动能居主要地位,侧面反应出流速梯度逐渐增大,可能是由于陡坡条件下雨强的击溅对流速有促进作用。
表 3 各实验组次流态指数m的实测值Table 3. Measured m value of different experimental groups粗糙度Surface
roughness/mm降雨强度Rainfall intensity/(mm·h-1) 0 30 60 100 0.009 0.477 0.412 0.398 0.532 0.180 0.378 0.486 0.446 0.501 0.425 0.343 0.291 0.331 0.537 0.380 0.290 0.463 0.334 0.473 表 4 各实验组次流态指数m方差分析Table 4. Variance analysis of m of different experimental groups变异来源Source of variation SS df MS F F0.05 F0.01 粗糙度间Among surface roughness 0.021 3 0.006 9 1.895 3.86 6.99 雨强间Among rainfall intensities 0.050 3 0.016 5 4.549* 3.86 6.99 误差Error 0.033 9 0.003 6 总变异Total variation 0.104 15 注:SS表示平方和;MS表示平均平方和;F表示平方和之比;F0.05和F0.01表示显著水平。Notes: SS represents sum of square; MS represents average sum of square; F is ratio of SS; F0.05 and F0.01 represent significant levels. 无降雨时,在试验粗糙度范围内,较大试验坡度的流态指数随着粗糙度的增加而明显减小,这与张宽地等[1]研究中的变化趋势相同,粗糙度继续增大时流态指数是否增加有待进一步研究。分析本试验的流态指数变化趋势,降雨引起水面失稳并产生滚波,其可能会影响水流流态。潘成忠等[13]认为降雨对断面滚波数具有增加效应,而对波高和波长影响不显著。在坡面薄层水流试验中,滚波可能是进一步的研究重点。
3.3 水流流型流态
水流流区是指坡面内的紊动水能,分为层流区、过渡区和紊流区,根据雷诺数Re判断,水流流型是指坡面流是缓流、临界流还是急流,根据弗劳德数Fr判断[22]。张宽地等[31]综合考虑雷诺数和弗劳德数对水流流态的判断标准,Re=500、Re=2 000和Fr=1将水流分为6区流态(图 5),分别是缓层流、缓过渡流、缓紊流、急层流、急过渡流和急紊流。将判断流态的雷诺数和判断流型的弗劳德数综合体现于一张图中,能够更为清晰地说明降雨、粗糙度和流量对坡面流状态的综合影响。
![]() 图 5 不同降雨条件下水流流态分区A.缓紊流区; B.缓过渡流区;C.急紊流区;D.缓层流区;E.急过渡流区;F.急层流区。Figure 5. Flow state zoning under different rainfall conditionsA, subcritical turbutent flow; B, subcritical transition flow; C, supercritical turbulent flow; D, subcritical laminar flow; E, supercritical transition flow; F, supercritical laminar flow.
图 5 不同降雨条件下水流流态分区A.缓紊流区; B.缓过渡流区;C.急紊流区;D.缓层流区;E.急过渡流区;F.急层流区。Figure 5. Flow state zoning under different rainfall conditionsA, subcritical turbutent flow; B, subcritical transition flow; C, supercritical turbulent flow; D, subcritical laminar flow; E, supercritical transition flow; F, supercritical laminar flow.陡坡时,在降雨和坡面流胁迫下,水流雷诺数为500~2 000,水流流态均属于层紊流过渡区,且多数属于急流区,少数属于缓流区。说明较大坡度增大了重力方向的分力,水流耗能主要以增加流速为主[13]。图 5a中,ks=0.425 mm时,雷诺数数值上是层流,然而实际中存在滚波现象,不符合“层层不混搀”的层流特性,因而属于“伪层流”[4]。模拟降雨条件下的薄层水流流态与流量密切相关,随着流量的增加,水流流态由层流区向过渡流区延伸[31]。
由图 5可知,粗糙度对坡面薄层流流态起重要作用。无雨时,坡面颗粒越大水流越趋近缓流,这与敬向锋等[22]得到的“床面越粗糙坡面流流态越倾向于向层流区延伸”结果一致。在水流运动过程中,遇到颗粒产生绕流,绕流过程中流速方向改变,流速大小减小,动能减少而阻力作功增加,粗糙程度越大,坡面对水流流动形成的阻力越大,流速减缓越显著。降雨时,粗糙度与流态不再具有相关关系。说明降雨对坡面流的击溅作用使薄层流产生扰动,所以水流流态均不处于层流区,多数处于急过渡流区,扰动程度相对削弱粗糙度对流态的影响。
与无降雨的坡面流相比,有降雨水流更趋向急流,说明降雨起到增加流速的作用。一般认为,由于雨滴击溅作用产生的附加阻力会使流速减小,张宽地等[1]通过试验认为在坡度较大(大于10.5%)条件下,基本上降雨均不同程度地增大表层流速,这与本试验结果一致,可能主要是因为陡坡条件下雨滴动量沿坡面的分量较大有关,能量更多地转化为动能。雨强越大,能够转化为动能的能量越多。
3.4 阻力特征
本试验在降雨条件下通过砂纸模拟下垫面,阻力规律只考虑颗粒阻力和降雨阻力的综合体现。
由图 6可知,坡面薄层流阻力系数与雷诺数呈负相关关系,随着雷诺数的增加阻力系数逐渐减小,且减小的幅度越来越小,最后趋于稳定。说明随着流量的增大,水流克服阻力所消耗的能量增加,而用于坡面侵蚀的能量减小。随着流量的增大,水深增加到一定程度后,坡面水砂纸处于完全淹没状态,由于水砂纸引起的坡面粗糙无法影响到主流区,该工况下阻力系数与雷诺数无关,而是趋近于一个常数。
降雨对坡面水流阻力系数的影响目前并无定论,吴普特等[32]认为降雨减少水流摩阻系数,但潘成忠等[13]认为降雨对坡面阻力系数无显著影响。本实验中与有降雨相比,无降雨时的阻力系数相对稳定,无明显波动。说明本实验条件下,降雨对坡面阻力系数的影响不显著,降雨主要起到扰动坡面流的作用。进一步地,采用逐步回归分析,定量研究糙度(ks)、单宽流量(q)和降雨强度(P) 3个影响因子对坡面流的阻力贡献率,计算结果见表 5。其中,降雨强度被排除,表明其对阻力系数无显著影响;由自变量系数可知,坡面流阻力系数与粗糙度呈正相关,与单宽流量呈负相关,这与上述的讨论相符合。
表 5 达西阻力系数影响因子的逐步回归分析Table 5. Stepwise regression of impact factors of resistance coefficient模型Model 相关变量Related variable 标准系数
Standardized coefficient自变量系数
Independent variable coefficient标准误差Standard error 常量Constant value 3.022 0.611 粗糙度Surface roughness 10.772 1.454 0.533 单宽流量Unit discharge -3 122.434 488.542 -0.460 已有研究成果表明,当颗粒阻力起主要作用时,阻力系数与雷诺数的幂函数关系 f=aRbe 才成立[10],为进一步研究降雨强度对坡面流阻力的影响,将本试验模拟降雨条件下裸坡薄层流数据进行拟合,得到阻力计算公式。
f′=2108.19R−1.227eP0.257,R2=0.3977 (7) 将降雨强度的因式去除,拟合得到阻力计算公式。
f′=5938.16R−1.227eP0.257,R2=0.3976 (8) 式中:f′为阻力系数;P为降雨强度(mm/h)。
对比式(7)和式(8),剔除雨强后,相关系数下降0.01%,说明降雨对坡面阻力系数的影响不显著,降雨主要起到扰动坡面流的作用。王俊杰在模拟降雨条件下得到相关系数下降了7.76%,雨强对于阻力系数的影响不能忽视[21]。这可能是由于下垫面因素不同,与降雨强度大小可能也有关系,降雨对坡面流的作用有待进一步研究。
4. 结论
本实验通过开展陡坡不透水下垫面条件下的模拟人工降雨和水槽放水冲刷试验,基于流态指数和紊动能量耗散规律,定性分析了降雨和坡面流共同作用下的坡面薄层流水动力学特性,得到以下结论。
1) 水流的平均流速随着单宽流量的增大呈幂函数增加,随粗糙度的增加而减小。粗糙度和单宽流量相同时,降雨强度增大一倍引起平均流速的变化程度为15.6%。粗糙度和降雨强度相同时,单宽流量增大一倍引起平均流速的变化程度为68.7%。无降雨时,流速系数随着平均流速的增加而增加,流速系数范围为0.04~0.37,粗糙度间的流速系数差异较小。降雨时,流速系数取值范围为0.42~0.98,随着粗糙度的增大而增大,随着平均流速的增加而增加。降雨条件下的流速修正系数数值相对较大且分布较为分散,最大值接近1。试验降雨下对坡面流起到扰动作用,有增大坡面平均流速的趋势。
2) 流态指数范围为0.291~0.538,无降雨时,流态指数随着粗糙度的增加而明显减小,其减小程度分别为21%、28%和39%;中小雨强时,流态指数无明显规律,雨强为30 mm/h时,变化程度分别为18%、-29%和12%,雨强为60 mm/h时,变化程度分别为12%、-17%和-16%;大雨强时,流态指数呈现出较为明显下降的趋势,其变化程度分别为-6%、1%和-11%。粗糙度继续增大时流态指数是否增加有待进一步研究。降雨引起水面失稳并产生滚波会影响水流流态,所以进一步的坡面薄层水流试验中滚波研究是不可忽略的一部分。
3) 水流雷诺数为500~2 000,所有实验工况下水流流型均属于层紊流过渡区;水流流态整体趋于急流状态;无雨时,粗糙度与流态关系明显,其值越小水流越趋近急流,而降雨时,由于降雨的扰动作用二者不再具有相关关系。
4) 定量研究糙度(ks)、单宽流量(q)和降雨强度(P)3个影响因子对坡面流的阻力贡献率,表明降雨对阻力系数无显著影响,坡面流阻力系数与粗糙度呈正相关关系,与单宽流量呈负相关关系,裸坡条件下考虑雨强影响的坡面流阻力计算公式与剔除雨强的公式相比,相关系数下降0.01%,说明降雨对阻力系数无显著影响,主要起到扰动坡面流的作用。另有研究表明模拟降雨条件下的相关系数下降了7.76%,雨强对于阻力系数的影响不能忽视。所以降雨对坡面流的作用有待进一步研究。
坡面薄层流是坡面径流的初始阶段和侵蚀演变的初始动力,本试验为深入研究降雨和坡面流共同作用下的坡面薄层流水动力学特性提供科学依据,对土壤侵蚀预报模型、水土流失治理方法、泥沙灾害及环境工程等问题均有重要的科学及实践意义。
-
表 1 50个样本名录
Table 1 List of samples
编号No. 种/品种名Specie/variety 组Section 倍性Ploidy level 1 月月红R. chinensis ‘Slater’s Crimson China’ 月季组Sect. Chinenses 2[1] 2 月月粉R. chinensis ‘Old Blush’ 月季组Sect. Chinenses 2[1] 3 单瓣月季花R. chinensis var. spontanea 月季组Sect. Chinenses 2[1] 4 ‘云蒸霞蔚’R. ‘Yunzheng Xiawei’ 月季组Sect. Chinenses 4[23] 5 香水月季R. odorata 月季组Sect. Chinenses 2[24] 6 粉红香水月季R. odorata var. erubescens 月季组Sect. Chinenses 未知Unkonwn 7 粉红香水R. odorata var. erubescens 月季组Sect. Chinenses 未知Unkonwn 8 香水月季R. odorata 月季组Sect. Chinenses 2[24] 9 ‘软香红’R.‘Ruanxianghong’ 月季组Sect. Chinenses 4[24] 10 ‘四面镜’R.‘Simianjing’ 月季组Sect. Chinenses 3[24] 11 桔黄香水月季R. odorata var. pseudindica 月季组Sect. Chinenses 2[24] 12 大花香水月季R. odorata var. gigantea 月季组Sect. Chinenses 2[24] 13 香水月季R. odorata 月季组Sect. Chinenses 2[24] 14 亮叶月季R. lucidissima 月季组Sect. Chinenses 未知Unkonwn 15 弯刺蔷薇R. beggeriana 桂味组Sect. Cinnamomeae 2[1] 16 弯刺蔷薇R. beggeriana 桂味组Sect. Cinnamomeae 2[1] 17 刺蔷薇R. acicularis 桂味组Sect. Cinnamomeae 4, 8[1] 18 疏花蔷薇R. laxa 桂味组Sect. Cinnamomeae 2[1] 19 疏花蔷薇R. laxa 桂味组Sect. Cinnamomeae 2[1] 20 疏花蔷薇R. laxa 桂味组Sect. Cinnamomeae 2[1] 21 美蔷薇R. bella 桂味组Sect. Cinnamomeae 4[11] 22 山刺玫R. davurica 桂味组Sect. Cinnamomeae 2, 4, 6[11] 23 华西蔷薇R. moyesii 桂味组Sect. Cinnamomeae 未知Unkonwn 24 大叶蔷薇R. macrophylla 桂味组Sect. Cinnamomeae 2[1] 25 西藏蔷薇R. tibetica 桂味组Sect. Cinnamomeae 未知Unkonwn 26 密刺蔷薇R. spinosissima 芹叶组Sect. Pimpinellifoliae 4[11] 27 黄刺玫R. xanthina 芹叶组Sect. Pimpinellifoliae 2[1] 28 异味蔷薇R. foetida 芹叶组Sect. Pimpinellifoliae 4[11] 29 绢毛蔷薇R. sericea 芹叶组Sect. Pimpinellifoliae 2[1] 30 单瓣黄刺玫R. xanthina f. normalis 芹叶组Sect. Pimpinellifoliae 2, 4[1] 31 报春刺玫R. primula 芹叶组Sect. Pimpinellifoliae 2[24] 32 密刺蔷薇R. spinosissima 芹叶组Sect. Pimpinellifoliae 4[11] 33 峨眉蔷薇R. omeiensis 芹叶组Sect. Pimpinellifoliae 2[11] 34 毛叶蔷薇R. mairei 芹叶组Sect. Pimpinellifoliae 未知Unkonwn 35 少对峨眉蔷薇R. omeiensis f. paucijuga 芹叶组Sect. Pimpinellifoliae 未知Unkonwn 36 绢毛蔷薇R. sericea 芹叶组Sect. Pimpinellifoliae 2[1] 37 川西蔷薇R. sikangensis 芹叶组Sect. Pimpinellifoliae 未知Unkonwn 38 白玉堂R. multiflora var. albo-plena 合柱组Sect. Synstylae 2, 3[1] 39 野蔷薇R. multiflora 合柱组Sect. Synstylae 2, 3[1] 40 长尖叶蔷薇R. longicuspis 合柱组Sect. Synstylae 2[11] 41 小叶川滇蔷薇R. soulieana var. microphylla 合柱组Sect. Synstylae 2[11] 42 卵果蔷薇R. helenae 合柱组Sect. Synstylae 2[11] 43 悬钩子蔷薇R. rubus 合柱组Sect. Synstylae 2, 3[11] 44 银粉蔷薇R. anemoniflora 合柱组Sect. Synstylae 4[24] 45 金樱子R. laevigata 金缨子组Sect. Laevigatae 2[11] 46 木香花R. banksiae 木香组Sect. Banksianae 2, 4[1] 47 单瓣木香花R. banksiae var. normalis 木香组Sect. Banksianae 2[1] 48 硕苞蔷薇R. bracteata 硕苞组Sect. Bracteatae 2[24] 49 缫丝花R. roxburghii 小叶组Sect. Microphyllae 2[11] 50 中甸刺玫R. praelucens 小叶组Sect. Microphyllae 10[11] 表 2 SSR引物信息
Table 2 Information of SSR primers
SSR编号SSR No. 基序Motif 连锁群[23] Linkage group 退火温度Annealing temperature/℃ 目标片段大小Expected size/bp 上游引物Forward primer (5′-3′) 下游引物Reverse primer (5′-3′) CL2996 (CCG)17 2 55.0 183 GCCACCATAGCCAGAGACAT AGAAGAAGTTGACGACAGGGAC H22E04 (AAG)7 6 55.0 241 GACATCACCACCACCACAAG AACCAAGGTTTCCAGTTCCA H23017 (CT)11 1 55.0 218 ACACCAAGCAAACCAAAACC AGCACGAAAACCGAGAGAGA Rw22A3 (TTC)6 6 52.9 150 AGAGAATTGAAAAGGGCAAG GAGCAAGCAAGACACTGTAA 327 (CTT)6 59.8 277, 232, 202 ACTCCTCCAAAGCTTCACCA CCTCATCGACAGAGTCGTCA 336 (TC)9 4 59.9 171, 185, 213 CAAACGAAACCCTCTGCTTC GACGATGCATTTGGTGTGAC 353 (TC)7 5 59.7 212, 107, 223 CGCCCTAGTCTCCTCTCTCTC CTCAAGCTGAAGCTCGGAGT 373 (CT)10 6 58.9 100, 106, 233 ACAAACTTCGCGATTCCTCT AGTTCCAGACGTTGGAGTGC 387 (CT)9 2 58.7 202, 229, 223 GCACTCTTGACGTTGTCCAT GTCAATGTAGTCCGGTTCGG 397 (CT)14 4 59.9 222, 221, 252 GGCCTAGCAAAGCAACAAAC AGTGGAGGGCAGTCTCTGAA 405 (ATG)5 7 59.9 265, 183, 237 CAGCGAAAAGAACAAGGACC CAGAAGCTAATAAATTAACAATCACCA 464 (TCGGA)3 59.5 134, 250, 149 TCTTTCGGTTCAGAAAGTTCG CTCGCTGATCTTGTCCATCA 467 (CGA)5 2 60.0 173, 174, 161 GTACGCTCTCTGGTCTTCGG CCCATGTCTCTGGCTATGGT 490 (TCT)6 60.0 128, 125, 272 ACAACCAACCCAAGAACTCG TCCCAGCTTCAGTCTCACCT 509 (CAC)5 7 60.0 208, 252, 261 CAACTGGGTTGGGTCAGTCT TCAAATGTACCTTGCGCTTG 510 (AAG)5 2 60.1 190, 157, 156 AGAGGTTTAGGGCAGCCATT GCGAATGATGGTGGAGAGTT 521 (GA)8 6 60.5 225, 224, 229 GTTCCAGCAGCACTCCAAGT AGAGGGGATTAGCTGCACTG 541 (AG)7 6 59.7 241, 243, 242 CTACTCCAATGTCCGCTTCC GTTGGAGAAGAAGCCGTGAG 593 (GA)7 60.1 136, 134, 119 TAACCAGGTCCTCACGAAGG AACAAATCCCCCAGGATAGG 596 (AGG)5 5 60.6 211, 212, 222 CGAGGAAAAACCCAAAATCC TGGAAGCAAGAAAAGGCAGT 625 (TC)8 7 59.0 143, 213, 142 CGCGTCTCTCACATCTCAAA AAGATCTTCTCTCCGGCCTT 629 (CTT)7 6 59.7 270, 273, 223 CACGAGCTCTCTCTCCCCTA TTGGTCTGTGAAGTGGTGGA 632 (GCCACC)3 60.1 153, 152, 154 AACTCATGGGTTCGTTGAGC GGTTGCGGAGAGAGAACAAG 637 (TTGATT)3 7 60.1 280, 279, 280 GCCGTAATTCGTGGAAAGAA ATGCCACCAGAACCTTGAAC 648 (CT)8 6 60.5 167, 169, 217 CCTAAAGCTTAAGCCCCCAA GCAATAGACTTGGCAGCCTC 651 (CAG)5 6 60.2 166, 167, 165 TCTGAGCACGACTCAACAGG AGGCATGTAATGCTGTGGGT 682 (TC)10 3 59.7 205, 131, 207 TTCTTGAGCTAAAAGTGCATCG CAGATCCAAACCGAACCCTA 686 (GAA)8 6 59.9 139, 150, 190 CACGAGTGTCACTGTTGCCT AGAATTGGCTTAGCTTGGCA 695 (TA)8 3 59.8 248, 244, 264 AGAAAAGCGAAAGCACAAGC CTTAAATGCGCCACCAATTT 表 3 部分样本的倍性预测
Table 3 Ploidy levels of the unknown samples
编号No. 种/品种名Specie/variety 倍性Ploidy level 7 粉红香水月季R. odorata var. erubescens 4 14 亮叶月季R. lucidissima 2 23 华西蔷薇R. moyesii 4 25 西藏蔷薇R. tibetica 6 34 毛叶蔷薇R. mairei 4 35 少对峨眉蔷薇R. omeiensis f. paucijuga 4 37 川西蔷薇R. sikangensis 4 表 4 SSR位点变异
Table 4 Variation of the 29 SSR lici in 97 rose samples
位点名称Locus name 等位基因数量Number of alleles 有效等位基因数[30] Effective number of alleles 多态信息含量Polymorphism information content RW22A3 14 6.50 0.846 2 H23017 22 11.40 0.912 4 H22E04 8 4.78 0.790 9 CL2996 9 5.32 0.812 1 695 19 7.85 0.872 7 686 10 6.60 0.848 5 682 17 8.53 0.882 9 651 9 2.71 0.630 9 648 13 7.57 0.868 1 637 6 2.19 0.542 7 632 4 2.38 0.580 0 629 13 5.77 0.826 7 625 21 13.68 0.927 0 596 7 3.78 0.735 8 593 20 14.69 0.932 0 541 16 9.40 0.893 7 521 16 9.71 0.897 2 510 6 1.71 0.413 9 509 8 3.81 0.737 5 490 12 7.07 0.858 7 467 4 2.38 0.579 2 464 5 2.10 0.524 5 405 17 9.81 0.898 2 397 22 14.28 0.930 1 387 16 8.94 0.888 2 373 16 10.02 0.900 3 353 17 6.86 0.854 4 336 23 15.13 0.934 0 327 12 6.62 0.849 1 总计Total 382 平均Mean 13.20 7.30 0.798 9 表 5 不同组的SSR位点信息
Table 5 Variation of the SSR loci in 6 sections
组Section 样本容量Sample size 等位基因数量Number of alleles 有效等位基因数Effective number of alleles 多态信息含量Polymorphism information content 月季组Sect. Chinenses 14 5.97 3.96 0.677 2 芹叶组Sect. Pimpinellifoliae 12 8.38 5.82 0.740 3 桂味组Sect. Cinnamomeae 11 7.17 5.79 0.691 6 合柱组Sect. Synstylae 7 6.48 6.09 0.753 2 金缨子组Sect. Laevigatae 1 1.38 1.07 0.344 8 硕苞组Sect. Bracteatae 1 1.28 1.07 0.241 4 小叶组Sect. Microphyllae 2 3.45 3.8 0.685 2 木季组Sect. Banksianae 2 2.41 2.61 0.540 0 平均Mean 6 4.57 3.78 0.584 2 -
[1] Ku C Z, Robertson K R. Flora of China[M]. Beijing: Science Press, 2003.
[2] Rehder A. Bibliography of cultivated trees and shrubs hardy in the cooler temperate regions of the northern hemisphere[M]. Boston: Arnold Arboretum of Harvard Univ, 1949.
[3] Roberts A V. Encyclopedia of rose science[M]. San Diego: Academic Press, 2003.
[4] 俞德浚, 谷粹芝.中国植物志:第36卷[M].北京:科学出版社, 1985. Yu D J, Gu C Z. Flora reipublicae popularis sinicae:Vol.36[M]. Beijing: Science Press, 1985.
[5] 白锦荣, 张启翔, 潘会堂.云南滇西北地区蔷薇属(Rosa L.)植物资源调查与评价[J].植物遗传资源学报, 2009, 10(2): 218-223. http://d.old.wanfangdata.com.cn/Periodical/zwyczyxb200902009 Bai J R, Zhang Q X, Pan H T. Investigation on germplasm resources of the genus Rosa L. in northwest Yunnan[J]. Journal of Plant Genetic Resources, 2009, 10(2):218-223. http://d.old.wanfangdata.com.cn/Periodical/zwyczyxb200902009
[6] Yu C, Luo L, Pan H T, et al. Karyotype analysis of wild Rosa species in Xinjiang, Northwestern China[J]. Journal of the American Society for Horticultural Science, 2014, 139(1): 39-47. doi: 10.21273/JASHS.139.1.39
[7] Tanaka N, Uchiyama H, Matoba H, et al. Karyological analysis of the genus Canna (Cannaceae)[J]. Plant Systematics and Evolution, 2009, 280(1-2): 45-51. doi: 10.1007/s00606-009-0165-9
[8] Song Z Q, Wang J H, Xie Y L. Karyological studies of Salvia miltiorrhiza in China[J]. Caryologia, 2010, 63(3): 269-277. doi: 10.1080/00087114.2010.10589737
[9] Zhu Z M, Gao X F, Fougère-Danezan M. Phylogeny of Rosa sections Chinenses and Synstylae (Rosaceae) based on chloroplast and nuclear markers[J]. Molecular Phylogenetics and Evolution, 2015, 87: 50-64. doi: 10.1016/j.ympev.2015.03.014
[10] Fougère-Danezan M, Joly S, Bruneau A, et al. Phylogeny and biogeography of wild roses with specific attention to polyploids[J]. Annals of Botany, 2015, 115(2): 275-291. doi: 10.1093/aob/mcu245
[11] Bruneau A, Starr J R, Joly S. Phylogenetic relationships in the genus Rosa: new evidence from chloroplast DNA sequences and an appraisal of current knowledge[J]. Systematic Botany, 2007, 32(2): 366-378. doi: 10.1600/036364407781179653
[12] Wissemann V, Ritz C M. The genus Rosa (Rosoideae, Rosaceae) revisited: molecular analysis of nrITS-1 and atpB-rbcL intergenic spacer (IGS) versus conventional taxonomy[J]. Botanical Journal of the Linnean Society, 2005, 147(3): 275-290. doi: 10.1111/j.1095-8339.2005.00368.x
[13] Wissemann V, Ritz C M. Evolutionary patterns and processes in the genus Rosa (Rosaceae) and their implications for host-parasite co-evolution[J]. Plant Systematics and Evolution, 2007, 266(1-2): 79-89. doi: 10.1007/s00606-007-0542-1
[14] Meng J, Fougère-Danezan M, Zhang L B, et al. Untangling the hybrid origin of the Chinese tea roses: evidence from DNA sequences of single-copy nuclear and chloroplast genes[J]. Plant Systematics and Evolution, 2011, 297(3-4): 157-170. doi: 10.1007/s00606-011-0504-5
[15] Varshney R K, Graner A, Sorrells M E. Genic microsatellite markers in plants: features and applications[J]. Trends in Biotechnology, 2005, 23(1): 48-55. http://d.old.wanfangdata.com.cn/OAPaper/oai_doaj-articles_65d8a77851fbed58b25cc5f7530ab914
[16] Bruvo R, Michiels N K, D'souza T G, et al. A simple method for the calculation of microsatellite genotype distances irrespective of ploidy level[J]. Molecular Ecology, 2004, 13(7): 2101-2106. doi: 10.1111/mec.2004.13.issue-7
[17] Esselink G D, Nybom H, Vosman B. Assignment of allelic configuration in polyploids using the MAC-PR (microsatellite DNA allele counting-peak ratios) method[J]. Theoretical and Applied Genetics, 2004, 109(2): 402-408. doi: 10.1007/s00122-004-1645-5
[18] Clark L V, Jasieniuk M. POLYSAT: an R package for polyploid microsatellite analysis[J]. Molecular Ecology Resources, 2011, 11(3): 562-566. doi: 10.1111/men.2011.11.issue-3
[19] Bisognin C, Seemüller E, Citterio S, et al. Use of SSR markers to assess sexual vs. apomictic origin and ploidy level of breeding progeny derived from crosses of apple proliferation-resistant Malus sieboldii and its hybrids with Malus × domestica cultivars[J]. Plant Breeding, 2009, 128(5): 507-513. doi: 10.1111/pbr.2009.128.issue-5
[20] Robertson A, Rich T C G, Allen A M, et al. Hybridization and polyploidy as drivers of continuing evolution and speciation in Sorbus[J]. Molecular Ecology, 2010, 19(8): 1675-1690. doi: 10.1111/mec.2010.19.issue-8
[21] Joly S, Starr J R, Lewis W H, et al. Polyploid and hybrid evolution in roses east of the Rocky Mountains[J]. American Journal of Botany, 2006, 93(3): 412-425. doi: 10.3732/ajb.93.3.412
[22] 于超.四倍体月季遗传连锁图谱的构建及部分观赏性状的QTLs分析[D].北京: 北京林业大学, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10022-1015319545.htm Yu C. Construction of a genetic linkage map and QTLs analysis for phenotypic traits in tetraploid roses[D]. Beijing: Beijing Forestry University, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10022-1015319545.htm
[23] Mathilde L, Alix P, Li S, et al. Nineteenth century French rose (Rosa sp.) germplasm shows a shift over time from a European to an Asian genetic background[J]. Journal of Experimental Botany, 2016, 67(15): 4711-4725. doi: 10.1093/jxb/erw269
[24] Hibrand-Saint Oyant L, Crespel L, Rajapakse S, et al. Genetic linkage maps of rose constructed with new microsatellite markers and locating QTL controlling flowering traits[J]. Tree Genetics & Genomes, 2008, 4(1): 11-23. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=008d914b0415cd187048d300eb94bf8a
[25] Boykin L M, Kubatko L S, Lowrey T K. Comparison of methods for rooting phylogenetic trees: a case study using Orcuttieae (Poaceae: Chloridoideae)[J]. Molecular Phylogenetics & Evolution, 2010, 54(3): 687-700. http://med.wanfangdata.com.cn/Paper/Detail/PeriodicalPaper_JJ0216377053
[26] Katoh K, Rozewicki J, Yamada K D. MAFFT online service: multiple sequence alignment, interactive sequence choice and visualization[J]. Briefings in Bioinformatics, 2017, 18(4):1-7. http://cn.bing.com/academic/profile?id=bab223f59b0e19bfe72ce124a5fa88f4&encoded=0&v=paper_preview&mkt=zh-cn
[27] Xia X H. DAMBE6: new tools for microbial genomics, phylogenetics, and molecular evolution[J]. Journal of Heredity, 2017, 108(4): 431-437. doi: 10.1093/jhered/esx033
[28] Darriba D, Taboada G L, Doallo R, et al. jModelTest 2: more models, new heuristics and parallel computing[J]. Nature Methods, 2012, 9(8): 772. http://d.old.wanfangdata.com.cn/NSTLQK/NSTL_QKJJ0227995424/
[29] Bouckaert R, Heled J, Kühnert D, et al. BEAST 2: a software platform for Bayesian evolutionary analysis[J/OL]. PLoS Computational Biology, 2014, 10(4): e1003537[2018-09-21]. https://doi.org/10.1371/journal.pcbi.1003537.
[30] Nielsen R, Tarpy D R, Reeve H K. Estimating effective paternity number in social insects and the effective number of alleles in a population[J]. Molecular Ecology, 2003, 12(11): 3157-3164. doi: 10.1046/j.1365-294X.2003.01994.x
[31] Esselink G D, Smulders M J, Vosman B. Identification of cut rose (Rosa hybrida) and rootstock varieties using robust sequence tagged microsatellite site markers[J]. Theoretical and Applied Genetics, 2003, 106(2): 277-286. doi: 10.1007/s00122-002-1122-y
[32] Hibrand-Saint Oyant L, Ruttink T, Hamama L, et al. A high-quality genome sequence of Rosa chinensis to elucidate ornamental traits[J/OL]. Nature Plants, 2018[2018-09-29]. https://doi.org/10.1038/s41477-018-0166-1.
[33] Matsumoto S, Wakita H, Fukui H. Molecular classification of wild roses using organelle DNA probes[J]. Scientia Horticulturae, 1997, 68(1-4): 191-196. doi: 10.1016/S0304-4238(96)00970-3
[34] Rowley G. The Scotch rose and its garden descendants[J]. Journal of the Royal Horticultural Society of London, 1961, 86: 433-437. http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=10.1177/030639687101300201
[35] Roberts A V. Relationship between species in the genus Rosa, section Pimpinellifoliae[J]. Botanical Journal of the Linnean Society, 1977, 74(4): 309-328. doi: 10.1111/boj.1977.74.issue-4
[36] Jian H Y, Zhang H, Tang K X, et al. Decaploidy in Rosa praelucens Byhouwer (Rosaceae) endemic to Zhongdian Plateau, Yunnan, China[J]. Caryologia, 2010, 63(2): 162-167. doi: 10.1080/00087114.2010.10589722
[37] Cronn R, Wendel J F. Cryptic trysts, genomic mergers, and plant speciation[J]. New Phytologist, 2004, 161(1): 133-142. http://d.old.wanfangdata.com.cn/NSTLQK/NSTL_QKJJ0213550427/
[38] Joly S, Mclenachan P A, Lockhart P J. A statistical approach for distinguishing hybridization and incomplete lineage sorting[J]. American Naturalist, 2009, 174(2): E54-E70. doi: 10.1086/600082
[39] Linder C R, Rieseberg L H. Reconstructing patterns of reticulate evolution in plants[J]. American Journal of Botany, 2004, 91(10): 1700-1708. doi: 10.3732/ajb.91.10.1700
[40] Fisher M C, Koenig G, White T J, et al. A test for concordance between the multilocus genealogies of genes and microsatellites in the pathogenic fungus Coccidioides immitis[J]. Molecular Biology & Evolution, 2000, 17(8): 1164-1174. http://cn.bing.com/academic/profile?id=64f55e344d62f5b483b0445c429481c4&encoded=0&v=paper_preview&mkt=zh-cn
[41] Scariot V, Akkak A, Botta R. Characterization and genetic relationships of wild species and old garden roses based on microsatellite analysis[J]. Journal of the American Society for Horticultural Science, 2006, 131(1): 66-73. doi: 10.21273/JASHS.131.1.66
[42] Nauta M J, Weissing F J. Constraints on allele size at microsatellite loci: implications for genetic differentiation[J]. Genetics, 1996, 143(2):1021-1032. http://d.old.wanfangdata.com.cn/OAPaper/oai_pubmedcentral.nih.gov_1207320
[43] Takezaki N, Nei M. Geneticdistances and reconstruction of phylogenetic trees from microsatellite DNA[J]. Genetics, 1996, 144(1): 389-399. http://med.wanfangdata.com.cn/Paper/Detail/PeriodicalPaper_PM8878702
-
期刊类型引用(3)
1. 李辉,林沂,孟祥爽,史振伟,蔡万园. 基于地基激光雷达的栾树分形特征分析. 山东农业大学学报(自然科学版). 2022(03): 475-483 .  百度学术
百度学术
2. 何东健,熊虹婷,芦忠忠,刘建敏. 基于多视角立体视觉的拔节期玉米水分胁迫预测模型. 农业机械学报. 2020(06): 248-257 .  百度学术
百度学术
3. 郭彩玲,刘刚. 基于颜色取样的苹果树枝干点云数据提取方法. 农业机械学报. 2019(10): 189-196 .  百度学术
百度学术
其他类型引用(7)




 下载:
下载: