Development and identification of SSR molecular markers based on whole genomic sequences of Punica granatum
-
摘要:目的利用cDNA文库筛选的石榴SSR多态性标记数量有限,为进一步推动石榴遗传多样性分析、遗传图谱构建、品种鉴定等研究,有必要系统开发高效稳定的分子标记位点。方法本研究根据已发表的石榴基因组测序数据利用MISA软件对1 ~ 6核苷酸重复的SSR位点进行了查找,分析了不同类型SSR位点的序列特征,进而设计引物并检测了引物的有效性和多态性。结果(1)石榴基因组中共检测到146 445个SSR位点,其中以单核苷酸重复型SSR最多(占51.95%),六核苷酸重复型最少(仅占0.38%);SSR序列以A/T碱基占主导,具有偏向性。(2)石榴基因组SSR序列长度变化范围为10 ~ 252 bp,平均长度15.48 bp。不同长度重复单元类型的SSR序列长度存在丰富变异,呈现随着重复次数增多,SSR序列丰度减少的趋势。(3)根据不同类型SSR位点设计并合成引物140对,其中119对在12份石榴种质中可扩增出有效条带,41对可产生多态性条带,PIC值在0.007 ~ 0.566之间;从中筛选出多态性较高、稳定性好的引物15对,共检测到等位基因44个,平均每个SSR位点检测到2.933 3个等位基因。结论利用石榴全基因组序列可实现SSR标记的大规模开发,并可鉴定出大量适用于石榴遗传多样性分析、遗传图谱构建、品种鉴定等研究的SSR引物。相关研究为石榴的遗传育种研究提供了丰富的SSR序列信息和标记资源。Abstract:Objective The number of polymorphic SSR markers developed through cDNA library is limited. In order to promote studies on genetic diversity, genetic mapping and variety identification, it is necessary to develop molecular marker loci.Method In this study, SSRs with 1−6 nucleotide repeats were searched from the published whole genome sequences of Punica granatum using MISA and the characterization of SSR sequences and their polymorphisms were analyzed.Result (1) A total of 146 445 SSR loci were detected in whole genome sequences of Punica granatum. Then content of mono-nucleotide repeat SSRs was the highest, accounting for 51.95%; in contrast, the content of hexa-nucleotide repeat SSRs was the lowest, only 0.38%. In all SSR sequences, most of bases were A/T, showing a bias in SSR sequences. (2) The range of SSR length ranged from 10 to 252 bp, with an average of 15.48 bp. The SSR length among different types of SSRs varied a lot, and the abundance of SSR sequences tended to decrease with the increase of repeat number. (3) In designed 140 pairs of primers including different types of SSRs, valid fragments could be produced with 119 pairs in 12 germplasms of Punica granatum and 41 pairs could produce polymorphic fragments with 0.007−0.566 polymorphic information content (PIC). We further screened 15 pairs of primers with polymorphic and stable fragments, which detected 44 alleles in these germplasms of Punica granatum, with average of 2.933 3 alleles per SSR locus.Conclusion SSR markers can be developed in large-scale based on whole genome sequences of Punica granatum and a large number of SSR primers also can be identified for genetic diversity analysis, genetic mapping and variety identification. This study provides lots of SSR information and marker resources for researching genetics and breeding of Punica granatum.
-
Keywords:
- Punica granatum /
- genome /
- SSR /
- marker development /
- polymorphism
-
石榴(Punica granatum)为千屈菜科(Lythraceae)石榴属(Punica)果树,具有很高的观赏、药用和保健价值,经济、生态和社会效益巨大[1]。石榴分布范围广,栽培时间长,在长期的自然选择和人为活动下产生了丰富多样的品种和类型,但由于石榴遗传背景复杂、亲缘关系模糊,难以通过形态特征进行准确的品种鉴定,限制了石榴的品种选育和产业发展[2]。因此,加强石榴遗传多样性分析、遗传图谱构建和品种鉴定技术等研究,对于保障石榴新品种选育和产业健康发展具有重要意义。
分子标记被广泛应用于遗传多样性分析、遗传图谱构建和品种鉴定。在众多分子标记技术中,简单重复序列(simple sequence repeat, SSR,也称微卫星)分子标记以其多态性高、重复性好、共显性、多等位位点变异、成本低廉等优点而受到青睐[3]。Ebrahimi等[4]利用磁珠富集法筛选鉴定了25对石榴基因组SSR引物,利用这些引物对74份来自世界各地的石榴样品成功进行了遗传分析[5]。杨健基于石榴cDNA文库检索到179个SSR位点,从中筛选出8对多态性较高的EST-SSR引物,并对38个石榴品种进行了遗传相似性分析[6]。尽管如此,适用于石榴遗传分析的SSR引物数量仍然有限,难以满足石榴多样性分析和遗传图谱构建等研究需要。2018年石榴全基因组序列的发表[7]为实现大规模SSR位点开发提供了条件。本研究以石榴全基因组序列为基础,分析了石榴基因组SSRs序列特征,并开发了大量引物信息,对不同类型SSR位点的有效性和多态性水平进行了分析,为进一步开展石榴遗传多样性分析、遗传图谱构建和品种鉴定等研究奠定了基础。
1. 材料与方法
1.1 材 料
从山东省枣庄市石榴国家种质资源库中随机选择12个石榴品种(表1)用于SSR引物有效性和多态性水平的鉴定。采集各品种幼嫩叶片,液氮速冻后保存于− 80 ℃冰箱用于DNA提取。
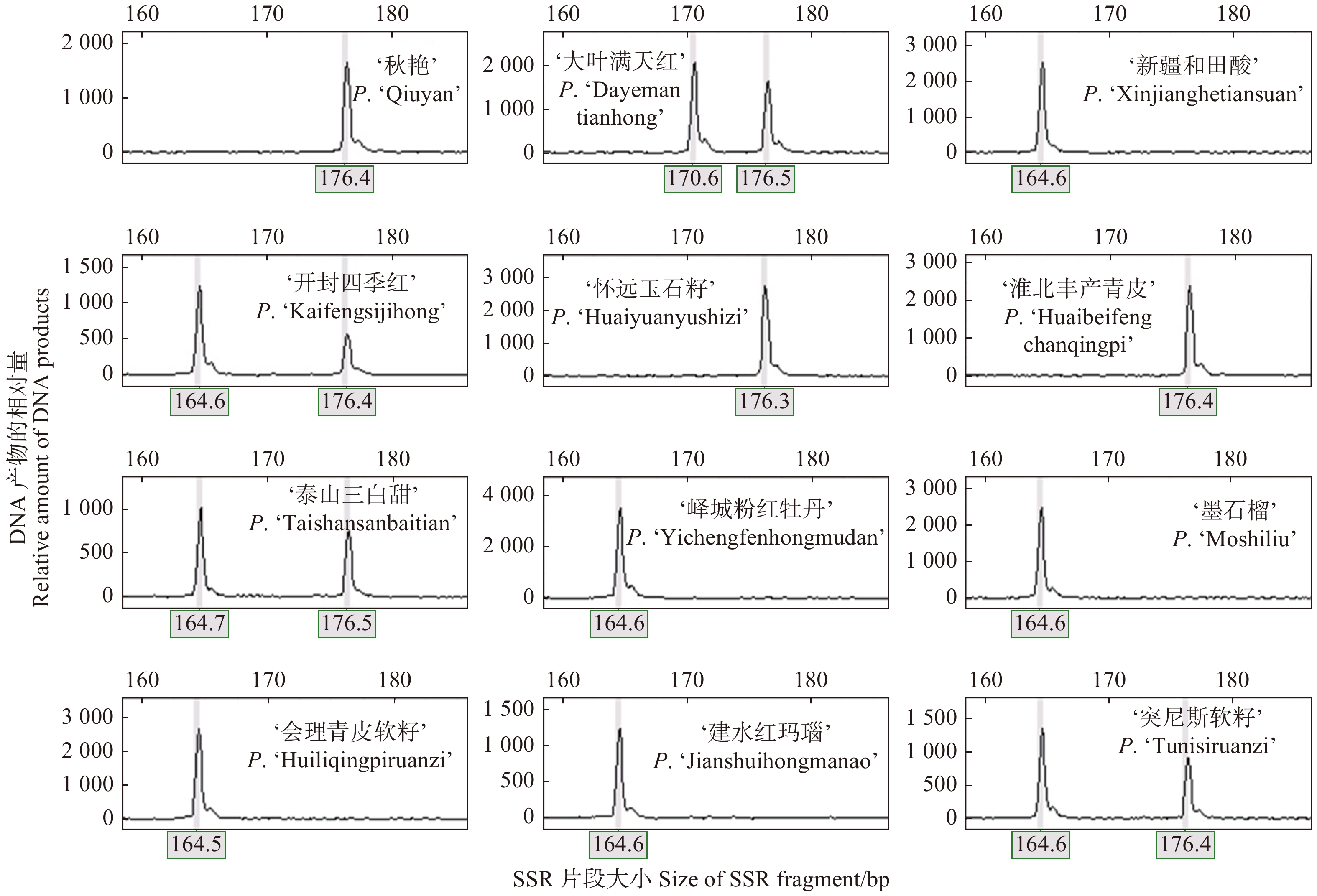
表 1 石榴测试品种编号Table 1. Tested varieties of Punica granatum编号 No. 品种 Variety 来源 Source 1 ‘秋艳’ P. ‘Qiuyan’ 山东枣庄市峄城 Yicheng, Zaozhuang City, Shandong Province 2 ‘大叶满天红’ P. ‘Dayemantianhong’ 河北元氏县 Yuanshi County, Hebei Province 3 ‘新疆和田酸’ P. ‘Xinjianghetiansuan’ 新疆和田 Hetian, Xinjiang Autonomous Region 4 ‘开封四季红’ P. ‘Kaifengsijihong’ 河南开封市 Kaifeng City, Henan Province 5 ‘怀远玉石籽’ P. ‘Huaiyuanyushizi’ 安徽怀远县 Huaiyuan County, Anhui Province 6 ‘淮北丰产青皮’ P. ‘Huaibeifengchanqingpi’ 安徽淮北市 Huaibei City, Anhui Province 7 ‘泰山三白甜’ P. ‘Taishansanbaitian’ 山东泰安市 Taian City, Shandong Province 8 ‘峄城粉红牡丹’ P. ‘Yichengfenhongmudan’ 山东枣庄市峄城 Yicheng, Zaozhuang City, Shandong Province 9 ‘墨石榴’ P. ‘Moshiliu’ 山东枣庄市峄城 Yicheng, Zaozhuang City, Shandong Province 10 ‘会理青皮软籽’ P. ‘Huiliqingpiruanzi’ 四川会理县 Huili County, Sichuan Province 11 ‘建水红玛瑙’ P. ‘Jianshuihongmanao’ 云南建水县 Jianshui County, Yunnan Province 12 ‘突尼斯软籽’ P. ‘Tunisiruanzi’ 突尼斯 Tunisia 1.2 方 法
1.2.1 DNA提取
参考Yuan等[7]的方法,采用改良CTAB法提取石榴测试品种的基因组DNA。经1%琼脂糖凝胶电泳检测后,利用NanoDrop 2000分光光度计(Thermo Scientific)对DNA纯度和浓度进行测定,将A260/A280值在1.8 ~ 2.0之间的DNA样品浓度统一稀释为25 ng/μL后,保存于− 20 ℃冰箱待用。
1.2.2 SSR位点的查找与引物设计
根据已发表的石榴基因组序列数据[7],利用MIcroSAtellite identification tool(MISA,http://pgrc.ipk-gatersleben.de/misa/)软件对石榴基因组序列中的SSR位点进行查找。查找标准为:单、二、三、四、五、六核苷酸重复单元重复次数分别不小于10、6、5、5、5、5次,而且两个SSR之间允许的最大间隔碱基对为100 bp。
借助Primer3软件(v2.3.7; http://primer3.sourceforge.net/)对所查找的石榴基因组SSR位点进行引物设计,参数设置为:引物长度18 ~ 27 nt,最优为20 nt;熔解温度(Tm)为59 ~ 61 ℃,最优为60 ℃;GC含量为40% ~ 60%,最优为50%;预期PCR扩增产物大小为100 ~ 280 bp。
1.2.3 PCR扩增及SSR检测
针对单、二、三、四、五、六核苷酸重复和复合型等不同类型SSRs,各随机挑选20个SSR位点,共设计140对引物,由生工生物工程(上海)股份有限公司进行合成,并用12份石榴样品对其进行有效性鉴定及多态性分析。PCR反应体系为7.5 μL 2 × Taq PCR MasterMix(擎科生物公司),1.5 μL模板DNA(25 ng/μL),0.3 μL M13F(10 μM)上游引物序列结合的荧光标记物,0.06 μL(10 μM)上游引物和0.24 μL(10 μM)下游引物,5.4 μL去离子水补足15 μL的PCR反应体系。其中对M13F序列进行修饰的荧光标记分为FAM、HEX、ROX、TAMRA(睿博兴科公司)4种,以便在同一试验中检测多对引物。PCR扩增程序为:95 ℃预变性5 min;94 ℃变性30 s,56 ℃复性30 s,72 ℃延伸30 s(30个循环);94 ℃变性30 s,53 ℃复性30 s,72 ℃延伸30 s(10个循环);72 ℃延伸10 min。PCR反应产物送至睿博兴科公司进行毛细管电泳检测。
1.2.4 数据统计与分析
统计稳定且易于区分的目的条带,用POPGENE32软件计算具有多态性的SSR位点的等位基因数(Na),有效等位基因数(Ne)和Shannon信息指数(I);用CERVUS 3.0软件[8]分析各SSR位点的多态信息含量值(Polymorphic Information Content, PIC)。Pearson相关分析采用OriginPro 2018完成。
2. 结果与分析
2.1 石榴基因组SSR位点的查找及其组成分析
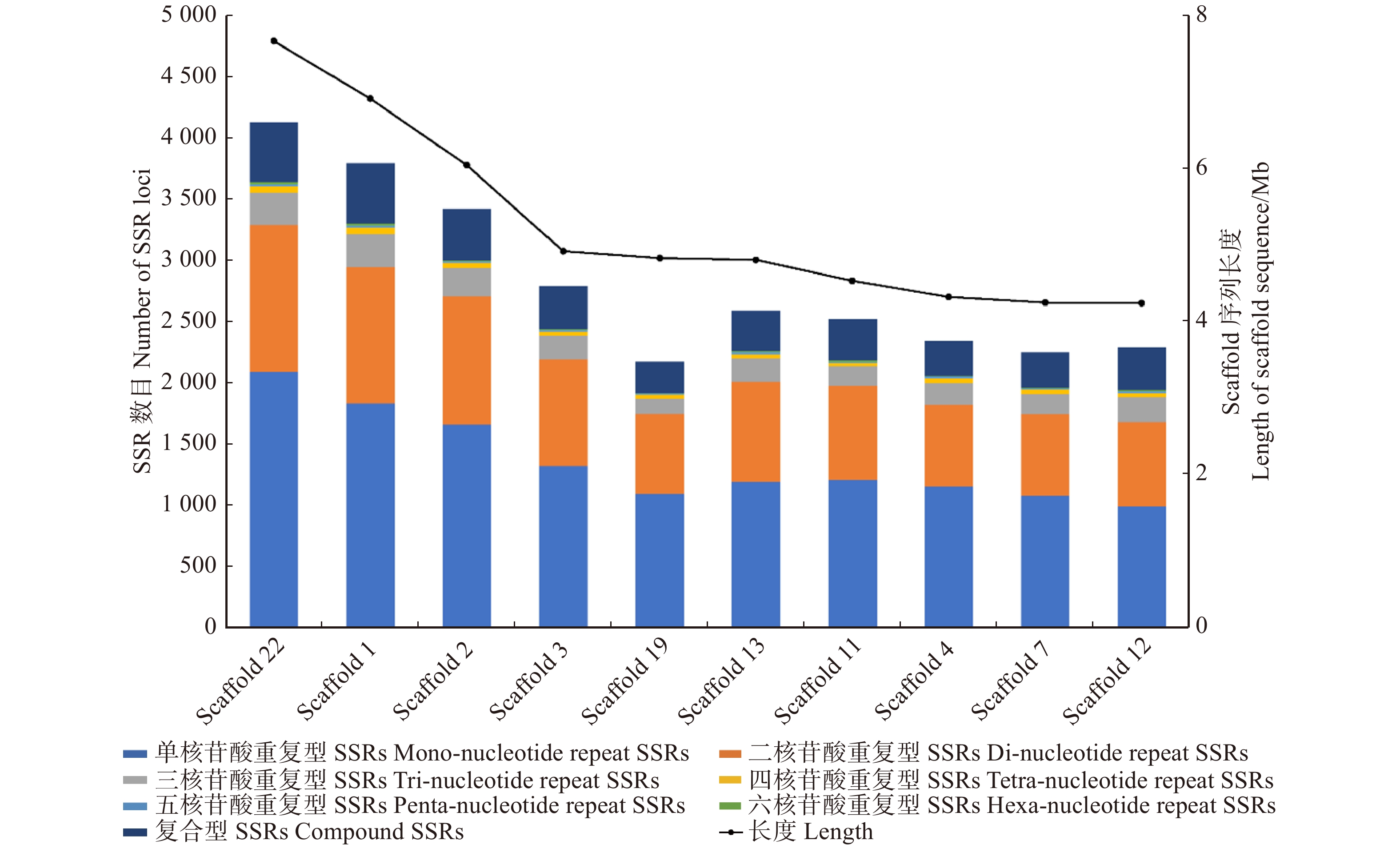
基于石榴基因组测序数据对基因组中1 ~ 6核苷酸重复的SSRs进行了查找分析。共查找到146 445个SSR位点,其中复合型微卫星有19 315个。查找到的SSR位点分布于基因组2 117条Scaffold中的815条,平均每条Scaffold含有179个SSR位点,只含有1个SSR位点的Scaffold有187条。在最长的10条Scaffold上检测到了28 293个SSR位点(图1),占总位点数的19.32%,其中以单核苷酸重复型SSRs最多,占48.20%。Pearson相关分析表明Scaffold长度与查找到的SSR位点数目呈极显著正相关(r=0.991 0,P < 0.000 1)。
在石榴基因组中,平均1 871 bp出现1个SSR位点。其中,单核苷酸重复型SSR含量最多,占总数的51.95%;其次为二核苷酸重复型,占36.58%;六核苷酸重复型最少,仅占0.38%。对重复单元的碱基组合进行统计(表2),发现单核苷酸重复型SSRs以(A/T)n为主,有72 892个,占95.82%;二核苷酸重复型SSRs以(AT/TA)n最多,有34 270个,占63.97%,其次为(AG/CT)n,占29.25%;三核苷酸重复型SSRs以(AAT/ATT)n最多(5 339个),占41.85%,(ACG/CGT)n最少,仅占1.00%;四核苷酸重复型SSRs以(AAAT/ATTT)n所占比例最大,为36.77%,而(ACCC/GGGT)n和(ACCT/AGGT)n均仅查找到1个位点;五核苷酸和六核苷酸重复型SSRs中比例最高的分别为(AAAAG/CTTTT)n和(AAAATC/ATTTTG)n,分别占15.24%和32.50%。
表 2 石榴基因组中部分SSR重复单元的分布特征Table 2. Feature of parts of SSR motifs in Punica granatum genome重复单元 Repeat motif 数目 Number 重复次数 Repeat time 平均长度 Average length/bp 比例 Proportion/% A/T 72 892 10 ~ 56 11.42 49.77 C/G 3 179 10 ~ 222 15.20 2.17 AT/TA 34 270 6 ~ 58 19.07 23.40 CG/GC 163 6 ~ 10 13.30 0.11 AC/GT 3 470 6 ~ 41 16.52 2.37 AG/CT 15 671 6 ~ 126 19.96 10.70 AAC/GTT 326 5 ~ 26 18.51 0.22 AAG/CTT 3 458 5 ~ 43 20.61 2.36 AAT/ATT 5 339 5 ~ 24 21.76 3.65 ACC/GGT 323 5 ~ 12 17.63 0.22 ACG/CGT 127 5 ~ 15 17.55 0.09 ACT/AGT 156 5 ~ 27 19.10 0.11 AGC/CTG 599 5 ~ 19 17.69 0.41 AGG/CCT 1 272 5 ~ 18 18.80 0.87 ATC/ATG 983 5 ~ 16 18.17 0.67 CCG/CGG 173 5 ~ 15 16.63 0.12 2.2 石榴基因组微卫星长度分布及变异
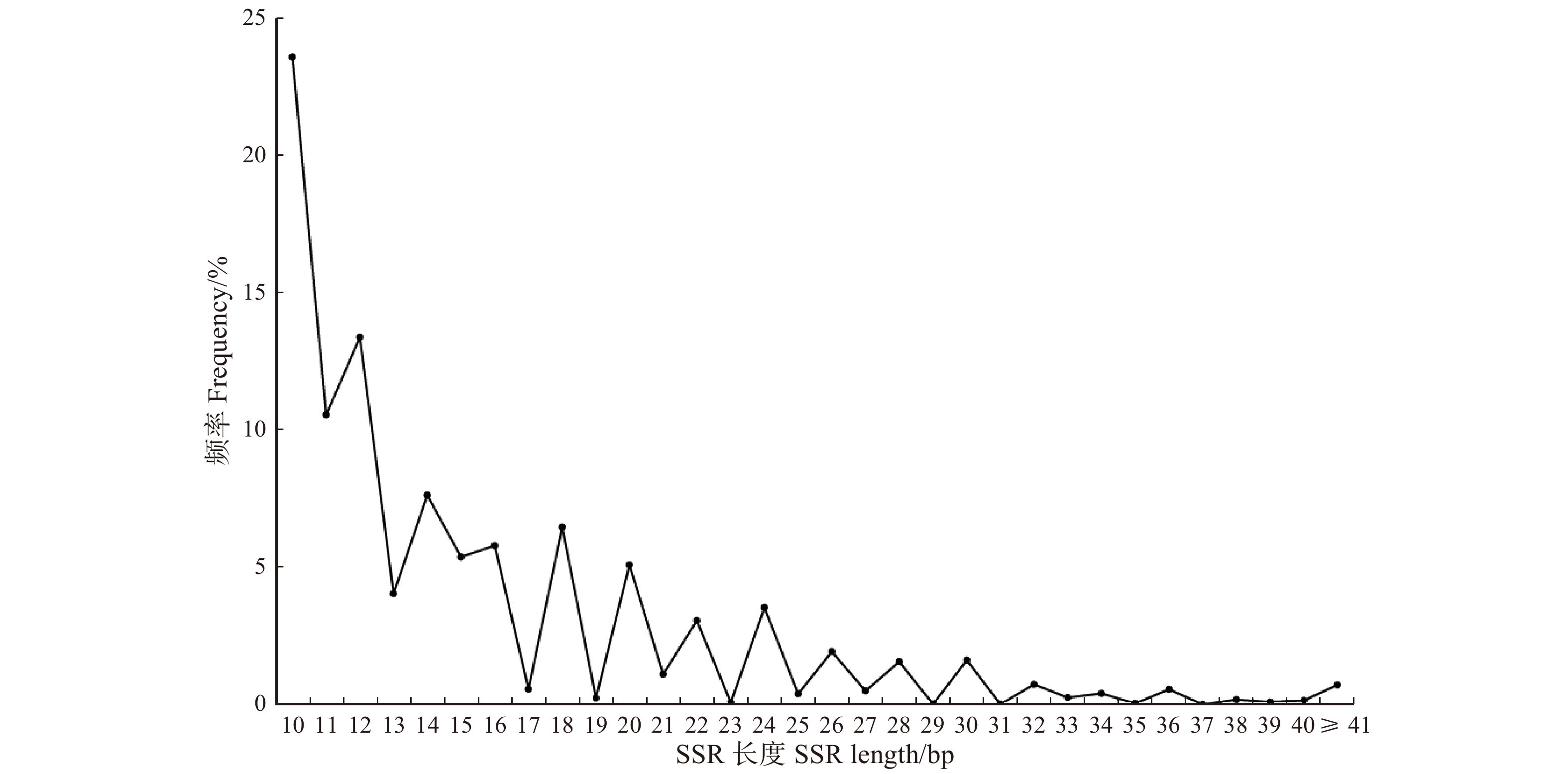
对石榴基因组SSR序列进行分析,发现其长度变化范围为10 ~ 252 bp,平均长度15.48 bp。其中,长度为10 bp的SSR序列所占比例最大,为23.62%,其次为长度12 bp的SSR序列,占13.41%(图2)。
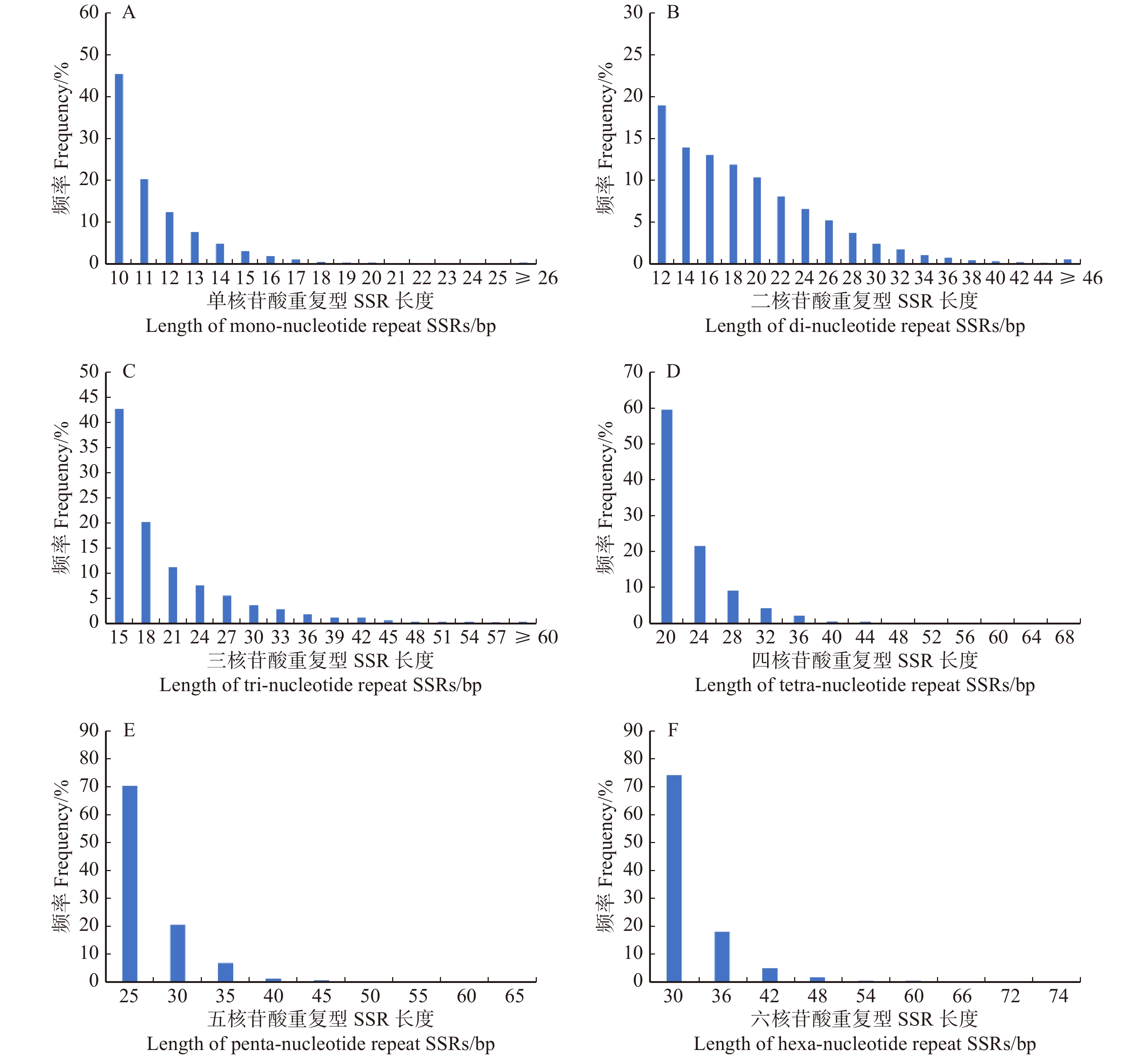
不同长度重复单元的SSR序列长度也存在丰富变异,均呈现随着重复次数的增多,SSR丰度减少的趋势(图3、表2)。二核苷酸重复型SSR序列的长度变化范围最大(12 ~ 252 bp),平均长度为19.15 bp,其中以(AG/CT)n序列平均长度最大,为19.96 bp,(CG/GC)n平均长度最小,仅13.30 bp;单核苷酸重复型SSR序列长度变化范围为10 ~ 222 bp,平均长度为11.58 bp,其中(A/T)n平均长度为11.42 bp,(C/G)n的平均长度为15.20 bp。SSR序列的平均长度与重复单元长度呈正相关(r = 0.979 2,P < 0.000 5)。
2.3 石榴基因组SSR引物的有效性检测及多态性分析
利用12份石榴样品对140对引物进行检测,有132对引物成功扩增,占94.29%。其中,12对引物条带多、峰形差,1对引物的产物小于目的片段范围,因此扩增产物与预期产物一致且较稳定的引物有119对,引物有效率为85%。
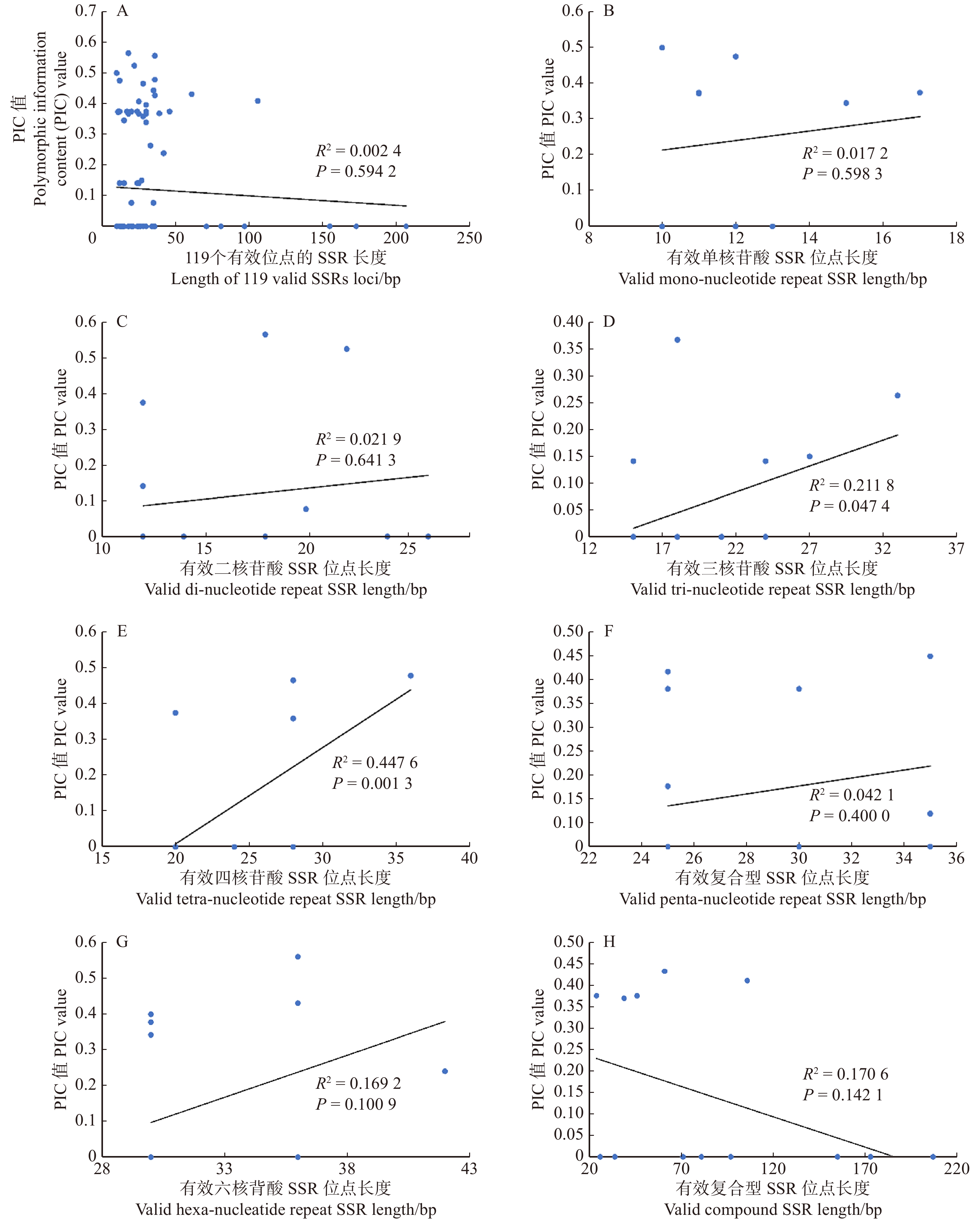
Pearson相关分析发现,该119对引物的PIC值与SSR长度间无显著相关性(图4A)。进一步按照SSR类型分类后,发现三核苷酸和四核苷酸重复型SSR位点的PIC值与SSR长度均呈显著正相关(图4D、4E),而单、二、五、六核苷酸重复型以及复合型SSR位点的PIC值与SSR长度间无显著相关性(图4B、4C、4F、4H)。
筛选到41对SSR引物在12份石榴样品中能扩增出多态性产物,其PIC值在0.007 ~ 0.566之间,平均为0.353。其中,PIC < 0.25的有8对,PIC在0.25 ~ 0.5之间的有29对,PIC > 0.5的有4对。进一步从中挑选出主带清晰、多态性较高、稳定性好的15对引物(表3、图5)进行统计,共检测到44个等位基因,每个位点的等位基因数在2 ~ 5之间,平均等位基因数为2.933 3;有效等位基因数在1.766 9 ~ 2.742 9之间,平均为2.102 7;PIC值在0.359 ~ 0.566之间,平均为0.437,最大值为0.566,最小值为0.359;Shannon信息指数在0.661 6 ~ 1.139 5之间,平均为0.837 4。
表 3 筛选的15对石榴基因组SSR位点引物及多态性信息Table 3. SSR site primer and polymorphic information for selected 15 pairs of pomegranate genomic groups引物名称
Primer IDSSR类型
Type of SSR重复单元
Repeat motif引物序列
Primer sequence等位
基因数
Number of alleles (Na)有效等位
基因数
Effective allelic marker number (Ne)Shannon
信息指数
Shannon diversity index (I)多态
信息含量
Polymorphic information content (PIC)PG_25_548 p2 (AT)9 F: ATATGGCGGCATGAGAGTTC 4.000 0 2.742 9 1.139 5 0.566 R: GTAATAGCATGCCCTTTGCC PG_98_83 p2 (TA)11 F: AGGGGCAAAACCCTTACATC 5.000 0 2.285 7 1.117 1 0.525 R: TCGGCCCAATAAATGGAAT PG_44_17 p3 (AGA)6 F: GGGCGAAGAATTACAGGTGA 2.000 0 1.945 9 0.679 2 0.368 R: ATAATGGCTCCTGTGGAACG PG_140_166 p4 (TATC)9 F: AAAGCATGCGAAAGGATGAT 3.000 0 2.341 5 0.922 2 0.479 R: CAATGCCAATGTTACGGATG PG_4_1646 p4 (AAAT)5 F: CCAAAGGATGAGGAATCGAA 2.000 0 1.882 4 0.661 6 0.359 R: TTTGACCCGATCTACCTCGT PG_105_152 p4 (GTCT)7 F: CGATGTGTACAGTTGTGGGC 3.000 0 2.104 3 0.896 1 0.466 R: CCGTTTTAGCTCCAGTCTGC PG_128_96 p5 (ATAGG)7 F: GAGGAGTCAATTCGACCCAA 3.000 0 2.071 9 0.860 2 0.444 R: CATCGAATTCTATTCCCTCCC PG_13_2441 p5 (AGTTG)5 F: CCGTTTTGGTTTGTTCTGGT 3.000 0 1.967 5 0.792 5 0.408 R: CTCAACTCAACTCCACTTATCTTCA PG_32_463 p5 (TTGTT)5 F: CCCACGTAGGAAGGTGAAAA 2.000 0 1.945 9 0.679 2 0.368 R: GGCTGGCATTTCAGTTCAGT PG_47_500 p5 (AATAT)5 F: TATGCCCGGTGTTAACCAAT 2.000 0 1.945 9 0.679 2 0.368 R: TGATCACTCACACCAGCCTC PG_20_939 p6 (TATAGA)6 F: GGGTCCACAACAACTCCACT 4.000 0 2.594 6 1.109 7 0.557 R: TTTTCCATTCCTTTTCCCCT PG_22_2120 p6 (AATCAA)5 F: CCCCGTTTGGATTCAAAGAT 3.000 0 1.811 3 0.778 1 0.397 R: GAGGGGAAGCTGAGACAGAG PG_66_86 p6 (GAGGGA)6 F: GCTGGTAGGAGTGCTTGAGG 3.000 0 2.133 3 0.829 3 0.428 R: TTCCCCTAATTTGGTGGGTT PG_28_765 p6 (CAAAAT)5 F: TTGTCGACGAACTCGAACAG 2.000 0 2.000 0 0.693 1 0.375 R: TGCATGCAGACAGCTTTAGG PG_117_30 C (GCA)5(GCC)8 F: GGACGAGATACGGCAGAGAC 3.000 0 1.766 9 0.723 3 0.420 R: TGCAGAGGATTGCTGAGATG 平均值 Mean 2.933 3 2.102 7 0.837 4 0.437 3. 讨 论
随着新一代测序技术的不断应用,利用海量测序数据进行分子标记的开发和挖掘已受到广泛重视[9]。本研究利用MISA软件从石榴基因组中共查找到146 445个SSR位点,数量众多,类型多样,为我们进一步开展有效SSR位点的筛选和鉴定奠定了坚实的基础。其中,单核苷酸重复型SSR位点所占比例最高,达51.95%,而六核苷酸重复型SSR位点含量最低,仅0.38%。与杜仲(Eucommia ulmoides)等树种随着重复单元长度的增加,其含量逐渐降低的结果相似[10]。然而,对枣(Ziziphus jujuba)基因组SSR位点特征的研究却发现其优势重复类型为六核苷酸重复(40.1%)[11],可能与不同物种的基因组大小差异有关[12]。
石榴基因组SSR序列存在明显的偏好性。其中,单核苷酸重复型SSRs以(A/T)n为主,占95.82%;二核苷酸重复型SSRs以(AT/TA)n最多,占63.97%;在其他类型SSRs中也存在相似的趋势。显然,A/T碱基在石榴基因组SSR序列中占了主导地位。对水稻(Oryza sativa)等禾本科(Gramineae)植物、毛果杨(Populus trichocarpa)、杜仲、枣等植物基因组SSR序列的研究,也都得到相似的结果[10-11,13-14]。Gur-Arie等[15]认为这种现象可能与富含A/T碱基的重复序列在DNA中解链较易有关。此外,这种偏向性的结果也可能与SSR位点查找工具中的参数设置有关[16]。
石榴基因组SSR序列长度变异丰富(10 ~ 252 bp),平均长度15.48 bp,其中二核苷酸重复型SSR序列的长度变化范围最大(12 ~ 252 bp),单核苷酸重复型SSR序列长度变化范围次之,为10 ~ 222 bp。这种SSR长度的变异可能与进化过程中SSR获得(或丢失)重复单元速率的快慢有关[10]。Weber发现完美重复序列PIC值随着重复次数的增加呈现增加的趋势[17]。本研究Pearson相关分析也表明三核苷酸和四核苷酸重复型SSR位点的PIC值与SSR长度均呈显著正相关,因此,在我们未来选择SSR标记时应相应选择重复次数较多、序列较长的SSR位点,可能会获得更高的多态性水平。
根据Botsein等[18]提出的度量基因座位变异程度高低的多态信息含量标准:当PIC < 0.25时,此位点仅表现为低度多态信息含量;当0.25 < PIC < 0.5时,此位点表现为中度的多态信息含量;当PIC > 0.5时,则该位点表现为高度的多态信息含量。本研究从140对引物中筛选出在12份石榴样品中能扩增出多态性产物的SSR引物41对,其中PIC > 0.5的仅有4对,大部分引物PIC值在0.25 ~ 0.5之间(29对)。可见,本研究所筛选的多态性SSR位点在12份样品中所携带的多态信息总体处于中等水平,这可能与所选择的12个石榴品种间差异不大有关;同时,参考Luo等[19]对136个石榴品种的SSR分析结果,13个SSR位点的PIC值仅在0.14 ~ 0.29之间,推测可能由于长期的驯化以及对芽变的利用偏向,我国石榴品种间的变异水平本身也有限。
石榴栽培历史悠久,品种繁多,单纯以形态学差异等特征作为品种区分标准容易受到环境因素和栽培措施的影响,因此,开发分子标记指纹图谱是开展石榴品种注册、品种鉴定的主要趋势。本研究基于石榴基因组测序数据开发了大量的石榴SSR引物,为进一步开展石榴重要品种和种质的SSR指纹图谱构建奠定了基础,而且充分利用毛细管电泳检测技术成本低、污染少、精确度高等高通量检测特点[20],为实现石榴种质资源的遗传多样性分析、遗传图谱构建等研究提供了可靠的支撑。
-
表 1 石榴测试品种编号
Table 1 Tested varieties of Punica granatum
编号 No. 品种 Variety 来源 Source 1 ‘秋艳’ P. ‘Qiuyan’ 山东枣庄市峄城 Yicheng, Zaozhuang City, Shandong Province 2 ‘大叶满天红’ P. ‘Dayemantianhong’ 河北元氏县 Yuanshi County, Hebei Province 3 ‘新疆和田酸’ P. ‘Xinjianghetiansuan’ 新疆和田 Hetian, Xinjiang Autonomous Region 4 ‘开封四季红’ P. ‘Kaifengsijihong’ 河南开封市 Kaifeng City, Henan Province 5 ‘怀远玉石籽’ P. ‘Huaiyuanyushizi’ 安徽怀远县 Huaiyuan County, Anhui Province 6 ‘淮北丰产青皮’ P. ‘Huaibeifengchanqingpi’ 安徽淮北市 Huaibei City, Anhui Province 7 ‘泰山三白甜’ P. ‘Taishansanbaitian’ 山东泰安市 Taian City, Shandong Province 8 ‘峄城粉红牡丹’ P. ‘Yichengfenhongmudan’ 山东枣庄市峄城 Yicheng, Zaozhuang City, Shandong Province 9 ‘墨石榴’ P. ‘Moshiliu’ 山东枣庄市峄城 Yicheng, Zaozhuang City, Shandong Province 10 ‘会理青皮软籽’ P. ‘Huiliqingpiruanzi’ 四川会理县 Huili County, Sichuan Province 11 ‘建水红玛瑙’ P. ‘Jianshuihongmanao’ 云南建水县 Jianshui County, Yunnan Province 12 ‘突尼斯软籽’ P. ‘Tunisiruanzi’ 突尼斯 Tunisia 表 2 石榴基因组中部分SSR重复单元的分布特征
Table 2 Feature of parts of SSR motifs in Punica granatum genome
重复单元 Repeat motif 数目 Number 重复次数 Repeat time 平均长度 Average length/bp 比例 Proportion/% A/T 72 892 10 ~ 56 11.42 49.77 C/G 3 179 10 ~ 222 15.20 2.17 AT/TA 34 270 6 ~ 58 19.07 23.40 CG/GC 163 6 ~ 10 13.30 0.11 AC/GT 3 470 6 ~ 41 16.52 2.37 AG/CT 15 671 6 ~ 126 19.96 10.70 AAC/GTT 326 5 ~ 26 18.51 0.22 AAG/CTT 3 458 5 ~ 43 20.61 2.36 AAT/ATT 5 339 5 ~ 24 21.76 3.65 ACC/GGT 323 5 ~ 12 17.63 0.22 ACG/CGT 127 5 ~ 15 17.55 0.09 ACT/AGT 156 5 ~ 27 19.10 0.11 AGC/CTG 599 5 ~ 19 17.69 0.41 AGG/CCT 1 272 5 ~ 18 18.80 0.87 ATC/ATG 983 5 ~ 16 18.17 0.67 CCG/CGG 173 5 ~ 15 16.63 0.12 表 3 筛选的15对石榴基因组SSR位点引物及多态性信息
Table 3 SSR site primer and polymorphic information for selected 15 pairs of pomegranate genomic groups
引物名称
Primer IDSSR类型
Type of SSR重复单元
Repeat motif引物序列
Primer sequence等位
基因数
Number of alleles (Na)有效等位
基因数
Effective allelic marker number (Ne)Shannon
信息指数
Shannon diversity index (I)多态
信息含量
Polymorphic information content (PIC)PG_25_548 p2 (AT)9 F: ATATGGCGGCATGAGAGTTC 4.000 0 2.742 9 1.139 5 0.566 R: GTAATAGCATGCCCTTTGCC PG_98_83 p2 (TA)11 F: AGGGGCAAAACCCTTACATC 5.000 0 2.285 7 1.117 1 0.525 R: TCGGCCCAATAAATGGAAT PG_44_17 p3 (AGA)6 F: GGGCGAAGAATTACAGGTGA 2.000 0 1.945 9 0.679 2 0.368 R: ATAATGGCTCCTGTGGAACG PG_140_166 p4 (TATC)9 F: AAAGCATGCGAAAGGATGAT 3.000 0 2.341 5 0.922 2 0.479 R: CAATGCCAATGTTACGGATG PG_4_1646 p4 (AAAT)5 F: CCAAAGGATGAGGAATCGAA 2.000 0 1.882 4 0.661 6 0.359 R: TTTGACCCGATCTACCTCGT PG_105_152 p4 (GTCT)7 F: CGATGTGTACAGTTGTGGGC 3.000 0 2.104 3 0.896 1 0.466 R: CCGTTTTAGCTCCAGTCTGC PG_128_96 p5 (ATAGG)7 F: GAGGAGTCAATTCGACCCAA 3.000 0 2.071 9 0.860 2 0.444 R: CATCGAATTCTATTCCCTCCC PG_13_2441 p5 (AGTTG)5 F: CCGTTTTGGTTTGTTCTGGT 3.000 0 1.967 5 0.792 5 0.408 R: CTCAACTCAACTCCACTTATCTTCA PG_32_463 p5 (TTGTT)5 F: CCCACGTAGGAAGGTGAAAA 2.000 0 1.945 9 0.679 2 0.368 R: GGCTGGCATTTCAGTTCAGT PG_47_500 p5 (AATAT)5 F: TATGCCCGGTGTTAACCAAT 2.000 0 1.945 9 0.679 2 0.368 R: TGATCACTCACACCAGCCTC PG_20_939 p6 (TATAGA)6 F: GGGTCCACAACAACTCCACT 4.000 0 2.594 6 1.109 7 0.557 R: TTTTCCATTCCTTTTCCCCT PG_22_2120 p6 (AATCAA)5 F: CCCCGTTTGGATTCAAAGAT 3.000 0 1.811 3 0.778 1 0.397 R: GAGGGGAAGCTGAGACAGAG PG_66_86 p6 (GAGGGA)6 F: GCTGGTAGGAGTGCTTGAGG 3.000 0 2.133 3 0.829 3 0.428 R: TTCCCCTAATTTGGTGGGTT PG_28_765 p6 (CAAAAT)5 F: TTGTCGACGAACTCGAACAG 2.000 0 2.000 0 0.693 1 0.375 R: TGCATGCAGACAGCTTTAGG PG_117_30 C (GCA)5(GCC)8 F: GGACGAGATACGGCAGAGAC 3.000 0 1.766 9 0.723 3 0.420 R: TGCAGAGGATTGCTGAGATG 平均值 Mean 2.933 3 2.102 7 0.837 4 0.437 -
[1] 苑兆和, 吕菲菲. 石榴文化艺术与功能利用[M]. 北京: 中国农业出版社, 2018. Yuan Z H, Lü F F. Culture, art and functional utilization of pomegranate[M]. Beijing: China Agriculture Press, 2018.
[2] 赵丽娜, 侯乐峰, 郝兆祥, 等. 遗传标记在石榴种质资源研究中的应用[J]. 中国农学通报, 2015, 31(4):135−140. Zhao L N, Hou L F, Hao Z X, et al. Application of genetic markers in Punica granatum germplasm research[J]. Chinese Agricultural Science Bulletin, 2015, 31(4): 135−140.
[3] 黄秦军, 苏晓华, 张香华. SSR分子标记与林木遗传育种[J]. 世界林业研究, 2002, 15(3):14−21. doi: 10.3969/j.issn.1001-4241.2002.03.003 Huang Q J, Su X H, Zhang X H. Microsatellite markers and its application in tree genetics and breeding[J]. World Forestry Research, 2002, 15(3): 14−21. doi: 10.3969/j.issn.1001-4241.2002.03.003
[4] Ebrahimi S, Sayed-Tabatabaei B, Sharifnabi B. Microsatellite isolation and characterization in pomegranate (Punica granatum L.)[J]. Iranian Journal of Biotechnology, 2010, 8(3): 156−163.
[5] Parvaresh M, Talebi M, Sayed-Tabatabaei B. Molecular diversity and genetic relationship of pomegranate (Punica granatum L.) genotypes using microsatellite markers[J]. Scientia Horticulturae, 2012, 138: 244−252. doi: 10.1016/j.scienta.2012.02.038
[6] 杨健. 基于EST-SSR分子标记的中国石榴遗传多样性研究[D]. 合肥: 安徽农业大学, 2015. Yang J. Genetic analysis of pomegranate in China based on EST-SSR markers[D].Hefei: Anhui Agricultural University, 2015.
[7] Yuan Z, Fang Y, Zhang T, et al. The pomegranate (Punica granatum L.) genome provides insights into fruit quality and ovule developmental biology[J]. Plant Biotechnology Journal, 2018, 6(7): 1363−1374.
[8] Marshall T C, Slate J, Kruuk L E B, et al. Statistical confidence for likelihood-based paternity inference in natural populations[J]. Molecular Ecology, 1998, 7(5): 639−655. doi: 10.1046/j.1365-294x.1998.00374.x
[9] Davey J W, Hohenlohe P A, Etter P D, et al. Genome-wide genetic marker discovery and genotyping using next-generation sequencing[J]. Nature Review Genetics, 2011, 12: 499−510. doi: 10.1038/nrg3012
[10] 吴敏, 杜红岩, 乌云塔娜, 等. 杜仲基因组微卫星特征及SSR标记开发[J]. 林业科学研究, 2015, 28(3):387−393. doi: 10.3969/j.issn.1001-1498.2015.03.013 Wu M, Du H Y, Wuyuntana, et al. Characterization of genomic microsatellites and development of SSR markers of Eucommia ulmoides[J]. Forest Research, 2015, 28(3): 387−393. doi: 10.3969/j.issn.1001-1498.2015.03.013
[11] 马秋月, 戴晓港, 陈赢男, 等. 枣基因组的微卫星特征[J]. 林业科学, 2013, 49(12):81−87. Ma Q Y, Dai X G, Chen Y N, et al. Characterization of microsatellites in the genome of Ziziphus jujuba[J]. Scientia Silvae Sinicae, 2013, 49(12): 81−87.
[12] Karaoglu H, Lee C M Y, Meyer W. Survey of simple sequence repeats in completed fungal genomes[J]. Molecular Biology & Evolution, 2005, 22: 639−649.
[13] 郑燕, 张耿, 吴为人. 禾本科植物微卫星序列的特征分析和比较[J]. 基因组学与应用生物学, 2011, 30(5):513−520. doi: 10.3969/gab.030.000513 Zheng Y, Zhang G, Wu W R. Characterization and comparison of microsatellites in Gramineae[J]. Genomics and Applied Biology, 2011, 30(5): 513−520. doi: 10.3969/gab.030.000513
[14] Tuskan G A, Gunter L E, Yang Z K, et al. Characterization of microsatellites revealed by genomic sequencing of Populus trichocarpa[J]. Canadian Journal of Forest Research, 2004, 34: 85−93. doi: 10.1139/x03-283
[15] Gur-Arie R, Cohen C J, Eitan Y, et al. Simple sequence repeats in Escherichia coli: abundance, distribution, composition, and polymorphism[J]. Genome Research, 2000, 10(1): 62−71.
[16] 阎毛毛, 戴晓港, 李淑娴, 等. 松树、杨树及桉树表达基因序列微卫星比对分析[J]. 基因组学与应用生物学, 2011, 30(1):103−109. doi: 10.3969/gab.030.000103 Yan M M, Dai X G, Li S X, et al. Sequence analysis and comparison of EST-SSRs in pine, poplar and Eucalyptus[J]. Genomics and Applied Biology, 2011, 30(1): 103−109. doi: 10.3969/gab.030.000103
[17] Weber J L. Informativeness of human (dC-dA)n·(dG-dT)n polymorphisms[J]. Genomics, 1990, 7: 524−530. doi: 10.1016/0888-7543(90)90195-Z
[18] Botsein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. American Journal of Human Genetics, 1980, 32(3): 314−331.
[19] Luo X, Gao S Y, Hao Z X, et al. Analysis of genetic structure in a large sample of pomegranate (Punica granatum L.) using fluorescent SSR markers[J]. The Journal of Horticultural Science and Biotechnology, 2018, 93(6): 659−665. doi: 10.1080/14620316.2018.1432994
[20] 麻丽颖, 孔德仓, 刘华波, 等. 36份枣品种SSR指纹图谱的构建[J]. 园艺学报, 2012, 39(4):647−654. Ma L Y, Kong D C, Liu H B, et al. Construction of SSR fingerprint on 36 Chinese jujube cultivars[J]. Acta Horticulturae Sinica, 2012, 39(4): 647−654.
-
期刊类型引用(15)
1. 童海浪,张华健,李健,邓红达,韩志强. 四倍体油茶全基因组SSR位点开发与特征分析. 中南林业科技大学学报. 2024(03): 126-135 .  百度学术
百度学术
2. 张雪云,张晓龙,李娜,孔青洋,于超,潘会堂,张启翔,罗乐. 单叶蔷薇基因组SSR标记开发与应用. 中南林业科技大学学报. 2024(06): 186-196 .  百度学术
百度学术
3. 熊远芳,江淑珍,连辉,刘丹,李煜,叶宝鉴,陈世品. 闽楠基因组SSR的开发及其通用性分析. 分子植物育种. 2024(23): 7764-7773 .  百度学术
百度学术
4. 王瑞,洪文娟,罗华,赵丽娜,陈颖,王君. 石榴品种SSR指纹图谱构建及杂种父本鉴定. 园艺学报. 2023(02): 265-278 .  百度学术
百度学术
5. 乐琳琳,杨晓明,郁万文,蔡金峰,汪贵斌,李守科,赵祥树,曹福亮. 基于文冠果(Xanthoceras sorbifolium Bunge.)基因组SSR标记的开发及初步验证. 分子植物育种. 2023(09): 2971-2979 .  百度学术
百度学术
6. 林锦彬,伍永久,韩艺娟,陈松彪,唐唯其. 海带全基因组SSR分子标记开发. 分子植物育种. 2023(12): 3964-3972 .  百度学术
百度学术
7. 冯立娟,程云,王传增,焦其庆,尹燕雷. 基于SSR标记的石榴品种遗传多样性分析和指纹图谱构建. 山东农业科学. 2023(12): 26-33 .  百度学术
百度学术
8. 张芹,王延晖,王冰柯,屈长义. 团头鲂基因组微卫星特征及SSR位点开发. 江苏农业科学. 2022(03): 79-85 .  百度学术
百度学术
9. 左茜茜,宋英杰,马心妍,杨云卉,王轶菲,郭泽光,朱雄智,刘越. 苦荞全基因组SSR位点挖掘及遗传多样性分析. 中国农业科技导报. 2022(04): 38-51 .  百度学术
百度学术
10. 葛大朋,任媛,赵俊,王玉婷,刘学庆,苑兆和. 西藏石榴野生群体的SSR遗传多样性分析. 南京林业大学学报(自然科学版). 2022(03): 127-133 .  百度学术
百度学术
11. 张曼,田娟,孙墨可,董玉迪,巴桑平措,李慧英,贾云峰,牛庆杰. 基于向日葵全基因组序列的SSR标记开发及鉴定. 分子植物育种. 2022(10): 3294-3299 .  百度学术
百度学术
12. 李慧,刘东超,徐瑞瑞,侯立娜,王天琪,刘忠华,付晓,李圣波. 基于RAD-seq技术的金银花SSR标记开发及鉴定. 北京林业大学学报. 2021(06): 108-117 .  本站查看
本站查看
13. 范雪娇,汪贵斌,杨晓明,周婷婷,任重. 银杏转录组SSR位点分布及其序列特征分析. 经济林研究. 2021(02): 63-73 .  百度学术
百度学术
14. 詹海仙,王颖莉,杜晨晖,张丹,李睿,杨梦茹,张朔生. 基于甘草全基因组序列的SSR分子标记开发. 分子植物育种. 2020(18): 6093-6100 .  百度学术
百度学术
15. 陆欢欢,狄怡琳,王小军,范晓东,黄敦元. 大分舌蜂EST-SSR微卫星分子标记开发与特征分析. 重庆师范大学学报(自然科学版). 2020(04): 33-41 .  百度学术
百度学术
其他类型引用(4)



 下载:
下载: