Identification of SAUR gene family in Pinus tabuliformis and analysis on its expression patterns under drought stress
-
摘要:目的
本研究旨在鉴定油松生长素响应基因SAUR家族,分析其基本特征及其在干旱胁迫中的作用,以期为油松及其他针叶树 SAUR基因家族功能解析提供参考。
方法以油松全基因组数据信息为基础,blast比对鉴定出SAUR基因家族,通过生物信息学方法分析其基因结构、氨基酸特性、染色体定位、基因进化、基因功能,并通过RNA-Seq数据分析其在干旱胁迫下的表达模式。
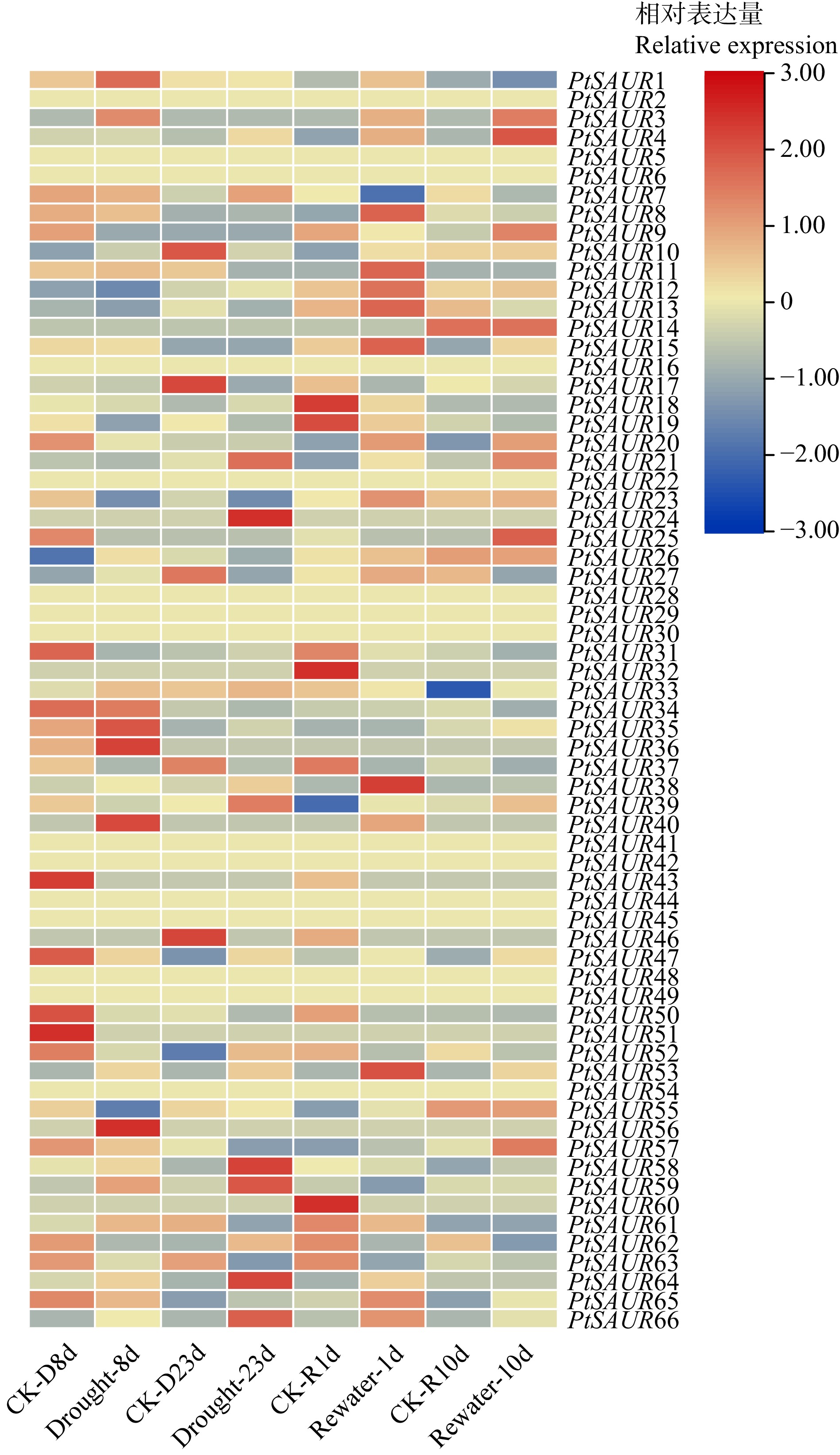
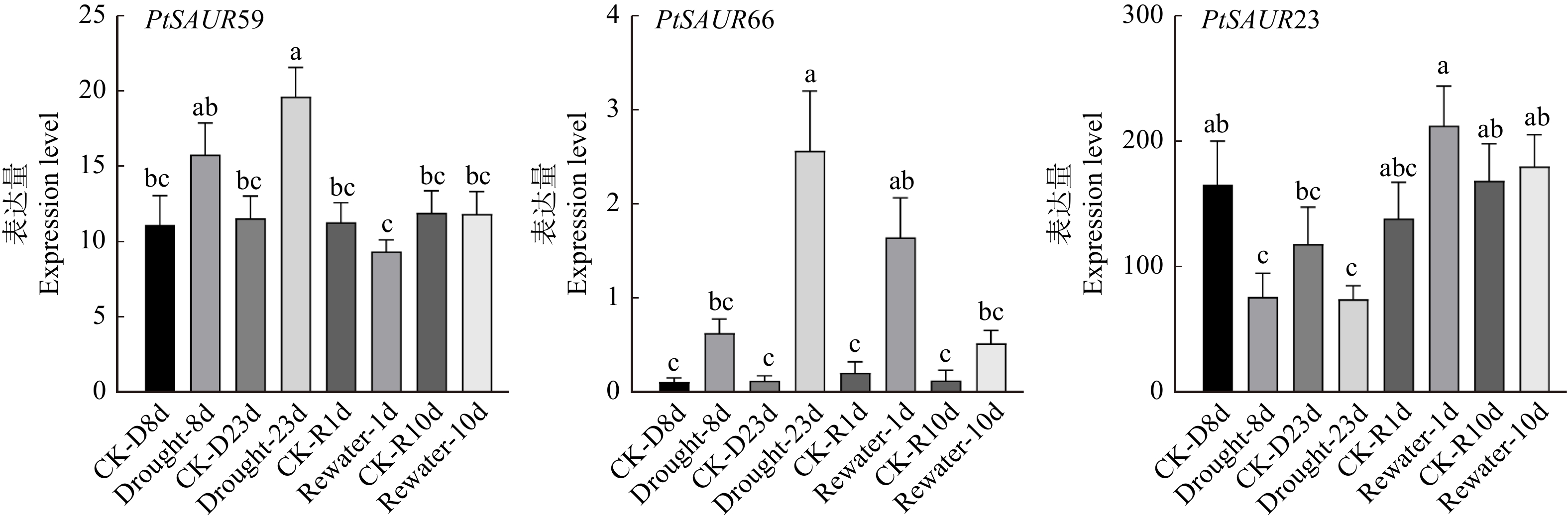
结果(1)在油松中共鉴定出66个SAUR家族基因,命名为PtSAUR1 ~ PtSAUR66,其中 60个SAUR家族成员不均匀地分布在9 条染色体上,多呈现簇状分布。(2)蛋白理化特征分析结果表明,76%的SAUR蛋白呈碱性;亚细胞预测结果显示,74%的SAUR蛋白可定位到细胞核中。(3)油松与银杏和巨杉SAUR基因的共线关系结果显示:与银杏相比,油松与巨杉的同源关系更近。(4)顺式作用元件预测结果显示:SAUR 家族基因的启动子中预测到多种激素(茉莉酸甲酯、脱落酸、生长素等)、非生物胁迫(低温、干旱等)相关的顺式作用元件,其中茉莉酸甲酯相关的元件数最多,生长素相关的元件数最少。(5)系统进化分析显示:油松SAUR家族蛋白可分为7个类群,油松中既存在相近于被子植物的SAUR蛋白,也存在前期分离出来的油松独有的SAUR蛋白。(6)RNA-Seq数据分析结果显示:油松SAUR基因家族对于干旱胁迫具有一定的调节作用,其中PtSAUR23、PtSAUR59和PtSAUR66 3个基因成员变化显著,推测为抗旱的关键基因。
结论油松SAUR家族基因可参与调控干旱胁迫,PtSAUR23、PtSAUR59和PtSAUR66可能在这个过程中起到关键的作用。
Abstract:ObjectiveThis paper aims to identify the small auxin-up RNA (SAUR) family of auxin-responsive genes in Pinus tabuliformis, analyze its basic characteristics and role in drought stress, in order to provide a reference for the functional analysis of SAUR gene family in P. tabuliformis and other conifers.
MethodBased on the whole genome data of P. tabuliformis, the SAUR gene family was identified by blast comparison. The gene structure, amino acid characteristics, chromosome localization, gene evolution, and gene function were analyzed using bioinformatics methods, and the expression patterns under drought stress were analyzed using RNA-Seq data. [Result] (1) A total of 66 SAUR family genes were identified in P. tabuliformis, named PtSAUR1−PtSAUR66. Among them, 60 SAUR family members were unevenly distributed on 9 chromosomes, mostly clustered. (2) The analysis of protein physicochemical characteristics showed that 76% of SAUR proteins were alkaline, and the subcellular prediction results showed that 74% of SAUR proteins can be localized in the nucleus. (3) The collinear relationship of SAUR genes of P. tabuliformis with Ginkgo biloba and Sequoiadendron giganteum showed that, compared with G. biloba, the homologous relationship between P. tabuliformis and S. giganteum was closer. (4) The prediction results of cis-acting elements showed that multiple hormones (methyl jasmonate, abscisic acid, auxin, etc.) and non-biological stress (low temperature, drought, etc.) related cis-acting elements were predicted in the promoters of the SAUR family genes. Among them, the number of elements related to methyl jasmonate was the highest, while the number of elements related to auxin was the lowest. (5) Systematic evolutionary analysis showed that the SAUR family proteins of P. tabuliformis can be divided into 7 groups. There were both SAUR proteins in P. tabuliformis, which was similar to angiosperms, as well as SAUR proteins unique to P. tabuliformis, which was previously isolated. (6) The RNA-Seq data analysis results showed that P. tabuliformis SAUR gene family had a certain regulatory effect on drought stress, with significant changes in the members of PtSAUR23, PtSAUR59, and PtSAUR66 genes, suggesting that they were key genes for drought resistance.
ConclusionThe SAUR family genes of P. tabuliformis can participate in regulating drought stress, among which PtSAUR23, PtSAUR59, and PtSAUR66 may play a key role in this process.
-
Keywords:
- Pinus tabuliformis /
- SAUR gene family /
- drought stress /
- gene expression
-
磷脂酰乙醇胺结合蛋白(PEBP)家族是一类由具有与磷脂酰乙醇胺结合能力的蛋白质组成,这类蛋白广泛存在于植物和动物中[1]。该家族的基因特点是所编码的蛋白有一个非常保守的PEBP/RKIP结构域,占基因编码序列比例的80%[2−3]。PEBP在动物体内通过一种内源性Raf-1激酶发挥作用,在信号转导过程中起着重要的调节作用[4]。同时医学研究中认为PEBP蛋白对于癌症的治疗有着重要的意义,PEBP4基因的表达上调与肿瘤细胞增殖、侵袭及耐药相关[5−6],PEBP4的过表达可提高肺癌细胞中蛋白激酶B(protein kinase B,Akt)和雷帕霉素靶蛋白(mammalian target of rapamycin,mTOR)的磷酸化水平[7]。同时,肺癌和宫颈癌等癌症研究中表明,PEBP4有望成为癌症特异性治疗的新靶点[8−9]。在植物中,目前PEBP基因家族已被证实是植物从营养期向生殖期转变的关键调节者,并决定着植物的某些形态结构构建[10−12]。PEBP基因家族主要分为FT-like、TFL-like和MFT-like 3个亚家族[2],FT-like亚家族有2个成员分别为FT和TSF;MFT-like亚家族只有一个成员,即MFT;TFL-like亚家族有3个成员,分别为TFL1、ATC和BFT[11−12]。FT-like能促进开花,而TFL-like与FT-like的功能相反,TFL-like能抑制茎顶端分生组织形成花原基,延迟开花,具有维持花序无限生长的特点[13−14];MFT-like主要在种子中表达,通过调控ABA和GA信号途径,在种子发育和萌发中起重要作用[15−16]。
PEBP蛋白家族已经在多种植物中被发现,例如葡萄(Vitis vinifera)[17]、拟南芥(Arabidopsis thaliana)[18]、水稻(Oryza sativa)[18]、大豆(Glycine max)[19]、大麦(Hordeum vulgre)[20]、黑杨(Populus nigra)[21]、滴水珠(Pinellia cordata)[22]、陆地棉(Gossypium hirsutum)[23]、水曲柳(Fraxinus mandschurica)[24]及云杉(Picea asperata)[25]等。PEBP基因在被子植物中的研究相对较多,研究发现单子叶植物中PEBP基因家族的基因比双子叶植物中多3 ~ 4倍,且在3个亚家族的分布中也不同[26]。水稻中的Hd3a(Heading date 3a)基因属于FT的同源基因,在水稻和拟南芥中过量表达OsHd3a基因,转基因植株的开花时间均比野生型明显提前[18]。水曲柳中,FmFT基因在雌雄花的不同发育时期表达量不同,雄花在减数分裂时期表达量最高;雌花在胚囊成熟期达到最高值。这表明FmFT在水曲柳雌雄花的发育中起着重要作用[24]。云杉中的PaFTL1和PaFTL2基因在拟南芥中过量表达,转基因植株能表现出晚花表型,这与被子植物中TFL1-like蛋白功能结果相似[25]。此外,研究还发现PEBP基因参与植物的逆境胁迫过程,陆地棉中过表达雪莲(Saussurea involucrata)的SiPEBP基因能够增强陆地棉的抗寒及抗旱性[27]。目前对植物PEBP基因家族研究多集中在调控植物开花时间和植物生长形态上,而在裸子植物及逆境研究中其功能和作用机理尚不明确。
青杄(Picea wilsonii)为松科(Pinaceae)云杉属(Picea)高大绿色乔木,是我国特有的针叶树种。青杄不仅是园林绿化的常用树种,而且被多个地区列为水源涵养林及用材林的主要造林和更新树种[28]。青杄对干旱、阴冷等恶劣环境有较强的适应力,具有较丰富的抗逆基因资源,但利用分子生物学手段开展优质抗逆基因资源的挖掘和利用的研究相对较少,特别是针对PEBP基因调控青杄响应逆境的作用机理尚待深入研究。本文从青杄转录组测序结果中得到一个PwPEBP基因的cDNA序列,通过对PwPEBP蛋白进行生物信息学分析,利用染色体步移法克隆出PwPEBP的启动子并瞬时转化烟草验证其启动子功能,同时利用RT-qPCR分析该基因在不同组织中和不同逆境及激素条件下的表达情况,为深入探究青杄中PEBP家族基因的功能提供理论基础。
1. 材料与方法
1.1 试验材料与处理方法
青杄花粉、根、茎、当年生针叶、成熟叶片及种子均采集于北京植物园,用于PwPEBP的组织特异性表达试验。用于响应逆境的青杄种子置于实验室4 ℃冰箱内保存。将保存的青杄种子播种在含有湿润滤纸的玻璃培养皿上,放置在温度为21 ℃、光照16 h/黑暗8 h的光照培养箱中培养至种子萌发(约10 d开始萌发)。待萌发的芽长至2 ~ 3 cm时,将萌发的种子移到规格为7 cm × 7 cm的育苗盆中(营养土与蛭石的体积比为1:1),放在温度为22 ℃,光照强度为120 μmol/(m 2·s),日照时间为16 h的温室中生长;8周后选取长势一致的青杄幼苗进行相应的逆境响应试验。逆境响应试验的青杄幼苗处理方法是在周燕妮等[29]研究方法的基础上进行略微改动。干旱处理:将青杄幼苗在裸根状态下置于滤纸上室温(25 ℃)下干旱处理0、3、6和12 h;盐胁迫处理:将裸根的青杄幼苗用200 mL浓度为200 mmol/L的NaCl溶液浸泡在培养皿中0、3、6和12 h;冷处理:将整株带土的青杄幼苗置于4 ℃冰箱中处理0、3、6和12 h;高温处理:将整株带土的青杄幼苗放置在42 ℃的光照培养箱中处理0、3、6和12 h。为了验证PwPEBP启动子序列对预测中顺式作用元件的响应情况,分别用200 mL的100 μmol/L ABA、100 μmol/L MeJA、400 μmol/L GA和500 μmol/L SA溶液将青杄裸根幼苗浸泡在培养皿中0、3、6和12 h[30]。处理后用液氮速冻,放置于− 80 ℃冰箱中保存备用。每个处理选取4棵长势一致的青杄幼苗,设3次重复。
验证启动子活性试验所选用的烟草品种为“本氏”烟草,由实验室常温保存。将烟草种子播种于盆中(营养土与蛭石的体积比为1:1),置于温室(温度为22 ℃,光照强度为120 μmol/(m 2·s),日照时间为16 h)中,待其萌发2周后,将其进行单株移栽。待烟草正常生长5 ~ 6周时选取长势一致的烟草进行实验。处理的激素浓度及逆境条件与青杄一致,干旱略有差别,烟草采用喷施20%的PEG模拟干旱[31],烟草的处理方法为相对应浓度的处理液对注射过农杆菌的烟草叶片进行连续喷施处理12 h。
1.2 试验方法
1.2.1 PwPEBP cDNA的获得及PwPEBP生物信息学分析
PwPEBP的cDNA是以本实验室前期干旱处理的青杄幼苗为材料进行转录组测序所得,测序由博瑞德生物科技(Breeding Biotechnologies)公司完成。根据所得序列设计引物pEASY-T1-PwPEBP-F、pEASY-T1- PwPEBP-R(表1),从青杄cDNA文库克隆得到PwPEBP的ORF,连接到pEASY-T1上,获得PwPEBP单克隆载体。用DNAMAN软件进行序列的蛋白分析;利用在线软件ProParam(http://web.Expasy.org/compute_pi/和http://web.Expasy.org/protpa-ram/)预测该蛋白分子式、等电点、相对分子质量和不稳定指数;运用ProtParam(http://biopython.org/wiki/ProtPAram)分析氨基酸组成;运用SignalP(http://www.cbs.dtu.dk/services/SignalP/)做信号肽预测;利用ProtScale(http://web.expasy.org/protscale/)分析蛋白疏水性;用NetPhos 3.1(http://www.cbs.dtu.dk/services/NetPhos/)预测蛋白的磷酸化位点;运用TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)预测跨膜结构域;用GOR4在线工具(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)预测蛋白二级结构。根据朱岩等[27]对PEBP家族基因在植物中功能的研究进展及NCBI数据库,获得其他物种中PEBP蛋白的同源氨基酸序列,并用ClustalX2.0做多序列对比图及MEGA5.0构建系统发育进化树。
表 1 所用引物序列Table 1. Primer sequences引物功能 Primer function 引物名称 Primer name 引物序列 Primer sequence (5′−3′) pEASY-T1-PwPEBP引物
Primer for pEASY-T1-PwPEBPpEASY-T1-PwPEBP-F
pEASY-T1-PwPEBP-RGAGAGATCAATATGGCGCAGGTAG
ACGGGCATGTAAGAGTCGCRT-qPCR-PwPEBP 引物
Primer for RT-qPCR-PwPEBPRT-PwPEBP-F
RT-PwPEBP-RCCTCCCACACTCAAAGGTCTG
TCGATGATCCCCAACAGGTG染色体步移特异性引物
Primers for the genome walkingPwPEBP-SP1
PwPEBP-SP2
PwPEBP-SP3ACTCTAATGGCGGAGACAAATCTTTC
TGTCAGCCTGAATACTTGTTCCTGTG
CAAAATTTTCTACATCTACCTGCGCCATpEASY-T1-PwPEBP promoter引物
Primers for pEASY-T1-PwPEBP promoterpEASY-T1-PwPEBPp-F
pEASY-T1-PwPEBPp-RCCCTTTGTCAGCCTGAATACTTGTTC
ATTGATCTCTCGTCCACAATTTCAACpBI121-PwPEBP promoter::GUS引物
Primers for pBI121-PwPEBP promoter::GUSpBI121-PwPEBP-F
pBI121-PwPEBP-RAAA$\scriptstyle \underline {{\rm{AGTACT}}} $CCCTTTGTCAGCCTGAATACTTGTTC
CG$\scriptstyle \underline {{\rm{GGATCC}}} $ATTGATCTCTCGTCCACAATTTCAACRT-qPCR内参引物
Primer for RT-qPCR reference genePwEF1-α-F
PwEF1-α-RAACTGGAGAAGGAACCCAAG
AACGACCCAATGGAGGATACP0800-PwPEBP promoter::LUC引物
Primers for P0800-PwPEBP promoter::LUCP0800-PwPEBP-F
P0800-PwPEBP-RGG$\scriptstyle \underline {{\rm{GGTACC}}} $CCCTTTGTCAGCCTGAATACTTGTTC
CG$\scriptstyle \underline {{\rm{GGATCC}}} $ATTGATCTCTCGTCCACAATTTCAAC注:下划线的部分表示酶切位点。Note: the underlined part represents the restriction site. 1.2.2 PwPEBP启动子序列的克隆
利用北京诚聚德安泰有限公司的植物基因组DNA快速提取试剂盒提取青杄针叶的基因组DNA,根据TakaRa公司的染色体步移Genome-Walking试剂盒的引物设计要求,在PwPEBP编码区的5′端设计3个向5′端的特异性巢式PCR后引物PwPEBP-SP1、PwPEBP-SP2、PwPEBP-SP3(表1)。以提取的基因组DNA为模板进行扩增,经过3轮巢式PCR,进行3次染色体步移,直到获得PwPEBP的开放阅读框(ORF)的起始密码子ATG上游的903 bp的碱基序列。pEASY-T1载体从北京全式金公司购买,测序由北京擎科生物公司完成。在所获得的片段上设计pEASY-T1-PwPEBPp-F和pEASY-T1-PwPEBPp-R引物(表1),用青杄的基因组DNA为模板进行PCR,连接到pEASY-T1载体上并对大肠杆菌TransT1进行转化,经过菌落PCR鉴定阳性菌落后,由北京擎科生物公司测序得到碱基片段,与染色体步移法克隆出来的片段比对一致后,进一步验证启动子的准确性。将903 bp启动子序列在在线数据库PlantCARE(http://bioinformatics.psb.ugent.be/webtools/plantcare/html)中预测其可能存在的顺式作用元件,利用在线生物信息学软件BDGP(http://www.Fruitfly.org/seq_tools/promoter.html)预测PwPEBP基因启动子的基础启动子和转录起始位点。
1.2.3 启动子表达载体的构建及在烟草叶片中的瞬时表达
构建带有GUS标签的表达载体PBI121(实验室− 80 ℃冰箱中保存)及带有Luc标签的表达载体pGreenⅡ0800(实验室− 80 ℃冰箱中保存),以下简称P0800载体。PBI121表达载体用于PwPEBP启动子的定性实验,P0800载体用于PwPEBP启动子的定量实验。根据PwPEBP的启动子序列设计PBI121和P0800引物(表1)。以PwPEBP单克隆Peasy-T1载体为模板,经过PCR、酶切、连接等步骤,将PwPEBP启动子序列构建到PBI121及P0800载体上。经过菌落PCR鉴定出阳性菌落,并由北京擎科生物公司测序获得正确结果,随后将重组质粒PB121-PwPEBP-p::GUS、P0800-PwPEBPp::Luc及空PBI121载体质粒转入农杆菌GV3101中。通过菌落PCR对菌落进行鉴定,并将鉴定后呈阳性的菌落挑于2 mL的YEB液体筛选培养基(50 mg/L卡那霉素 + 25 mg/L利福平)中,于28 ℃摇床上180 r/min进行过夜培养,待菌液OD600值为0.8 ~ 1.2时,将菌液重悬(5 000 r/min离心5 min),弃上清;用1 mL烟草缓冲液(10 mmol/L MES,10 mmol/L氯化镁,10 μmol/L乙酰丁香酮,pH为5.7)继续重悬2次,弃上清;最后用烟草缓冲液将菌体OD600值调为0.5左右,于室温中平放静置2 h后用于农杆菌瞬时转化实验。
以注射PBI121空载体的农杆菌作为阳性对照,烟草缓冲液作为阴性对照,含有pBI121-PwPEBP promoter::GUS质粒的农杆菌为实验组。分别将其注射到生长5 ~ 6周的烟草叶片中,待其正常生长24 ~ 36 h后,对阳性对照和阴性对照喷施清水,对注射含有pBI121-PwPEBP promoter::GUS质粒的农杆菌分别持续喷施ABA、GA、SA、MeJA、20% PEG、NaCl、清水12 h;对于高温和低温处理,将注射后的烟草分别在4 ℃和42 ℃条件下培养12 h。剪下喷施部位叶片,用GUS染色液浸泡染色后,在37 ℃的烘箱中黑暗处理12 ~ 24 h,之后用75%的酒精对其进行脱色处理,直至阴性对照的叶片变为白色,利用体式显微镜观察其染色情况。
为了进一步了解PwPEBP启动子的活性,以相同的方式处理注射了P0800-PwPEBP promoter::Luc的烟草,以喷施清水为对照。随后取注射点周围的叶片,剪碎磨样,用碧云天生物公司(上海)的双荧光素酶报告基因检测试剂盒对其进行蛋白提取,最后用荧光检测仪器Promega进行荧光测定,用测定的Luc/Ren比值进行PwPEBP启动子活性定量实验分析[32−33]。
1.2.4 PwPEBP组织特异性表达及逆境响应和激素响应分析
利用华越洋生物有限公司(北京)的植物快速提取RNA试剂盒提取各处理的青杄幼苗及青杄各组织部位的RNA,并用该公司的反转录试剂盒合成第1条cDNA链,置于− 20 ℃保存,以此为RT-qPCR的模板。根据基因序列的非保守区设计定量引物为RT-PwPEBP-F和RT-PwPEBP-R。选取青杄延伸因子蛋白EF1-α[34]作为内参基因,引物分别为PwEF1-α-F和PwEF1-α-R(表1)。将合成的cDNA均一化浓度后在StepOnePlus Real Time RT-PCR(ABI公司)仪器上进行实时荧光定量PCR,生物学重复3次,技术重复3次。并利用2− ΔΔCT法分析数据,用SPSS18与SigmaPlot 10.0分析数据并作图。
2. 结果与分析
2.1 PwPEBP基因的克隆与生物信息学分析
PwPEBP的cDNA是由本实验室前期青杄转录组测序获得,长度为1 408 bp。利用DNAMAN软件对其序列进行分析发现,在第455 bp处出现起始密码子ATG,在第1 039 bp处出现终止密码子TAA。开放阅读框(ORF)长度为585 bp,共编码194个氨基酸(图1)。
![]() 图 1 PwPEBP核苷酸序列及其编码区的氨基酸序列方框内为PwPEBP的起始密码子(ATG)和终止密码子(TAA),下划线表示配体结合的保守位点。The starting codon (ATG) and the stop codon (TAA) of the PwPEBP are showed in the box. The conserved sites of PwPEBP combined with ligand are underlined.Figure 1. Nucleotide sequence of PwPEBP and protein amino acid sequence in coding region
图 1 PwPEBP核苷酸序列及其编码区的氨基酸序列方框内为PwPEBP的起始密码子(ATG)和终止密码子(TAA),下划线表示配体结合的保守位点。The starting codon (ATG) and the stop codon (TAA) of the PwPEBP are showed in the box. The conserved sites of PwPEBP combined with ligand are underlined.Figure 1. Nucleotide sequence of PwPEBP and protein amino acid sequence in coding region经NCBI BLAST分析,确定该基因为PEBP基因,利用不同的生物信息学在线软件对PwPEBP进行分析可知,PwPEBP的分子式为C966H1 484N250O299S6,相对分子质量为21.58 kDa,pI值为4.57。其中脯氨酸(Pro)含量所占比例最高,为9.8%;其次是甘氨酸(Gly)和谷氨酸(Glu),均为8.8%;不稳定指数为40.57,略高于40的阈值,说明该蛋白不稳定。疏水性分析发现,位于该蛋白第186位的丙氨酸(Ala)分值最大,为2.322;位于第133位的天冬氨酸(Asn)分值最小,为− 2.100,且疏水位点与亲水位点分布均匀,所以推测该蛋白为亲水蛋白(图2A)。磷酸化位点预测结果显示该蛋白有15个丝氨酸(Ser)磷酸化位点,6个苏氨酸(Thr)磷酸化位点,4个酪氨酸(Tyr)磷酸化位点(图2B)。信号肽预测结果显示PwPEBP蛋白不具有信号肽结构域(图2C)。跨膜预测结果显示PwPEBP跨膜肽链在膜外测,不具有跨膜结构域(图2D)。PwPEBP蛋白二级结构预测结果发现,PwPEBP中α-螺旋含量为33.51%,延伸链含量为10.31%,不具有β-转角结构,无规则卷曲结构含量达到56.19%(图2E);亚细胞定位预测结果显示,该蛋白主要定位在细胞核和细胞质中。在NCBI网站上搜索不同物种PEBP蛋白的氨基酸序列,用ClustalX 2.0进行多序列比对分析,结果显示,PwPEBP序列与其他物种中的PEBP在C端相似性较高,N端差异性较大,其中PwPEBP蛋白含有PEBP家族特有的保守位点DPDXP基序[35],在C端有一段PwPEBP特有的氨基酸序列(图3)。利用MEGA5.0邻接法构建系统发育树,结果显示PwPEBP与北美云杉(Picea sitchensis)的PEBP单独聚为一簇,属于PEBP家族中新的PEBP亚家族(图4)。
![]() 图 2 PwPEBP理化性质分析A. 蛋白疏水性;B. 蛋白磷酸化位点;C. 蛋白信号肽结构域(C-score代表剪切位置分值,S-score代表信号肽分值,Y-score代表综合剪切位置分值);D. 蛋白的跨膜结构域;E. 蛋白的二级结构分析。A, protein hydrophobic analysis; B, protein phosphorylation sites; C, signal peptide domain of protein (C-score means cleavage site score, S-score means signal peptide score, Y-score means combined cleavage site score); D, protein transmembrane domains; E, two level structure analysis of PwPEBP protein.Figure 2. Analysis on PwPEBP physicochemical properties
图 2 PwPEBP理化性质分析A. 蛋白疏水性;B. 蛋白磷酸化位点;C. 蛋白信号肽结构域(C-score代表剪切位置分值,S-score代表信号肽分值,Y-score代表综合剪切位置分值);D. 蛋白的跨膜结构域;E. 蛋白的二级结构分析。A, protein hydrophobic analysis; B, protein phosphorylation sites; C, signal peptide domain of protein (C-score means cleavage site score, S-score means signal peptide score, Y-score means combined cleavage site score); D, protein transmembrane domains; E, two level structure analysis of PwPEBP protein.Figure 2. Analysis on PwPEBP physicochemical properties![]() 图 4 PwPEBP进化树分析该进化树是利用MEGA5.0软件构建的,计算方法为邻位相连法,各分支上的数字代表1 000次重复的置信度,*号表示本研究的基因PwPEBP。The evolutionary tree is constructed by MEGA 5.0 software, and the calculation method is neighborhood connection method. The numbers on each branch represent the confidence of 1 000 repetitions. * indicates the gene PwPEBP in this study.Figure 4. Evolutionary tree analysis of PwPEBP
图 4 PwPEBP进化树分析该进化树是利用MEGA5.0软件构建的,计算方法为邻位相连法,各分支上的数字代表1 000次重复的置信度,*号表示本研究的基因PwPEBP。The evolutionary tree is constructed by MEGA 5.0 software, and the calculation method is neighborhood connection method. The numbers on each branch represent the confidence of 1 000 repetitions. * indicates the gene PwPEBP in this study.Figure 4. Evolutionary tree analysis of PwPEBP2.2 PwPEBP基因启动子序列的克隆与顺式作用元件及转录起始位点的预测
对启动子序列基础区域及转录起始位点进行预测,结果如图5所示,在− 615 ~ − 765有一段基础启动子区域,并有一个转录起始位点。利用在线软件通过染色体步移法克隆出了903 bp启动子序列,序列如图4所示。利用在线软件BDGP对启动子序列进行顺式元件分析,结果显示PwPEBP启动子序列中具有保守核心元件TATA-box和增强元件CAAT-box,符合真核生物启动子的基本结构特征,并且启动子序列中包含了很多与激素相关的响应元件和响应环境胁迫相关的元件(表2)。激素响应元件包括ABA响应元件ABRE、SA响应元件TCA-element、MeJA响应元件CGTCA-motif和TGACG-motif、GA响应元件GARE-motif;光调控响应元件有G-box、ATCT-motif、GA-motif、chs-CMA2a、I-box;响应低温的元件为LTR,启动子序列中还包含很多与MYB和MYC互作的元件。
表 2 PwPEBP启动子序列响应元件分析Table 2. PwPEBP promoter sequence response element analysis功能
Function顺式作用元件
Cis-acting element核心序列
Core sequence位置
Postion/bp光响应顺式作用元件
Cis-acting elements for light responseATCT-motif
G-box
GA-motif
I-box
chs-CMA2aAATCTAATCC
CACGTT
CACGTC
ATAGATAA
gGATAAGGTC
TCACTTGA− 378 (+)
− 453 (−)
− 193 (−)
− 465 (+); − 736 (−)
− 288 (−)
− 823 (+)ABA响应顺式作用元件
Cis-acting elements for ABA responseABRE ACGTG − 194 (+); − 454 (+) 低温响应顺式作用元件
Cis-acting elements for low temperature responseLTR CCGAAA − 265 (+); − 816 (−) 赤霉素响应顺式作用元件
Cis-acting elements for gibberellin responseGARE-motif TCTGTTG − 514 (−) 茉莉酸响应顺式作用元件
Cis-acting elements for jasmonic acid responseCGTCA-motif
TGACG-motifCGTCA
TGACG− 422 (−)
− 422 (+)水杨酸响应顺式作用元件
Cis-acting elements for salicylic acid responseTCA-element CCATCTTTT − 91 (+) MYB
Myb
Myb-binding site
MYC
MycCAACAG
TAACTG
CAACAG
CATTTG
TCTCTTA− 514 (+); − 875 (−)
− 301 (+)
− 514 (+)
− 585 (−)
− 470 (−)注:括号内“+”表示正向序列,“−”表示互补序列。Notes: “+” in bracket represents forward sequence; “−” in bracket represents complementary sequence. 2.3 PwPEBP启动子序列的功能验证
利用注射农杆菌瞬时转化烟草叶片的方法,对PwPEBP启动子序列进行功能验证。注射的烟草叶片通过GUS组织化学染色。结果表明,在清水处理下,阳性对照叶片呈深蓝色,说明GUS基因强烈表达(图6A),阴性对照叶片为白色,说明GUS基因不表达(图6B),实验组叶片显示有蓝色(图6C),说明GUS基因在瞬时转化PwPEBP promoter::GUS的烟草叶片中有表达。在ABA(图6D)、SA(图6E)、GA(图6F)、MeJA(图6G)、干旱(图6H)、低温(图6J)和高温(图6K)处理下,GUS均有表达,且蓝色均比清水处理深,这与启动子中预测含有ABA、GA、MeJA、SA及低温顺式作用元件相符合,但在盐处理下(图6I)无响应。PwPEBP启动子活性定量分析结果显示与GUS表达趋势相一致(图7),在干旱处理下,PwPEBP启动子活性最强,Luc/Ren比值是清水处理的5.6倍(图7)。
![]() 图 6 不同激素和逆境处理下PwPEBP启动子在烟草叶片中的GUS染色A. 注射了含有CaMV35S::GUS的PBI121空载体农杆菌(阳性对照,清水);B. 注射了烟缓冲液(阴性对照,清水);C~K. 注射了带有PwPEBP promoter::GUS农杆菌,处理分别为清水(C)、ABA(D)、SA(E)、GA(F)、MeJA(G)、PEG(H)、NaCl(I)、4 ℃(J)及42 ℃(K);A, injected with CaMV35S::GUS-containing Agrobacterium pBI121 empty carrier (positive control, water); B, injected with smoke buffer (negative control, water); C-K, injected with PwPEBP promoter GUS Agrobacterium, respectively, with clear water (C), ABA (D), SA (E), GA (F), MeJA (G), PEG (H), NaCl (I), 4 ℃ (J) and 42 ℃ (K).Figure 6. GUS staining of PwPEBP promoter in Nicotiana tabacum leaves under different hormones and stress treatments
图 6 不同激素和逆境处理下PwPEBP启动子在烟草叶片中的GUS染色A. 注射了含有CaMV35S::GUS的PBI121空载体农杆菌(阳性对照,清水);B. 注射了烟缓冲液(阴性对照,清水);C~K. 注射了带有PwPEBP promoter::GUS农杆菌,处理分别为清水(C)、ABA(D)、SA(E)、GA(F)、MeJA(G)、PEG(H)、NaCl(I)、4 ℃(J)及42 ℃(K);A, injected with CaMV35S::GUS-containing Agrobacterium pBI121 empty carrier (positive control, water); B, injected with smoke buffer (negative control, water); C-K, injected with PwPEBP promoter GUS Agrobacterium, respectively, with clear water (C), ABA (D), SA (E), GA (F), MeJA (G), PEG (H), NaCl (I), 4 ℃ (J) and 42 ℃ (K).Figure 6. GUS staining of PwPEBP promoter in Nicotiana tabacum leaves under different hormones and stress treatments![]() 图 7 不同激素和逆境处理下PwPEBP的启动子在烟草叶片中的活性表达利用单因素方差分析进行差异显著性分析,多重比较方法为Duncan法,不同小写字母表示差异显著性(P < 0.05)。下同。Single factor analysis of variance is used to analyze the difference, Duncan test is used as the multiple comparison method, and different normal letters indicate significant difference (P < 0.05). The same below.Figure 7. Active expression analysis of PwPEBP promoter in Nicotiana tabacum leaves under different hormones and stress treatments
图 7 不同激素和逆境处理下PwPEBP的启动子在烟草叶片中的活性表达利用单因素方差分析进行差异显著性分析,多重比较方法为Duncan法,不同小写字母表示差异显著性(P < 0.05)。下同。Single factor analysis of variance is used to analyze the difference, Duncan test is used as the multiple comparison method, and different normal letters indicate significant difference (P < 0.05). The same below.Figure 7. Active expression analysis of PwPEBP promoter in Nicotiana tabacum leaves under different hormones and stress treatments2.4 PwPEBP在组织及逆境和激素处理下的表达特性分析
通过RT-qPCR实验探究PwPEBP基因在各组织及逆境和激素处理条件下的表达情况,结果显示,PwPEBP在不同逆境处理下的表达特性如图8所示。PwPEBP表达量在干旱处理3 h和6 h时缓慢上升,处理12 h时,表达量显著升高,为0 h的28.41倍(图8A);PwPEBP在低温处理6 h(图8B)和高温处理3 h时(图8C),与0 h相比表达量均显著下降,分别为0 h的0.18倍和0.25倍,处理12 h时无显著变化。PwPEBP在盐胁迫下,与0 h比,处理3、6、12 h的表达量无显著变化(图8D)。在不同激素处理中,PwPEBP表达量均有显著的变化(图8E ~ 8H)。在ABA处理12 h时,PwPEBP表达量显著升高,为0 h的2.61倍,处理3 h和6 h时,表达量无显著变化(图8E);在GA处理下,PwPEBP表达量在处理3 h和12 h时下降,且12 h时显著低于0 h,表达量为0 h的0.36倍,处理6 h时表达量恢复到0 h的表达水平,是0 h的1.06倍,即PwPEBP的表达量呈现出先下降,再上升,随后又明显下降的表达模式(图8F)。在MeJA处理下,PwPEBP表达量在MeJA处理3 h时显著降低,为0 h的0.51倍,6 h时明显升高,是0 h的1.68倍,12 h时下降到0 h的1.12倍(图8G)。在SA处理条件下,PwPEBP表达量在处理3 h和6 h时持续下降,6 h时达到0 h的0.29倍,12 h时明显上升,达到0 h的1.45倍(图8H)。PwPEBP在根、茎、嫩叶、成熟叶、花粉和种子中均有表达,其中在成熟叶中表达量最高,是根中表达量的3.68倍;其次为种子,是根中的2.12倍;在嫩叶中表达量最低,仅为根中的0.01倍(图8I)。
![]() 图 8 PwPEBP在逆境、激素及各组织中的表达量A ~ H中各处理0 h作为对照,I中各组织中根作为对照,表达量为1,基因表达量均为相对表达量。Each treatment in A−H was used as a control for 0 h, and the root of each tissue in I was used as a control, the expression level was 1, and the gene expression amount was relative expression.Figure 8. Gene expression of PwPEBP under stress, hormones and tissues
图 8 PwPEBP在逆境、激素及各组织中的表达量A ~ H中各处理0 h作为对照,I中各组织中根作为对照,表达量为1,基因表达量均为相对表达量。Each treatment in A−H was used as a control for 0 h, and the root of each tissue in I was used as a control, the expression level was 1, and the gene expression amount was relative expression.Figure 8. Gene expression of PwPEBP under stress, hormones and tissues3. 讨 论
在植物中,PEBP 3大亚家族主要参与花期调控、种子萌发和休眠等过程[13]。本研究从青杄转录组测序结果中获得PwPEBP基因的cDNA序列。通过生物信息学分析发现,PwPEBP具有25个磷酸化位点,Corbit等[36]相关研究中表明,PEBP具有多个磷酸化位点,这与本研究中磷酸化位点预测结果相符合。通过进化树分析发现PwPEBP不属于植物PEBP蛋白3大亚家族FT、TFL和MFT的任何一类,PwPEBP与北美云杉中PEBP单独聚成一簇。多序列比对结果显示PwPEBP蛋白在C端多出一段特有的由20个氨基酸组成的序列。前期研究发现,在陆地棉的19个PEBP基因中,能将其分为GhFT、GhTFL、GhMTF及GhPEBP 4个亚组,GhPEBP为植物中新的PEBP家族基因,其属于细菌特有的基因类型[23]。所以本研究推测PwPEBP基因可能同属于植物中第4类新的PEBP基因。
PwPEBP在青杄不同组织中均有表达,在成熟叶片中表达量最高,其次是种子,在花粉中也有一定的表达,在当年新生叶中表达量最低,推测PwPEBP在青杄成熟叶中大量表达可能与其发挥抗逆功能有关。前期研究发现陆地棉中GhPEBP2启动子基因主要在子叶、茎和主根中大量表达,说明GhPEBP2基因的启动子主要在营养生长阶段启动该基因的特异表达,研究还发现在拟南芥中过量表达GhPEBP2基因,获得的4个转基因株系中,有2个株系开花时间提前,而另外2个株系开花时间却延迟,因此说明GhPEBP2基因在调控提前或延迟开花方面不够专一,不能稳定调控植物的开花时间[23]。根据本研究中PwPEBP在青杄种子和花粉中的表达结果,推测其可能参与了花粉和种子的发育与生长过程,这符合PEBP基因家族参与花期调控、种子萌发与休眠的特性。在已有研究中,尚未有PEBP亚家族参与种子生长过程的报道,而本研究发现,PwPEBP基因在种子中有较高的表达量,说明青杄中的PEBP亚家族基因可能参与种子的发育过程。
前人对植物中PEBP基因的研究多集中在调控开花和形态构成方面[26]。周小云[27]等研究发现,在新疆陆地棉中过表达雪莲的SiPEBP基因能提高其抗寒性,然而关于PEBP基因家族在逆境胁迫中的作用却研究甚少。本研究对PwPEBP启动子序列的响应原件预测发现,其含有响应低温的顺式作用元件LTR,还具有多个对光响应及与MYB和MYC结合的顺式作用元件,如I-box、G-box、MYB和MYC等,推测PwPEBP可能具有抗低温和提高光合作用的能力,并且其可能与MYB和MYC互作共同发挥作用,但具体机理尚不明确,有待进一步探究。通过启动子GUS染色结果证实该基因在低温、高温、干旱胁迫下均有响应,结果与PwPEBP启动子Luc活性分析结果一致,其中PEG处理的Luc/Ren比值为清水的5.6倍,活性最高。通过RT-qPCR实验进一步验证PwPEBP基因在干旱、低温及高温条件下,相对表达量均有显著变化。其中干旱诱导12 h时,响应最明显,表达量是0 h的28.41倍。说明PwPEBP在青杄生长过程中是一个能参与多种逆境响应的基因。
激素在植物参与防御反应及生长过程中发挥着至关重要的作用[37]。当植物遭遇逆境时,如干旱、盐碱、高温及低温等非生物胁迫,将会诱导植物体内ABA的积累,调节植物体内的代谢平衡从而增强植物的抗逆性[38]。在拟南芥中,外源施加ABA可使植物叶片的气孔开放抑制率高达90%以上,因而减少叶片水分的流失[39]。在早期研究中发现,GA3可作为一种与植物抗寒相关的激素,对水稻施加外源GA3能提高其抗寒性[40]。也有研究表明SA是植物抵抗逆境胁迫的关键信号分子[41]。本研究发现,PwPEBP启动子序列中包含了响应ABA、SA、MeJA、GA激素的顺式作用元件;在PwPEBP启动子的GUS染色及Luc定量实验中,证实了PwPEBP的启动子确实能够响应ABA、GA、MeJA和SA处理。RT-qPCR结果表明,PwPEBP在ABA、GA、SA及MeJA处理下,其表达量与未经处理的表达量相比,均有显著性变化。在GA处理下,PwPEBP表达量均下调;在MeJA处理下,PwPEBP表达量先下降后上升再下降;在SA处理下,PwPEBP表达量先持续下降后上升,本结果与SA处理豌豆叶片后LOX基因活性和JAs含量变化一致,也是呈现先下降后上升的趋势,在24 h时达到最高,显著高于未经SA处理的叶片[42]。在ABA处理下,PwPEBP的表达量呈现逐渐上调的趋势,与干旱处理的结果一致,在12 h时PwPEBP相对表达量是0 h的2.61倍,说明该基因参与了调控ABA的途径,并推测PwPEBP响应干旱途径可能是依赖ABA途径进行的。以上结果表明,PwPEBP参与了ABA、GA、MeJA和SA激素的信号通路,其价值仍需继续深入挖掘。
4. 结 论
本研究从青杄转录组中获得PwPEBP基因的cDNA序列,共1 408 bp,并从青杄基因组中克隆出PwPEBP的编码区序列,共585 bp,其编码194个氨基酸。PwPEBP蛋白属于PEBP家族中新的PEBP亚家族。PwPEBP在干旱、低温和高温下均有明显响应,但不被盐诱导。PwPEBP还参与了ABA、MeJA、SA及GA激素的信号通路。这些结果表明PwPEBP是一个能广泛响应非生物胁迫且参与多种激素信号通路的重要基因。
-
图 6 油松 SAUR 基因家族在干旱胁迫下的表达模式
Drought-8d、Drought-23d 分别为干旱处理8 d、23 d;CK-D8d、CK-D23d分别为对照处理8 d、23 d;Rewater-1d、Rewater-10d 分别为复水后1 d和10 d;CK-R1d、CK-R10分别为Rewater-1d、Rewater-10d的对照处理。下同。Drought-8d and Drought-23d are drought treatments for 8 and 23 d, respectively. CK-D8d and CK-D23d are control treatments for 8 and 23 d, respectively. Rewater-1d and Rewater-10d are 1 and 10 d after rehydration, respectively. CK-R1d and CK-R10 are control treatments for Rewater-1d and Rewater-10d, respectively. The same below.
Figure 6. Expression patterns of SAUR gene family in P. tabuliformis under drought stress
表 1 油松SAUR基因家族成员的序列特征和亚细胞定位预测
Table 1 Sequence characteristics and subcellular localization prediction of members of the SAUR gene family of P. tabuliformis
基因名称
Gene name基因号
Gene ID氨基酸长度
Amino acid
length/aa蛋白质分子量
Protein molecular
mass/kDa等电点
Isoelectric point (PI)亚细胞定位预测
Predicted subcellular localizationPtSAUR1 Pt0G04300 146 17.05 6.21 细胞核 Nucleus PtSAUR2 Pt0G10820 132 15.33 9.71 细胞核 Nucleus PtSAUR3 Pt0G13730 196 21.92 9.67 叶绿体 Chloroplast PtSAUR4 Pt0G14520 118 13.59 9.66 叶绿体/细胞质/线粒体/细胞核/过氧化物酶体
Chloroplast/cytoplasm/mitochondrion/nucleus/peroxisomePtSAUR5 Pt0G24890 154 17.63 6.49 细胞核 Nucleus PtSAUR6 Pt0G42350 162 18.97 5.97 细胞核 Nucleus PtSAUR7 Pt1G07530 114 13.01 9.87 叶绿体/线粒体Chloroplast/mitochondrion PtSAUR8 Pt1G07600 114 13.03 9.30 细胞核 Nucleus PtSAUR9 Pt1G07630 118 13.55 9.30 细胞核 Nucleus PtSAUR10 Pt1G08120 196 21.84 9.67 叶绿体 Chloroplast PtSAUR11 Pt1G08660 142 16.28 9.45 细胞核 Nucleus PtSAUR12 Pt1G08670 181 20.41 9.14 细胞核 Nucleus PtSAUR13 Pt1G08690 145 16.29 9.34 细胞核 Nucleus PtSAUR14 Pt1G08700 184 20.82 8.31 细胞核 Nucleus PtSAUR15 Pt1G08710 145 16.36 9.23 叶绿体/线粒体/细胞核 Chloroplast/mitochondrion/nucleus PtSAUR16 Pt1G08740 136 15.53 9.32 细胞膜/叶绿体/高尔基体/线粒体/细胞核
Cell membrane/chloroplast/Golgi apparatus/mitochondrion/nucleusPtSAUR17 Pt1G08750 128 14.60 9.30 叶绿体 Chloroplast PtSAUR18 Pt1G08870 139 15.85 8.99 细胞核 Nucleus PtSAUR19 Pt1G08900 142 16.21 9.35 叶绿体/线粒体/细胞核 Chloroplast/mitochondrion/nucleus PtSAUR20 Pt1G09080 142 16.33 9.32 细胞核 Nucleus PtSAUR21 Pt1G09110 144 16.27 9.13 细胞核 Nucleus PtSAUR22 Pt1G09150 117 13.05 9.30 细胞核 Nucleus PtSAUR23 Pt1G09200 147 16.69 7.72 细胞质 Cytoplasm PtSAUR24 Pt1G09260 152 17.56 9.42 叶绿体 Chloroplast PtSAUR25 Pt1G09740 154 17.46 9.10 叶绿体/细胞核 Chloroplast/nucleus PtSAUR26 Pt1G09750 152 17.27 9.14 线粒体/细胞核 Mitochondrion/nucleus PtSAUR27 Pt1G09760 149 17.14 9.51 叶绿体/细胞质/线粒体/细胞核/过氧化物酶体
Chloroplast/cytoplasm/mitochondrion/nucleus/peroxisomePtSAUR28 Pt2G24850 117 13.09 9.10 细胞核 Nucleus PtSAUR29 Pt2G25380 165 19.04 8.46 叶绿体/细胞核 Chloroplast/nucleus PtSAUR30 Pt2G26010 158 17.90 8.41 细胞核 Nucleus PtSAUR31 Pt3G09210 170 19.53 9.77 细胞核 Nucleus PtSAUR32 Pt3G29990 149 17.06 9.28 细胞核 Nucleus PtSAUR33 Pt3G64810 196 22.48 8.83 叶绿体 Chloroplast PtSAUR34 Pt3G68980 206 23.73 8.99 叶绿体/细胞核 Chloroplast/nucleus PtSAUR35 Pt3G72940 201 23.08 9.78 叶绿体/细胞质/线粒体 Chloroplast/cytoplasm/mitochondrion PtSAUR36 Pt3G72970 193 22.10 8.73 叶绿体/细胞核 Chloroplast/nucleus PtSAUR37 Pt4G13060 155 17.85 8.50 叶绿体 Chloroplast PtSAUR38 Pt4G13090 177 19.77 8.41 叶绿体/细胞核 Chloroplast/nucleus PtSAUR39 Pt4G15910 136 15.23 5.05 叶绿体 Chloroplast PtSAUR40 Pt5G16460 196 22.22 9.98 叶绿体 Chloroplast PtSAUR41 Pt5G43140 151 17.63 5.96 细胞核 Nucleus PtSAUR42 Pt5G43160 154 17.60 6.49 细胞核 Nucleus PtSAUR43 Pt5G43260 146 16.77 5.95 细胞核 Nucleus PtSAUR44 Pt5G43280 152 17.58 5.79 细胞核 Nucleus PtSAUR45 Pt5G43310 154 17.85 8.77 细胞核 Nucleus PtSAUR46 Pt5G43380 144 16.54 5.39 细胞核 Nucleus PtSAUR47 Pt5G43400 137 15.49 6.06 叶绿体 Chloroplast PtSAUR48 Pt6G17180 149 17.35 5.07 细胞核 Nucleus PtSAUR49 Pt6G42330 201 23.35 10.27 叶绿体/细胞核 Chloroplast/nucleus PtSAUR50 Pt6G42410 85 9.76 7.74 细胞核 Nucleus PtSAUR51 Pt6G42420 102 12.07 9.79 叶绿体 Chloroplast PtSAUR52 Pt9G21730 163 19.18 9.46 细胞膜/叶绿体/线粒体/过氧化物酶体
Cell membrane/chloroplast/mitochondrion/peroxisomePtSAUR53 PtJG06370 196 22.02 9.87 叶绿体 Chloroplast PtSAUR54 PtJG14220 172 19.36 9.4 叶绿体/细胞核 Chloroplast/nucleus PtSAUR55 PtJG14230 143 16.57 9.34 叶绿体/细胞核 Chloroplast/nucleus PtSAUR56 PtJG31250 146 16.61 8.73 叶绿体 Chloroplast PtSAUR57 PtJG31720 158 17.93 7.69 叶绿体/细胞质/细胞核 Chloroplast/cytoplasm/nucleus PtSAUR58 PtJG33080 165 18.96 10.36 叶绿体 Chloroplast PtSAUR59 PtJG33090 227 25.74 10.22 叶绿体/细胞核 Chloroplast/nucleus PtSAUR60 PtQG10070 141 15.92 6.48 叶绿体/细胞质/细胞核 Chloroplast/cytoplasm/nucleus PtSAUR61 PtQG18490 153 17.26 9.30 细胞核 Nucleus PtSAUR62 PtQG18510 158 18.00 8.33 细胞核 Nucleus PtSAUR63 PtQG18540 166 18.85 6.50 细胞核 Nucleus PtSAUR64 PtQG18560 121 13.88 6.21 细胞核 Nucleus PtSAUR65 PtQG18620 142 16.25 6.01 细胞核 Nucleus PtSAUR66 PtQG18660 137 15.79 6.42 细胞核 Nucleus -
[1] Li X, Liu G, Geng Y, et al. A genome-wide analysis of the small auxin-up RNA (SAUR) gene family in cotton[J]. BMC Genomics, 2017, 18(1): 815. doi: 10.1186/s12864-017-4224-2
[2] Hagen G, Guilfoyle T. Auxin-responsive gene expression: genes, promoters and regulatory factors[J]. Plant Molecular Biology, 2002, 49(3): 373−385.
[3] 李亚男, 冯霞, 陈大清. ARF、Aux/IAA和生长素受体对基因表达的调控[J]. 安徽农学通报, 2008, 14(7): 36−39. doi: 10.3969/j.issn.1007-7731.2008.07.013 Li Y N, Feng X, Chen D Q. Regulation of ARF, Aux/IAA and auxin receptor for gene expression[J]. Anhui Agricultural Science Bulletin, 2008, 14(7): 36−39. doi: 10.3969/j.issn.1007-7731.2008.07.013
[4] van Mourik H, van Dijk A D J, Stortenbeker N, et al. Divergent regulation of Arabidopsis SAUR genes: a focus on the SAUR10-clade[J]. BMC Plant Biology, 2017, 17(1): 245. doi: 10.1186/s12870-017-1210-4
[5] Guo Y, Jiang Q, Hu Z, et al. Function of the auxin-responsive gene TaSAUR75 under salt and drought stress[J]. The Crop Journal, 2018, 6(2): 181−190. doi: 10.1016/j.cj.2017.08.005
[6] Kong Y, Zhu Y, Gao C, et al. Tissue-specific expression of SMALL AUXIN UP RNA41 differentially regulates cell expansion and root meristem patterning in Arabidopsis[J]. Plant and Cell Physiology, 2013, 54(4): 609−621. doi: 10.1093/pcp/pct028
[7] Wong J H, Spartz A K, Park M Y, et al. Mutation of a conserved motif of PP2C. D phosphatases confers SAUR immunity and constitutive activity[J]. Plant Physiology, 2019, 181(1): 353−366. doi: 10.1104/pp.19.00496
[8] Zhang H, Yu Z, Yao X, et al. Genome-wide identification and characterization of small auxin-up RNA (SAUR) gene family in plants: evolution and expression profiles during normal growth and stress response[J]. BMC Plant Biology, 2021, 21(1): 4. doi: 10.1186/s12870-020-02781-x
[9] 周丽霞, 杨蒙迪, 曹红星, 等. 油棕SAUR基因家族的全基因组鉴定及生物信息学分析[J]. 南方农业学报, 2022, 53(4): 1011−1020. doi: 10.3969/j.issn.2095-1191.2022.04.014 Zhou L X, Yang M D, Cao H X, et al. Genome-wide identification and bioinformatics analysis of the oil palm SAUR gene family[J]. Journal of Southern Agriculture, 2022, 53(4): 1011−1020. doi: 10.3969/j.issn.2095-1191.2022.04.014
[10] Jain M, Tyagi A K, Khurana J P. Genome-wide analysis, evolutionary expansion, and expression of early auxin-responsive SAUR gene family in rice (Oryza sativa)[J]. Genomics, 2006, 88(3): 360−371. doi: 10.1016/j.ygeno.2006.04.008
[11] 李傲, 崔梦杰, 陈珂, 等. 葡萄SAUR基因家族鉴定与生物信息学分析[J]. 植物遗传资源学报, 2018, 19(2): 326−337. Li A, Cui M J, Chen K, et al. Identification and bioinformatics analysis of the SAUR gene family in grape[J]. Journal of Plant Genetic Resources, 2018, 19(2): 326−337.
[12] Zhang N, Huang X, Bao Y, et al. Genome-wide identification of SAUR genes in watermelon (Citrullus lanatus)[J]. Physiology and Molecular Biology of Plants, 2017, 23(3): 619−628. doi: 10.1007/s12298-017-0442-y
[13] Ren H, Gray W M. SAUR proteins as effectors of hormonal and environmental signals in plant growth[J]. Molecular Plant, 2015, 8(8): 1153−1164. doi: 10.1016/j.molp.2015.05.003
[14] Hu W, Yan H, Luo S, et al. Genome-wide analysis of poplar SAUR gene family and expression profiles under cold, polyethylene glycol and indole-3-acetic acid treatments[J]. Plant Physiology and Biochemistry, 2018, 128: 50−65. doi: 10.1016/j.plaphy.2018.04.021
[15] Stortenbeker N, Bemer M. The SAUR gene family: the plant’s toolbox for adaptation of growth and development[J]. Journal of Experimental Botany, 2019, 70(1): 17−27. doi: 10.1093/jxb/ery332
[16] Chae K, Isaacs C G, Reeves P H, et al. Arabidopsis SMALL AUXIN UP RNA63 promotes hypocotyl and stamen filament elongation[J]. The Plant Journal, 2012, 71(4): 684−697. doi: 10.1111/j.1365-313X.2012.05024.x
[17] Tian Z, Han J, Che G, et al. Genome-wide characterization and expression analysis of SAUR gene family in melon (Cucumis melo L.)[J]. Planta, 2022, 255(6): 123. doi: 10.1007/s00425-022-03908-0
[18] Hu J, Yu Q, Jiang S, et al. Identification and expression analysis of the small auxin-up RNA (SAUR) gene family in Lycium ruthenicum[J]. Peer Journal, 2023, 11: e15941.
[19] 刘昊东, 于亚新, 冯岗, 等. 小麦SAUR基因家族的鉴定及表达分析[J]. 分子植物育种, 2022, 20(14): 4525−4538. Liu H D, Yu Y X, Feng G, et al. Identification and expression analysis of the SAUR gene family in Triticum aestivum L.[J]. Molecular Plant Breeding, 2022, 20(14): 4525−4538.
[20] Rodríguez S M, Ordás R J, Alvarez J M. Conifer biotechnology: an overview[J]. Forests, 2022, 13(7): 1061.
[21] 张晶星, 马彦广, 王辉丽, 等. 油松JAZ基因家族特征及其与DELLA蛋白互作的功能域鉴定[J]. 北京林业大学学报, 2022, 44(12): 12−22. doi: 10.12171/j.1000-1522.20220027 Zhang J X, Ma Y G, Wang H L, et al. Characteristic of JAZ gene family of Pinus tabuliformis and identification of functional domain of its interaction with DELLA protein[J]. Journal of Beijing Forestry University, 2022, 44(12): 12−22. doi: 10.12171/j.1000-1522.20220027
[22] Li J, Han F, Yuan T, et al. The methylation landscape of giga-genome and the epigenetic timer of age in Chinese pine[J]. Nature Communications, 2023, 14(1): 1947.
[23] Pervaiz T, Liu S W, Uddin S, et al. The transcriptional landscape and hub genes associated with physiological responses to drought stress in Pinus tabuliformis[J]. International Journal of Molecular Sciences, 2021, 22(17): 9604. doi: 10.3390/ijms22179604
[24] Chen C, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant, 2020, 13(8): 1194−1202. doi: 10.1016/j.molp.2020.06.009
[25] Scott A D, Zimin A V, Puiu D, et al. A reference genome sequence for giant sequoia[J]. G3: Genes, Genomes, Genetics, 2020, 10(11): 3907−3919.
[26] Zhao Y P, Fan G, Yin P P, et al. Resequencing 545 ginkgo genomes across the world reveals the evolutionary history of the living fossil[J]. Nature Communications, 2019, 10(1): 4201. doi: 10.1038/s41467-019-12133-5
[27] Wang Y, Tang H, DeBarry J D, et al. MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity[J]. Nucleic Acids Research, 2012, 40(7): e49−e49. doi: 10.1093/nar/gkr1293
[28] Niu S, Li J, Bo W, et al. The Chinese pine genome and methylome unveil key features of conifer evolution[J]. Cell, 2022, 185(1): 204−217.
[29] Du Y L, Zhang Q, Li W J, et al. Genome- and transcriptome-wide identification and analysis of B3 superfamily members and their association with salt stress response in the common bean (Phaseolus vulgaris L.)[J]. Scientia Horticulturae, 2022, 305: 111408. doi: 10.1016/j.scienta.2022.111408
[30] 王福生, 余洪, 胡洲, 等. 柑橘属SAUR基因家族的全基因组鉴定及表达分析[J]. 园艺学报, 2020, 47(1): 23−40. Wang F S, Yu H, Hu Z, et al. Genome-wide analysis of SAUR gene family in Citrus[J]. Acta Horticulturae Sinica, 2020, 47(1): 23−40.
[31] Sun N, Wang J, Gao Z, et al. Arabidopsis SAURs are critical for differential light regulation of the development of various organs[J]. Proceedings of the National Academy of Sciences, 2016, 113(21): 6071−6076. doi: 10.1073/pnas.1604782113
[32] de la Torre A R, Piot A, Liu B, et al. Functional and morphological evolution in gymnosperms: a portrait of implicated gene families[J]. Evolutionary Applications, 2020, 13(1): 210−227. doi: 10.1111/eva.12839
[33] 王红飞, 尚庆茂. 黄瓜SAUR基因家族的鉴定与表达分析[J]. 园艺学报, 2019, 46(6): 1093−1111. Wang H F, Shang Q M. Genome-wide identification and expression analysis of the SAUR gene family in Cucumis sativus[J]. Acta Horticulturae Sinica, 2019, 46(6): 1093−1111.
[34] Kodaira K S, Qin F, Tran L S P, et al. Arabidopsis Cys2/His2 Zinc-Finger proteins AZF1 and AZF2 negatively regulate abscisic acid-repressive and auxin-inducible genes under abiotic stress conditions[J]. Plant Physiology, 2011, 157(2): 742−756. doi: 10.1104/pp.111.182683
[35] Ma X, Dai S, Qin N, et al. Genome-wide identification and expression analysis of the SAUR gene family in foxtail millet (Setaria italica L.)[J]. BMC Plant Biology, 2023, 23(1): 31. doi: 10.1186/s12870-023-04055-8
[36] Hassan S, Berk K, Aronsson H. Evolution and identification of DREB transcription factors in the wheat genome: modeling, docking and simulation of DREB proteins associated with salt stress[J]. Journal of Biomolecular Structure and Dynamics, Taylor & Francis, 2022, 40(16): 7191−7204.
[37] Yin C, Sun A, Zhou Y, et al. The dynamics of H2A. Z on SMALL AUXIN UP RNAs regulate abscisic acid-auxin signaling crosstalk in Arabidopsis[J]. Journal of Experimental Botany, 2023, 74(14): 4158−4168. doi: 10.1093/jxb/erad131



 下载:
下载: