Analysis of synonymous codon usage bias in the chloroplast genome of Davidia involucrata
-
摘要:目的
珙桐是蓝果树科珙桐属唯一现存的第三纪孑遗植物,是我国一级保护的特有濒危树种。分析其叶绿体基因组同义密码子使用偏好性及其主要影响因素,旨在为珙桐分子水平上的深入研究、物种保护和种质创新提供参考。
方法从NCBI在线数据库下载完整的珙桐叶绿体基因组序列,并进行蛋白编码序列筛选,再利用CodonW软件计算各基因的有效密码子数(ENC)、密码子适应指数(CAI)、同义密码子相对使用度(RSCU)和密码子中A、T、C、G这 4种碱基的含量,最后利用R软件计算各参数间的相关性并绘图。
结果(1)从珙桐叶绿体基因组中共筛选出59条蛋白编码序列,总的鸟嘌呤和胞嘧啶碱基(GC)平均含量和密码子第3位碱基的GC平均含量分别为38.05%和30.63%,CAI平均值为0.18,ENC平均值为48.52,表明珙桐叶绿体基因组基因表达水平较低,且密码子使用偏好性较弱。(2)ENC-plot、PR2-plot、中性绘图和对应性分析表明,影响珙桐叶绿体基因组同义密码子使用偏好性的最主要因素为选择压力。(3)共筛选出12个最优密码子。
结论珙桐叶绿体基因组同义密码子使用偏好性较弱,除了主要受选择压力影响外,还受到突变压力、碱基组成和基因表达水平等因素的影响;同时筛选出12个最优密码子,可用于未来珙桐的遗传改良和种质创新研究之中。
Abstract:ObjectiveDavidia involucrata is the only existing tertiary relict plant of genus Davidia in the family Nyssaceae. It is a unique endangered tree species under first-class protection in China. The synonymous codon usage bias (SCUB) of chloroplast genome and its main influencing factors were analyzed to provide a reference for further research at the molecular level, species conservation and germplasm innovation of the D. involucrata.
MethodThe complete chloroplast genome of D. involucrata was downloaded from the NCBI online database and the protein-coding sequences were screened. The effective number of codon (ENC), codon adaptation index (CAI), the relative synonymous codon usage (RSCU), and the contents of four bases, A, T, C, and G in the codon were calculated by CodonW software. Finally, the correlation between the parameters were calculated and the plots were maken using R software.
Result(1) A total of 59 protein-coding sequences were screened from the chloroplast genome of D. involucrata. The average guanine and cytosine bases (GC) content and the average GC content of the third base of the codons were 38.33% and 30.68%, respectively. The average CAI was 0.16 and the average ENC was 46.55, indicating that the chloroplast genome gene expression level of D. involucrata was low and the codon use bias was weak. (2) Enc-plot, PR2-plot, neutral plot and correspondence analysis revealed that selection pressure was the most important factor affecting SCUB of D. involucrata chloroplast genome. (3) A total of 12 optimal codons were selected.
ConclusionThe SCUB of chloroplast genome in D. involucrata is weak, and its SCUB is not only mainly affected by selection pressure, but also affected by factors such as mutation pressure, base composition and gene expression level. Meanwhile, a total of 12 optimal codons are selected, which can be used in future genetic improvement and germplasm innovation research of D. involucrate.
-
Keywords:
- Davidia involucrata /
- synonymous codon usage bias /
- optimal codon /
- chloroplast genome /
- amino acid
-
密码子是mRNA翻译的基本信息单位,除甲硫氨酸和色氨酸仅由1个密码子编码外,其余氨基酸均由多个同义密码子编码[1]。同义密码子在翻译过程中并非随机使用,而是具有偏好性,即同义密码子偏好性(synonymous codon usage bias, SCUB) [2]。SCUB以非随机方式广泛存在于多种生物中,反映了生物适应环境过程中的一些进化事件[3]。SCUB主要由自然选择和突变压力所引起,此外还受到基因表达水平、tRNA丰度、蛋白质长度、基因翻译起始信号、蛋白质结构、鸟嘌呤和胞嘧啶碱基(guanine and cytosine bases,GC)含量、突变频率与模式、以及随机遗传漂移等因素的影响[2,4−7]。因此,同义密码子使用偏好性分析有助于新基因发现、基因功能预测、转基因设计以及对生物分子进化和环境适应的理解[8]。
叶绿体在植物光合作用和代谢物生物合成过程中起着重要作用[9]。与核基因相比,叶绿体基因转化具有外源基因表达效率高、定点整合无位置效应、遗传稳定、不随花粉漂移等优点,现已被广泛用于分子进化、系统发育和遗传表达等研究领域[10]。随着高通量测序技术的不断发展,目前关于叶绿体基因组密码子偏好性的研究越来越多,如巨桉(Eucalyptus grandis)[11]、金莲花(Trollius chinensis)[12]、紫菜(Porphyra umbilicalis)[13]、桔梗(Delphinium grandiflorum)[14]、大戟科(Euphorbiaceae)植物[15]和水稻(Oryza sativa) [16]等,极大地促进了这些物种的种质创新和开发应用。
珙桐(Davidia involucrata)是蓝果树科(Nyssaceae)珙桐属唯一现存的第三纪孑遗植物,也是我国特有的濒危树种,被称为“活化石”[17−18]。珙桐具有较高的研究价值、观赏价值和药用价值。自1869年被发现以来,科学家们在其分类学、形态学、生理学、生态学、繁殖育种等方面都进行了大量研究[19−22]。然而,在分子水平上的研究却进展缓慢[23],限制了该物种的深入研究。本研究通过生物信息学方法分析珙桐叶绿体基因组的同义密码子偏好性,旨在为珙桐的遗传进化、系统发育、分子育种和物种保护等方面的进一步研究提供参考。
1. 材料与方法
1.1 基因序列获取
从NCBI GenBank下载珙桐叶绿体基因组(登录号:KR061358.1)。序列全长为169 085 bp,包含82个蛋白编码基因。参照Duan等[14]的方法剔除长度小于300 bp的编码序列,并选择以ATG为起始密码子、以TAA、TAG和TGA为终止密码子的编码序列,最终筛选出59条序列进行后续分析研究。
1.2 密码子使用指数
使用在线工具(http://112.86.217.82:9919/#/tool/alltool/detail/214)计算珙桐叶绿体基因组的SCUB相关指标,包括基因GC含量(GCall)、密码子第1、2、3位GC含量(GC1、GC2、GC3)、同义密码子相对使用度(relative synonymous codon usage, RSCU)、密码子适应指数(codon adaptation index,CAI)和有效密码子数(effective number of codon,ENC)[14−15]。RSCU值表示编码氨基酸的所有密码子的平均使用次数,RSCU = 1时,表示同义密码子无偏好;RSCU > 1时,表示同义密码子偏好性强;而RSCU < 1时,则表示偏好性较弱[24]。CAI是指编码区同义密码子与最佳密码子使用频率的相符程度,用于评估外源基因在宿主内的表达水平,取值在0 ~ 1之间,CAI越高,表示外源基因在宿主内的表达水平越高。ENC常被用来衡量单个基因的密码子偏好性大小,取值范围在20 ~ 61之间。当ENC ≤ 35时,则被认为该基因具有强密码子偏好性;当ENC = 61时,则被认为密码子无偏好性,其标准计算公式为[6]
ENC=2+t+29t2+(1−t2)2 式中: ENC为有效密码子数,t为密码子第3位GC含量 (GC3)。
1.3 中性绘图分析
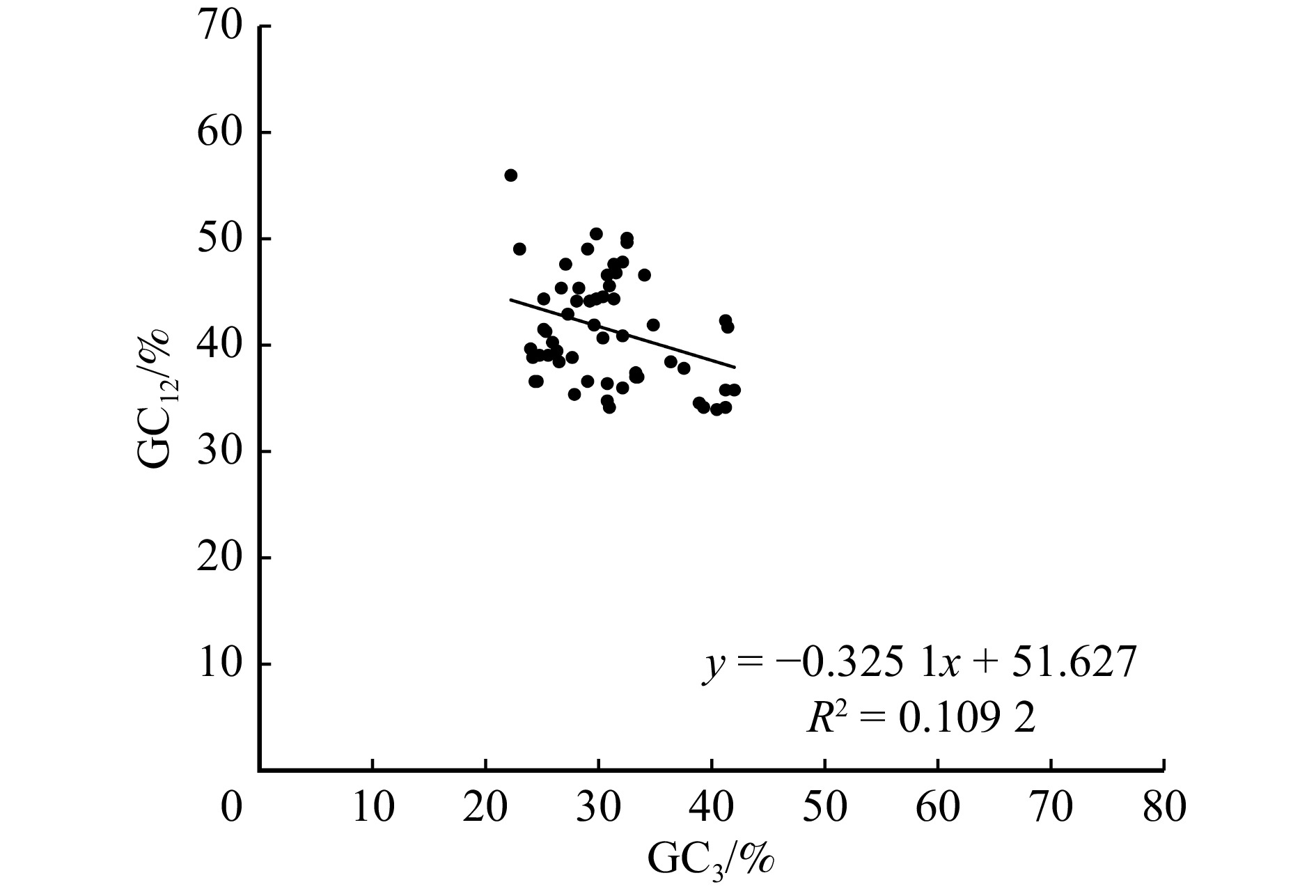
中性绘图是以GC1和GC2的均值(GC12)为纵坐标,以GC3为横坐标绘制的散点图,图中每1个点代表1个基因的位置。根据回归线的斜率和GC含量的分布,可以推断出突变压力和选择压力的作用。当回归线的斜率接近1时,突变压力被认为起着主要作用,而当回归线的斜率接近0时,则说明GC12和GC3之间缺乏相关性,表明选择压力起主要作用[25]。
表 1 珙桐叶绿体基因组密码子GC含量及CAI和ENC值Table 1. GC proportion, CAI and ENC in chloroplast genomics of Davidia involucrata基因
GeneCAI ENC GC含量 GC content 基因
GeneCAI ENC GC含量 GC content GCall/% GC1/% GC2/% GC3/% GCall/% GC1/% GC2/% GC3/% rps8 0.08 37.04 36.13 39.86 43.36 25.17 ycf2 0.15 48.53 37.78 37.08 35.00 41.25 rps18 0.11 37.57 35.29 35.29 44.12 26.47 ndhK 0.16 48.55 38.05 44.25 44.69 25.22 rps14 0.11 38.09 41.91 44.55 49.50 31.68 ycf4 0.16 48.82 38.10 42.33 39.68 32.28 ndhE 0.16 40.41 32.68 38.24 35.29 24.51 rpoB 0.15 48.85 38.94 50.89 37.72 28.20 psbA 0.32 40.82 42.66 50.28 43.50 34.18 psbB 0.20 48.86 44.47 54.81 45.97 32.61 ndhF 0.15 43.72 32.71 37.72 35.97 24.43 rpoA 0.16 49.64 34.62 44.38 32.84 26.63 ndhF 0.15 43.72 32.71 37.63 36.16 24.60 rps4 0.14 49.84 39.27 51.98 39.11 26.73 rps3 0.16 43.87 35.62 47.49 33.33 26.03 rps2 0.18 49.95 39.66 44.73 44.30 29.96 psbD 0.25 44.42 42.28 52.26 43.22 31.36 rpoC2 0.15 50.04 37.97 45.82 38.50 29.58 rps7 0.19 45.03 40.60 53.21 45.51 23.08 matK 0.15 50.13 34.19 41.39 32.08 29.11 rps7 0.19 45.03 40.60 53.21 45.51 23.08 atpE 0.16 51.02 39.80 51.49 39.55 28.36 rpl23 0.13 45.75 37.39 40.00 41.74 30.43 ndhH 0.16 51.13 39.26 51.52 37.06 29.19 ndhG 0.13 45.96 34.09 44.07 33.90 24.29 ycf2 0.21 51.22 37.78 36.70 32.11 39.45 atpB 0.21 46.12 42.48 56.31 42.08 29.06 ndhA 0.14 51.26 33.17 36.42 32.11 30.98 ndhD 0.14 46.24 35.29 41.37 36.86 27.65 clpP 0.16 51.31 36.21 37.28 38.06 33.29 psbC 0.18 46.25 44.02 53.38 46.20 32.49 ndhJ 0.15 51.37 40.25 51.57 37.74 31.45 ndhC 0.20 46.41 34.44 45.45 33.06 24.79 ndhB 0.17 51.78 37.84 36.60 39.33 37.58 rpl22 0.20 46.70 34.60 43.67 36.08 24.05 ndhB 0.16 52.51 37.84 35.82 41.33 36.40 rps11 0.15 46.75 44.84 54.68 57.55 22.30 petA 0.18 53.14 39.98 52.34 37.07 30.53 cemA 0.20 46.78 33.62 40.87 29.13 30.87 ycf3 0.15 53.19 35.92 34.78 39.49 33.48 rpl14 0.18 46.80 41.46 56.10 37.40 30.89 petD 0.16 53.20 36.72 38.41 30.44 41.31 atpF 0.13 46.85 36.25 33.88 34.30 40.58 rpl16 0.13 54.37 34.66 38.16 35.01 30.82 accD 0.19 46.90 33.04 36.01 35.12 27.98 rpoC1 0.15 56.05 37.99 38.82 33.12 42.03 atpI 0.18 47.17 37.90 48.39 37.90 27.42 rps16 0.16 56.33 34.92 40.63 31.93 32.19 rpl20 0.09 47.55 36.16 38.98 44.07 25.42 rps12 0.15 56.58 39.74 44.21 40.14 34.86 atpA 0.20 47.60 40.94 55.12 40.55 27.17 petB 0.15 57.40 36.23 38.52 31.15 39.01 psaB 0.18 47.71 40.91 48.71 42.99 31.02 rpl2 0.15 58.12 42.08 39.32 45.64 41.29 rbcL 0.28 47.86 43.77 57.77 43.70 29.83 rpl2 0.14 58.78 42.08 45.27 38.63 41.53 psaA 0.20 48.40 42.79 52.73 43.41 32.22 rpl23 0.23 48.68 37.39 41.03 33.33 33.33 ndhI 0.19 48.52 34.72 42.26 36.31 25.60 平均值 Mean 0.18 48.52 38.05 44.51 38.83 30.63 注:CAI. 密码子适应指数; ENC. 有效密码子数; GCall . 基因中所有密码子的的GC含量;GC1 . 基因中所有密码子的第1位的GC含量;GC2. 基因中所有密码子的第2位的GC含量;GC3. 基因中所有密码子的第3位的GC含量。Notes: CAI, codon adaptation index; ENC, effective number of codon; GCall, GC content of all codons in the gene; GC1, the GC content in position 1 of all codons in the gene; GC2, the GC content in position 2 of all codons in the gene; GC3, the GC content in position 3 of all codons in the gene. 1.4 ENC-plot绘图分析
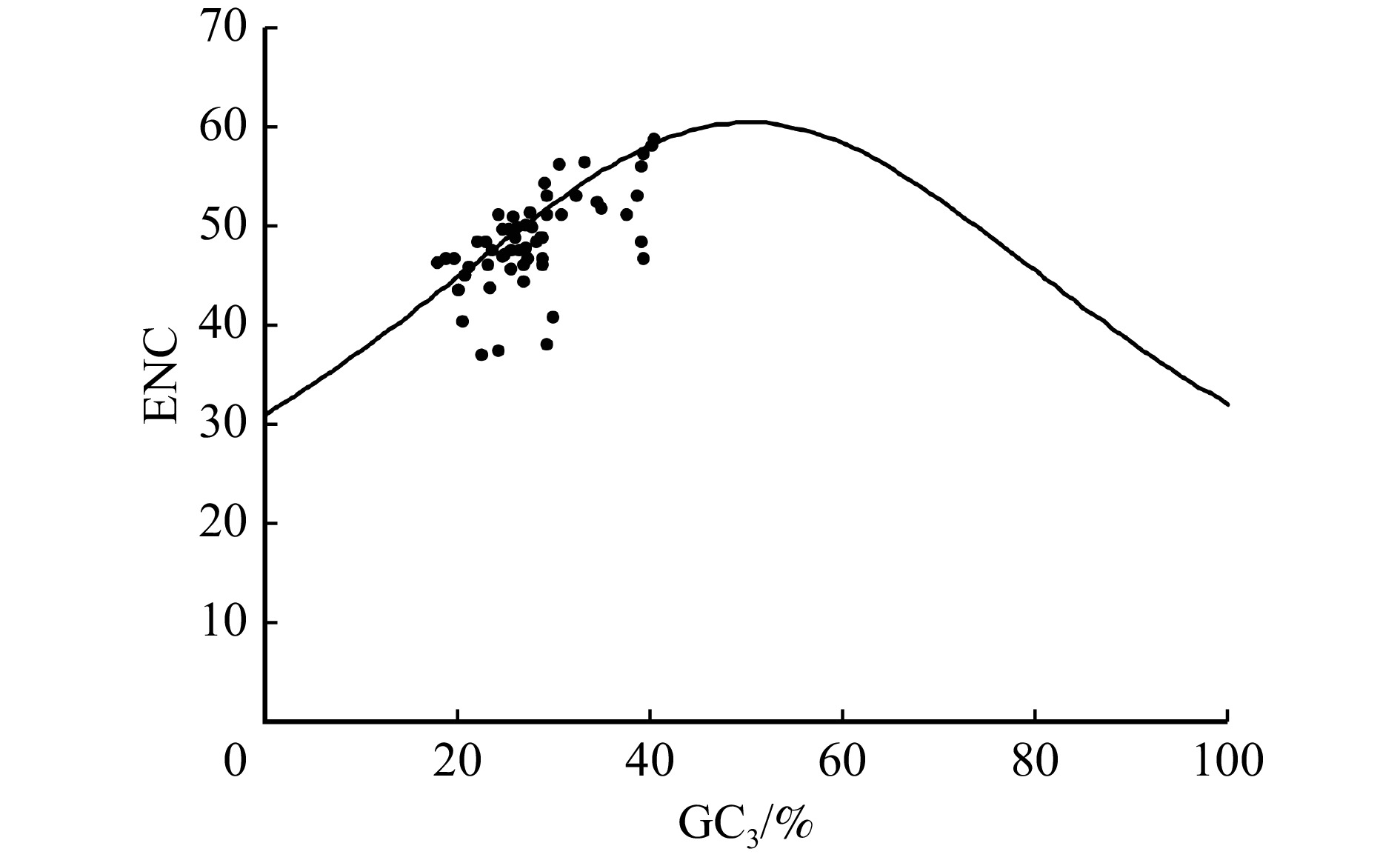
为了探究ENC与基因碱基组成之间的关系,以GC3为横坐标,以ENC为纵坐标绘制散点图,并在散点图中绘制标准ENC值的曲线[26]。当基因ENC值在标准曲线附近时,表明基因密码子使用偏好受突变压力的影响较大;而当ENC值偏离标准曲线较远时,表明密码子使用模式受选择压力的影响较大。以ENC检测值(ENCobs)和ENC标准值(ENCexp)两者的差值与ENC标准值的比值作为各基因有效密码子比值,并进行珙桐叶绿体基因组有效密码子比值频率分布分析。
1.5 PR2-plot偏倚分析
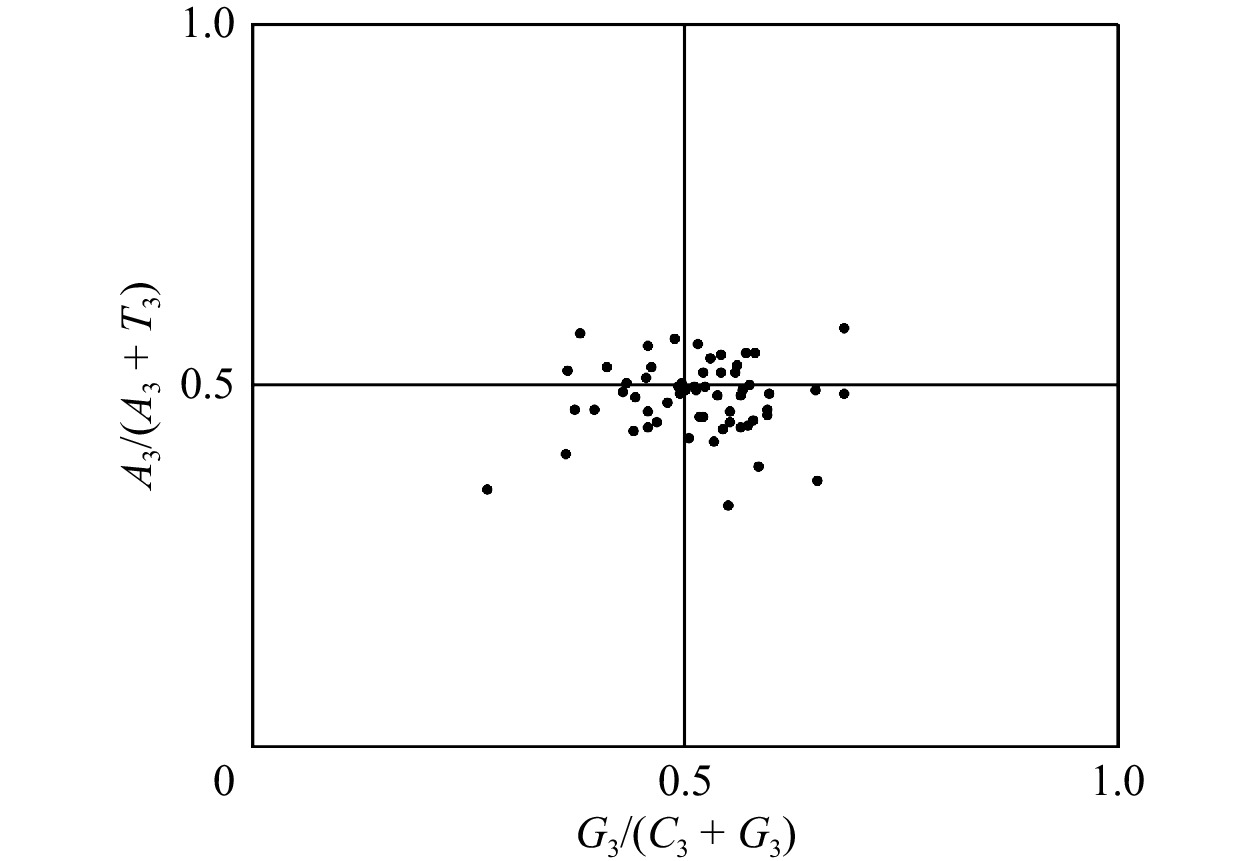
PR2-plot偏倚分析用于探求突变压力和选择压力对SCUB的影响。一般认为,在单个突变压力下,基因或基因组的简并密码子中A/T和C/G的比例是平衡的[27]。分别计算密码子第3位碱基上A、T、C、G的含量(A3、T3、C3、G3),以G3/(G3 + C3)为横坐标,A3/(A3 + T3)为纵坐标进行偏倚分析[14]。
1.6 对应性分析
基于同义密码子相对使用度值进行对应分析,比较59个密码子(不包括甲硫氨酸、色氨酸密码子和3个终止密码子)的使用模式,得到一系列正交坐标轴,可用于显示叶绿体基因组密码子使用模式的变化。依据基因同义密码子在59个轴空间分布情况,可以得出各基因的分布情况及基因变异的最大比例,从而分析密码子使用变异的主要来源[14]。
1.7 最优密码子分析
最优密码子的确定参考杨祥燕等[28]的分析方法,以CAI值作为高表达和低表达基因的参考依据。首先,将所有基因按照CAI值进行排序,然后从排序总基因前后端各取10%的基因作为高、低表达样本组。随后计算高、低表达组的平均同义密码子相对使用度,以RSCU > 1的密码子为高频密码子,高表达组与低表达组RSCU差(ΔRSCU) > 0.08的为高表达密码子,ΔRSCU > 0.08且RSCU > 1的密码子判定为最优密码子。
2. 结果与分析
2.1 珙桐叶绿体密码子组成特征
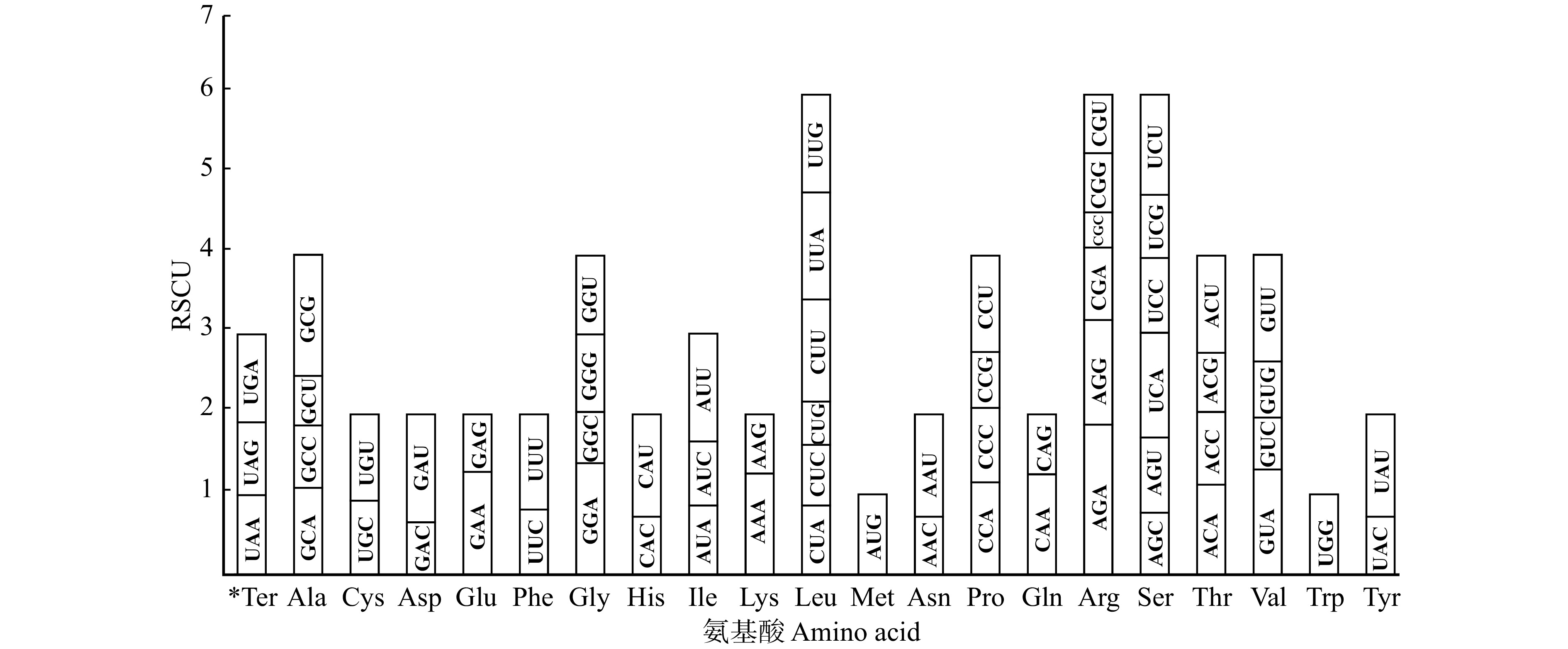
利用perl脚本除去长度小于300 bp的叶绿体基因,共获得59个独特基因,包括28个光合作用基因(psa、 psb、 pet、 atp、 ndh、 rbcL)、18个核糖体基因(rps、 rpl)、4个遗传系统基因(rpo)、4个保守基因(ycf)及5个其他基因(matK、 accD、 cemA、 clpP),这些基因全长85 229 bp,占基因组全长的54.1%,长度范围在303 ~ 1 070 bp之间,平均长度为386 bp。对其进行密码子偏好性指标分析(表1)发现:叶绿体基因平均GC含量为38.05%,其中GC1(44.51%) > GC2(38.83%) > GC3(30.63%),ENC值范围在37.04 ~ 58.78之间,平均值为48.52;CAI值范围在0.08 ~ 0.32之间,平均值为0.18。以上结果表明,珙桐叶绿体基因组的第3个位置上的GC含量明显低于前2个位置,偏好使用碱基A或U,基因表达水平较低,且密码子使用偏好性较弱。RSCU分析(图1)显示:高频密码子(RSCU > 1)共有27个,其中13个以U结尾,12个以A结尾,2个以G结尾,以U和A结尾的密码子占92.59%。说明珙桐叶绿体基因组密码子的使用更倾向于使用以U或A结尾的同义密码子。
2.2 中性绘图分析
图2显示:GC12(GC1和GC2的平均值)的取值范围在34.09% ~ 56.12%之间,GC3的取值范围在17.3% ~ 42.7%之间,GC12和GC3的相关系数和斜率分别为0.109 2和−0.325 1,斜率接近0。说明珙桐叶绿体基因组基因密码子的3个碱基在组成上存在较大差异,自然选择对珙桐叶绿体基因组密码子使用偏好的影响较大。
2.3 ENC-plot分析
通过构建ENC-plot图分析造成密码子使用偏好的主要影响因素,发现大部分基因都聚集在ENC标准曲线附近,说明大多数基因的ENC检测值(ENCobs)与ENC标准值(ENCexp)很接近(图3)。通过计算ENC比值,来进一步量化ENCobs与ENCexp之间的差异。结果(表2)显示:35个基因(59.32%)的ENC比值位于−0.05 ~ 0.05区间,表明ENCobs值与ENCexp值略有差异,意味着选择压力并不是影响珙桐密码子使用偏好性的唯一因素,突变压力也是偏好性的重要影响因素之一。
表 2 ENC比值的频率分布Table 2. Frequency distribution of ENC ratio组限
Group value range组中值
Group mid-value组数
Group number频率
Frequency/%−0.25 ~ 0.15 0.2 6 10.17 −0.15 ~ 0.05 0.1 12 20.34 −0.05 ~ 0.05 0 35 59.32 0.05 ~ 0.15 0.1 6 8.47 2.4 PR2-plot偏倚分析
PR2-plot分析结果(图4)显示:氨基酸密码子第3位碱基分布并不均匀,38个基因的A3/(A3 + T3)小于0.5,35个基因的G3/(C3 + G3)大于0.5,说明在总体上,基因组密码子中A与T的比例接近,G与 C的比例接近。然而,从图4也可以看出,很多基因并非聚集在平面图中心,而是分布在距离中心点较远的位置,大部分位于平面图的下半部和右半部,表明密码子第3位碱基嘧啶碱(T和C)的使用频率高于嘌呤碱(A和G),意味着突变压力不是珙桐叶绿体基因组的同义密码子使用偏好的唯一影响因素,其他因素对珙桐叶绿体基因组的同义密码子使用偏好也产生一定影响[11]。
![]() 图 4 珙桐叶绿体基因组PR2-plot绘图分析A3. 密码子第3位A碱基的含量;T3. 密码子第3位T碱基的含量;G3. 密码子第3位G碱基的含量;C3. 密码子第3位C碱基的含量。A3, adenine (A) content at the third position of codon; T3, thymine (T) content at the third position of codon; G3, guanine (G) content at the third position of codon; C3, cytosine (C) content at the third position of codon.Figure 4. Analysis of PR2-plot of chloroplast genomes of D. involucrata
图 4 珙桐叶绿体基因组PR2-plot绘图分析A3. 密码子第3位A碱基的含量;T3. 密码子第3位T碱基的含量;G3. 密码子第3位G碱基的含量;C3. 密码子第3位C碱基的含量。A3, adenine (A) content at the third position of codon; T3, thymine (T) content at the third position of codon; G3, guanine (G) content at the third position of codon; C3, cytosine (C) content at the third position of codon.Figure 4. Analysis of PR2-plot of chloroplast genomes of D. involucrata2.5 对应性分析
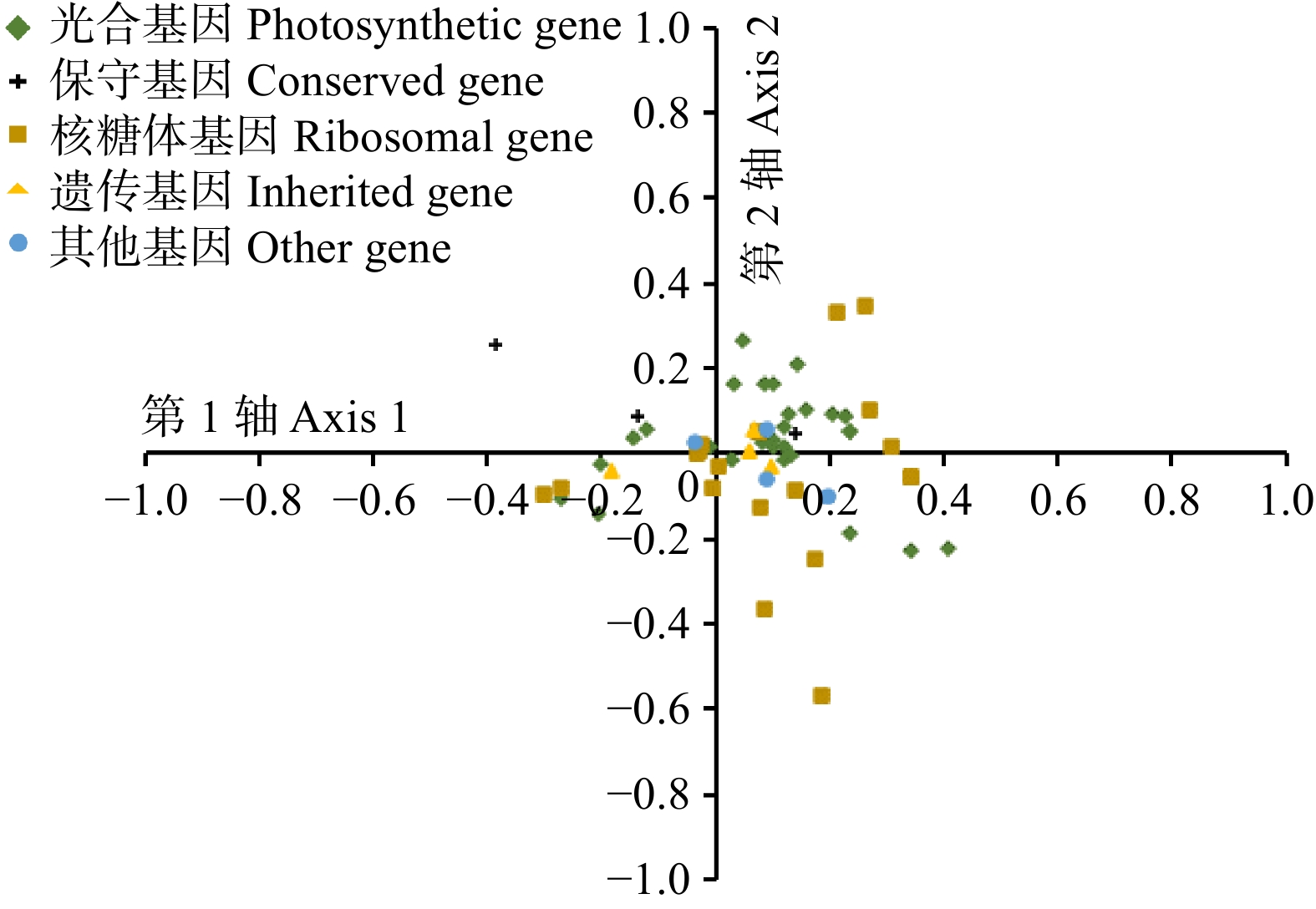
对珙桐所有基因的SCUB的参数进行对应性分析。结果表明:前4个轴占总贡献率的50.20%,第1、2、3和4轴(Axis 1、2、3和4)贡献率分别为18.64%、11.58%、10.34%和10.64%,说明Axis 1是密码子使用偏好性的主要变异来源。以Axis 1为横坐标,Axis 2为纵坐标,将5类基因分布于平面,发现光合基因、遗传基因和其他基因分布相对集中,而核糖体和保守基因较为分散(图5),说明核糖体蛋白和保守基因的密码子使用偏好性相差较大,而其他基因的密码子使用偏好性相差较小。为确定密码子使用偏好性的影响因素,分别计算CAI、ENC、GC3、GC12与Axis 1之间的相关系数,结果(表3)显示:Axis 1与ENC(r = −0.685 0,P < 0.01)、GC3 (r = −0.927 0,P < 0.01)和CAI(r = −0.139 0, P < 0.05)均显著或极显著负相关,与GC12极显著正相关(r = 0.406 0,P < 0.01),表明珙桐叶绿体基因组基因密码子使用模式除了受突变压力的影响外,还受选择压力的影响。
表 3 珙桐叶绿体基因组密码子偏好指标相关性分析Table 3. Correlation analysis of codon usage index of chloroplast genomes of D. involucrata参数
ParameterAxis 1 ENC CAI GC12 GC3 ENC −0.685 0** CAI 0.139 0* −0.065 5** GC12 0.406 0** −0.240 0 0.361 0** GC3 −0.927 0** 0.625 0** −0.040 4 −0.330 0 GCall −0.133 0 0.137 0 0.356 0** 0.817 0** 0.263 0* 注:*. 显著相关(P < 0.05);**. 极显著相关(P < 0.01)。Notes: *, a significant correlation at P < 0.05 level; **, a highly significant correlation at P < 0.01 level. 密码子使用指数之间的相关性分析(表3)表明:CAI分别与GCall (r = 0.356 0,P < 0.01)、GC12(r = 0.361 0,P < 0.01)极显著正相关,与ENC(r = −0.065 5, P < 0.01)极显著负相关,其中CAI值是基因表达水平的重要指标,说明基因表达水平除了主要受碱基的影响外,也受到密码子偏好性的显著影响。ENC仅与GC3极显著正相关(r = 0.625 0,P < 0.01),表明密码子第3位碱基组成对密码子偏好性影响较大;而碱基之间的相关分析(表3)表明,GCall分别与GC12、GC3呈极显著(r = 0.817 0, P < 0.01)和显著正相关(r = 0.263 0, P < 0.05)。
2.6 最优密码子分析
以RSCU > 1为标准共筛选出37个高频密码子,以ΔRSCU > 0.08为标准共筛选出27个高表达密码子(表4)。其中,高表达密码子以A或T结尾的共有21个,以C或G结尾的共有6个。以RSCU > 1且ΔRSCU > 0.08为标准共筛选出12个最优密码子,分别为UUA、AUU、GUU、GUA、GCU、UAU、UAA、CAU、AAU、AAA、GAU和UGU,所有最优密码子均以A或U结尾(表4),表明密码子更倾向于使用A或U结尾,这与前面RSCU分析的结果(图1)相一致。
表 4 珙桐叶绿体基因组的最优密码子Table 4. Optimal codons in chloroplast genome of D. involucrata氨基酸
Amino acid密码子
Codon高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 氨基酸
Amino Acid密码子
Codon高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 数目
NumberRSCU 数目
NumberRSCU 数目
NumberRSCU 数目
NumberRSCU 亮氨酸 Leu UUA** 54 1.59 26 1.28 0.31 酪氨酸 Tyr UAU** 50 1.39 25 1.04 0.35 UUG 45 1.32 30 1.48 −0.16 UAC 22 0.61 23 0.96 −0.35 CUU*** 41 1.21 12 0.59 0.62 *Ter UAA*** 5 1.88 32 1.16 0.72 CUC 17 0.5 19 0.93 −0.43 UGA 2 0.75 31 1.12 −0.37 CUA 32 0.94 24 1.18 −0.24 UAG 1 0.38 20 0.72 −0.34 CUG 15 0.44 11 0.54 −0.1 组氨酸 His CAU* 34 1.36 14 1.27 0.09 异亮氨酸 Ile AUU** 74 1.52 40 1.19 0.33 CAC 16 0.64 8 0.73 −0.09 AUC** 49 1.01 23 0.68 0.33 谷氨酰胺 Gln CAA 46 1.44 26 1.44 0 AUA 23 0.47 38 1.13 −0.66 CAG 18 0.56 10 0.56 0 甲硫氨酸 met AUG 58 1 21 1 0 天冬酰胺 Asn AAU* 66 1.28 25 1.02 0.26 缬氨酸 Val GUU* 49 1.39 19 1.21 0.18 AAC 37 0.72 24 0.98 −0.26 GUC 13 0.37 13 0.83 −0.46 赖氨酸 Lys AAA* 60 1.46 46 1.23 0.23 GUA*** 66 1.87 18 1.14 0.73 AAG 22 0.54 29 0.77 −0.23 GUG 13 0.37 13 0.83 −0.46 天冬氨酸 Asp GAU** 59 1.37 20 1.05 0.32 丝氨酸 Ser UCU*** 48 2.03 8 0.98 1.05 GAC 27 0.63 18 0.95 −0.32 UCC* 23 0.97 7 0.86 0.11 谷氨酸 Glu GAA*** 99 1.56 37 0.94 0.62 UCA* 25 1.06 8 0.98 0.08 GAG 28 0.44 42 1.06 −0.62 UCG 7 0.3 6 0.73 −0.43 半胱氨酸 Cys UGU* 12 1.2 10 1.11 0.09 AGU 26 1.1 11 1.35 −0.25 UGC 8 0.8 8 0.89 −0.09 AGC 13 0.55 9 1.1 −0.55 色氨酸 Trp UGG 41 1 25 1 0 脯氨酸 Pro CCU*** 52 2.12 9 0.97 1.15 精氨酸 Arg CGU*** 45 2.21 4 0.36 1.85 CCC 14 0.57 5 0.54 0.03 CGC** 12 0.59 1 0.09 0.5 CCA 19 0.78 16 1.73 −0.95 CGA** 23 1.13 8 0.72 0.41 CCG 13 0.53 7 0.76 −0.23 CGG 5 0.25 2 0.18 0.07 苏氨酸 Thr ACU*** 63 2 5 0.67 1.33 AGA 32 1.57 47 4.21 −2.64 ACC* 24 0.76 4 0.53 0.23 AGG 5 0.25 5 0.45 −0.2 ACA 28 0.89 15 2 −1.11 苯丙氨酸 Phe UUU 68 0.96 47 1.12 −0.16 ACG 11 0.35 6 0.8 −0.45 UUC* 74 1.04 37 0.88 0.16 丙氨酸 Ala GCU*** 92 2.19 5 1.25 0.94 甘氨酸 Gly GGU*** 87 1.87 7 0.46 1.41 GCC 20 0.48 3 0.75 −0.27 GGC 17 0.37 8 0.52 −0.15 GCA 40 0.95 4 1 −0.05 GGA 57 1.23 29 1.9 −0.67 GCG 16 0.38 4 1 −0.62 GGG 25 0.54 17 1.11 −0.57 注:加下划线的密码子的RSCU > 1;*代表0.08 < ΔRSCU < 0.3;**代表0.3 < ΔRSCU < 0.5;***代表ΔRSCU > 0.5,加粗密码子被认为是最优密码子。Notes: the underlined codon,RSCU > 1; *, 0.08 < ΔRSCU < 0.3; **, 0.3 < ΔRSCU < 0.5; ***, ΔRSCU > 0.5; the bold codons are thoght to be the optimal codons. 3. 讨 论
植物叶绿体和线粒体基因组的同义密码子使用偏好性与核基因的同义密码子使用偏好性在进化速率和模式上有所不同,植物叶绿体和线粒体基因组的同义密码子使用偏好性除了主要受DNA序列的定向突变压力和选择压力影响外[29],还受tRNA丰度、链特异突变偏倚、基因表达水平和基因长度等因素的影响[2,4−7],这些影响因素被广泛地用于解释种间密码子使用变异和基因组内密码子使用变异情况[8]。其中,叶绿体基因组的GC含量是与突变压力平衡适应的结果,这是密码子使用偏好性形成过程中最普遍的影响因素之一[14]。而密码子第3位碱基的同义突变虽然不能改变氨基酸的类型,但它仍然被认为是决定氨基酸类型的重要特征,因此GC3经常被用作密码子使用偏好性的重要指标[30]。当密码子的使用主要受选择压力的影响时,GC3值往往分布在一个较小的范围内,且GC12和GC3之间没有显著的相关性[31]。在本研究中,GC3含量平均值为30.63%,低于于水稻(Oryza sativa)[16]、玉米(Zea mays)[29]、小麦(Triticum aestivum) [32]等单子叶农作物[33]。这或许是因为:单子叶植物与双子叶植物进化分离后,单子叶植物叶绿体基因组中编码序列的GC含量有普遍增加的趋势[29]。探究突变压力和选择压力对植物进化及育种的影响具有重要意义,中性绘图分析发现GC12和GC3之间并不存在显著相关性,说明珙桐叶绿体基因组的同义密码子使用偏好性主要受选择压力的影响,这与大部分被子植物的研究结果[15,28,34−36]相一致,说明在长期进化过程中植物叶绿体基因组同义密码子使用偏好性相对保守且高度相似。
通过ENC-plot分析叶绿体基因组中同义密码子使用偏好性是否受到突变压力的影响,显示大多数基因位于标准曲线附近,只有少数基因偏离预期曲线较远(图3),表明除了选择压力外,突变压力也参与了某些基因的同义密码子使用偏好性。然而,仅凭ENC-plot无法单独区分同义密码子使用偏好性主要受到突变压力还是选择压力的影响。在本研究中,通过结合中性绘图和PR2-plot分析,发现G、C和A、T碱基对在密码子第3个位置上的使用并不均等(图4)。根据Frank[37]的观点,如果突变压力是同义密码子使用偏好性的主要原因,那么G、C和A、T碱基对会在密码子中被均匀使用;相反,如果选择压力是主要原因,则G、C和A、T碱基对不会被均匀使用。综合中性绘图和PR2-plot的分析结果发现,珙桐叶绿体基因组的同义密码子使用偏好性主要受到自然选择压力的影响。此外,RSCU通常表示密码子偏好性的强弱[24],为探究基因表达水平和碱基等因素对珙桐同义密码子使用偏好性的影响,基于RSCU的对应分析结果表明,Axis 1与CAI、GC3、GC12、ENC均显著或极显著相关(表3)。结合中性绘图、ENC-plot、PR2-plot和对应性分析结果发现:珙桐叶绿体基因组同义密码子使用偏好性除了主要受选择压力影响外,还受到突变压力、碱基组成、基因表达水平等因素的影响,这一结果与麻风树(Jatropha curcas)[38]和马尾松(Pinus massoniana) [39]等木本植物的研究结果相类似。
近年来,农作物叶绿体转化在抗盐、抗旱、抗除草剂等研究领域取得了重大进展,利用叶绿体基因进行育种研究时往往需要考虑叶绿体基因的稳定性,以及它们的遗传多样性[6]。同义密码子使用偏好性可以通过调节基因翻译的准确性和效率来影响基因表达,同义密码子使用偏好性越强,其基因的表达水平越高[40−42]。在设计叶绿体基因表达载体时,若基于其同义密码子使用偏好性对密码子事前进行优化,则可以提升叶绿体基因组中插入基因的表达水平,也可以利用已知密码子的使用模式来预测未知基因的表达和功能[43]。珙桐叶绿体基因组高度保守,共筛选出37个高频密码子和27个高表达密码子,其中12个均以A或U结尾的密码子被确定为最优密码子(表4),最优密码子第3位碱基的组成与拟南芥(Arabidopsis thaliana)、烟草(Nicotiana tabacum)、杨树(Populus spp.)等双子叶模式植物偏好相似[44−46]。这些最优密码子不仅可以作为检测基因或开放阅读框表达水平的指标,还可以用于引物设计和点突变引入等生物育种研究当中。
4. 结 论
珙桐叶绿体基因组密码子使用偏好性较弱,其同义密码子使用偏好性除了主要受选择压力影响外,还受到突变压力、碱基组成和基因表达水平等因素的影响。同时筛选出12个最优密码子,可用于珙桐未来的遗传改良和种质创新研究。
-
图 4 珙桐叶绿体基因组PR2-plot绘图分析
A3. 密码子第3位A碱基的含量;T3. 密码子第3位T碱基的含量;G3. 密码子第3位G碱基的含量;C3. 密码子第3位C碱基的含量。A3, adenine (A) content at the third position of codon; T3, thymine (T) content at the third position of codon; G3, guanine (G) content at the third position of codon; C3, cytosine (C) content at the third position of codon.
Figure 4. Analysis of PR2-plot of chloroplast genomes of D. involucrata
表 1 珙桐叶绿体基因组密码子GC含量及CAI和ENC值
Table 1 GC proportion, CAI and ENC in chloroplast genomics of Davidia involucrata
基因
GeneCAI ENC GC含量 GC content 基因
GeneCAI ENC GC含量 GC content GCall/% GC1/% GC2/% GC3/% GCall/% GC1/% GC2/% GC3/% rps8 0.08 37.04 36.13 39.86 43.36 25.17 ycf2 0.15 48.53 37.78 37.08 35.00 41.25 rps18 0.11 37.57 35.29 35.29 44.12 26.47 ndhK 0.16 48.55 38.05 44.25 44.69 25.22 rps14 0.11 38.09 41.91 44.55 49.50 31.68 ycf4 0.16 48.82 38.10 42.33 39.68 32.28 ndhE 0.16 40.41 32.68 38.24 35.29 24.51 rpoB 0.15 48.85 38.94 50.89 37.72 28.20 psbA 0.32 40.82 42.66 50.28 43.50 34.18 psbB 0.20 48.86 44.47 54.81 45.97 32.61 ndhF 0.15 43.72 32.71 37.72 35.97 24.43 rpoA 0.16 49.64 34.62 44.38 32.84 26.63 ndhF 0.15 43.72 32.71 37.63 36.16 24.60 rps4 0.14 49.84 39.27 51.98 39.11 26.73 rps3 0.16 43.87 35.62 47.49 33.33 26.03 rps2 0.18 49.95 39.66 44.73 44.30 29.96 psbD 0.25 44.42 42.28 52.26 43.22 31.36 rpoC2 0.15 50.04 37.97 45.82 38.50 29.58 rps7 0.19 45.03 40.60 53.21 45.51 23.08 matK 0.15 50.13 34.19 41.39 32.08 29.11 rps7 0.19 45.03 40.60 53.21 45.51 23.08 atpE 0.16 51.02 39.80 51.49 39.55 28.36 rpl23 0.13 45.75 37.39 40.00 41.74 30.43 ndhH 0.16 51.13 39.26 51.52 37.06 29.19 ndhG 0.13 45.96 34.09 44.07 33.90 24.29 ycf2 0.21 51.22 37.78 36.70 32.11 39.45 atpB 0.21 46.12 42.48 56.31 42.08 29.06 ndhA 0.14 51.26 33.17 36.42 32.11 30.98 ndhD 0.14 46.24 35.29 41.37 36.86 27.65 clpP 0.16 51.31 36.21 37.28 38.06 33.29 psbC 0.18 46.25 44.02 53.38 46.20 32.49 ndhJ 0.15 51.37 40.25 51.57 37.74 31.45 ndhC 0.20 46.41 34.44 45.45 33.06 24.79 ndhB 0.17 51.78 37.84 36.60 39.33 37.58 rpl22 0.20 46.70 34.60 43.67 36.08 24.05 ndhB 0.16 52.51 37.84 35.82 41.33 36.40 rps11 0.15 46.75 44.84 54.68 57.55 22.30 petA 0.18 53.14 39.98 52.34 37.07 30.53 cemA 0.20 46.78 33.62 40.87 29.13 30.87 ycf3 0.15 53.19 35.92 34.78 39.49 33.48 rpl14 0.18 46.80 41.46 56.10 37.40 30.89 petD 0.16 53.20 36.72 38.41 30.44 41.31 atpF 0.13 46.85 36.25 33.88 34.30 40.58 rpl16 0.13 54.37 34.66 38.16 35.01 30.82 accD 0.19 46.90 33.04 36.01 35.12 27.98 rpoC1 0.15 56.05 37.99 38.82 33.12 42.03 atpI 0.18 47.17 37.90 48.39 37.90 27.42 rps16 0.16 56.33 34.92 40.63 31.93 32.19 rpl20 0.09 47.55 36.16 38.98 44.07 25.42 rps12 0.15 56.58 39.74 44.21 40.14 34.86 atpA 0.20 47.60 40.94 55.12 40.55 27.17 petB 0.15 57.40 36.23 38.52 31.15 39.01 psaB 0.18 47.71 40.91 48.71 42.99 31.02 rpl2 0.15 58.12 42.08 39.32 45.64 41.29 rbcL 0.28 47.86 43.77 57.77 43.70 29.83 rpl2 0.14 58.78 42.08 45.27 38.63 41.53 psaA 0.20 48.40 42.79 52.73 43.41 32.22 rpl23 0.23 48.68 37.39 41.03 33.33 33.33 ndhI 0.19 48.52 34.72 42.26 36.31 25.60 平均值 Mean 0.18 48.52 38.05 44.51 38.83 30.63 注:CAI. 密码子适应指数; ENC. 有效密码子数; GCall . 基因中所有密码子的的GC含量;GC1 . 基因中所有密码子的第1位的GC含量;GC2. 基因中所有密码子的第2位的GC含量;GC3. 基因中所有密码子的第3位的GC含量。Notes: CAI, codon adaptation index; ENC, effective number of codon; GCall, GC content of all codons in the gene; GC1, the GC content in position 1 of all codons in the gene; GC2, the GC content in position 2 of all codons in the gene; GC3, the GC content in position 3 of all codons in the gene. 表 2 ENC比值的频率分布
Table 2 Frequency distribution of ENC ratio
组限
Group value range组中值
Group mid-value组数
Group number频率
Frequency/%−0.25 ~ 0.15 0.2 6 10.17 −0.15 ~ 0.05 0.1 12 20.34 −0.05 ~ 0.05 0 35 59.32 0.05 ~ 0.15 0.1 6 8.47 表 3 珙桐叶绿体基因组密码子偏好指标相关性分析
Table 3 Correlation analysis of codon usage index of chloroplast genomes of D. involucrata
参数
ParameterAxis 1 ENC CAI GC12 GC3 ENC −0.685 0** CAI 0.139 0* −0.065 5** GC12 0.406 0** −0.240 0 0.361 0** GC3 −0.927 0** 0.625 0** −0.040 4 −0.330 0 GCall −0.133 0 0.137 0 0.356 0** 0.817 0** 0.263 0* 注:*. 显著相关(P < 0.05);**. 极显著相关(P < 0.01)。Notes: *, a significant correlation at P < 0.05 level; **, a highly significant correlation at P < 0.01 level. 表 4 珙桐叶绿体基因组的最优密码子
Table 4 Optimal codons in chloroplast genome of D. involucrata
氨基酸
Amino acid密码子
Codon高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 氨基酸
Amino Acid密码子
Codon高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 数目
NumberRSCU 数目
NumberRSCU 数目
NumberRSCU 数目
NumberRSCU 亮氨酸 Leu UUA** 54 1.59 26 1.28 0.31 酪氨酸 Tyr UAU** 50 1.39 25 1.04 0.35 UUG 45 1.32 30 1.48 −0.16 UAC 22 0.61 23 0.96 −0.35 CUU*** 41 1.21 12 0.59 0.62 *Ter UAA*** 5 1.88 32 1.16 0.72 CUC 17 0.5 19 0.93 −0.43 UGA 2 0.75 31 1.12 −0.37 CUA 32 0.94 24 1.18 −0.24 UAG 1 0.38 20 0.72 −0.34 CUG 15 0.44 11 0.54 −0.1 组氨酸 His CAU* 34 1.36 14 1.27 0.09 异亮氨酸 Ile AUU** 74 1.52 40 1.19 0.33 CAC 16 0.64 8 0.73 −0.09 AUC** 49 1.01 23 0.68 0.33 谷氨酰胺 Gln CAA 46 1.44 26 1.44 0 AUA 23 0.47 38 1.13 −0.66 CAG 18 0.56 10 0.56 0 甲硫氨酸 met AUG 58 1 21 1 0 天冬酰胺 Asn AAU* 66 1.28 25 1.02 0.26 缬氨酸 Val GUU* 49 1.39 19 1.21 0.18 AAC 37 0.72 24 0.98 −0.26 GUC 13 0.37 13 0.83 −0.46 赖氨酸 Lys AAA* 60 1.46 46 1.23 0.23 GUA*** 66 1.87 18 1.14 0.73 AAG 22 0.54 29 0.77 −0.23 GUG 13 0.37 13 0.83 −0.46 天冬氨酸 Asp GAU** 59 1.37 20 1.05 0.32 丝氨酸 Ser UCU*** 48 2.03 8 0.98 1.05 GAC 27 0.63 18 0.95 −0.32 UCC* 23 0.97 7 0.86 0.11 谷氨酸 Glu GAA*** 99 1.56 37 0.94 0.62 UCA* 25 1.06 8 0.98 0.08 GAG 28 0.44 42 1.06 −0.62 UCG 7 0.3 6 0.73 −0.43 半胱氨酸 Cys UGU* 12 1.2 10 1.11 0.09 AGU 26 1.1 11 1.35 −0.25 UGC 8 0.8 8 0.89 −0.09 AGC 13 0.55 9 1.1 −0.55 色氨酸 Trp UGG 41 1 25 1 0 脯氨酸 Pro CCU*** 52 2.12 9 0.97 1.15 精氨酸 Arg CGU*** 45 2.21 4 0.36 1.85 CCC 14 0.57 5 0.54 0.03 CGC** 12 0.59 1 0.09 0.5 CCA 19 0.78 16 1.73 −0.95 CGA** 23 1.13 8 0.72 0.41 CCG 13 0.53 7 0.76 −0.23 CGG 5 0.25 2 0.18 0.07 苏氨酸 Thr ACU*** 63 2 5 0.67 1.33 AGA 32 1.57 47 4.21 −2.64 ACC* 24 0.76 4 0.53 0.23 AGG 5 0.25 5 0.45 −0.2 ACA 28 0.89 15 2 −1.11 苯丙氨酸 Phe UUU 68 0.96 47 1.12 −0.16 ACG 11 0.35 6 0.8 −0.45 UUC* 74 1.04 37 0.88 0.16 丙氨酸 Ala GCU*** 92 2.19 5 1.25 0.94 甘氨酸 Gly GGU*** 87 1.87 7 0.46 1.41 GCC 20 0.48 3 0.75 −0.27 GGC 17 0.37 8 0.52 −0.15 GCA 40 0.95 4 1 −0.05 GGA 57 1.23 29 1.9 −0.67 GCG 16 0.38 4 1 −0.62 GGG 25 0.54 17 1.11 −0.57 注:加下划线的密码子的RSCU > 1;*代表0.08 < ΔRSCU < 0.3;**代表0.3 < ΔRSCU < 0.5;***代表ΔRSCU > 0.5,加粗密码子被认为是最优密码子。Notes: the underlined codon,RSCU > 1; *, 0.08 < ΔRSCU < 0.3; **, 0.3 < ΔRSCU < 0.5; ***, ΔRSCU > 0.5; the bold codons are thoght to be the optimal codons. -
[1] Ikemura T. Codon usage and tRNA content in unicellular and multicellular organisms[J]. Molecular Biology and Evolution, 1985, 2(1): 13−34.
[2] Parvathy S T, Udayasuriyan V, Bhadana V. Codon usage bias[J]. Molecular Biology Reports, 2022, 49(1): 539−565. doi: 10.1007/s11033-021-06749-4
[3] Botzman M, Margalit H. Variation in global codon usage bias among prokaryotic organisms is associated with their lifestyles[J]. Genome Biology, 2011, 12(10): R109. doi: 10.1186/gb-2011-12-10-r109
[4] Srivastava S, Chanyal S, Dubey A, et al. Patterns of codon usage bias in WRKY genes of Brassica rapa and Arabidopsis thaliana[J]. Journal of Agricultural Science, 2019, 11(4): 76. doi: 10.5539/jas.v11n4p76
[5] Xin L, Wang X, Gong P, et al. Analysis of codon usage patterns in Giardia duodenalis based on transcriptome data from GiardiaDB[J]. Genes, 2021, 12(8): 1169. doi: 10.3390/genes12081169
[6] Li G, Zhang L, Xue P. Codon usage pattern and genetic diversity in chloroplast genomes of Panicum species[J]. Gene, 2021, 15(802): 145866.
[7] Wang P, Mao Y, Su Y, et al. Comparative analysis of transcriptomic data shows the effects of multiple evolutionary selection processes on codon usage in Marsupenaeus japonicus and Marsupenaeus pulchricaudatus[J]. BMC Genomics, 2021, 22(1): 781. doi: 10.1186/s12864-021-08106-y
[8] Nie X, Deng P, Feng K, et al. Comparative analysis of codon usage patterns in chloroplast genomes of the Asteraceae family[J]. Plant Molecular Biology Reporter, 2014, 32(4): 828−840. doi: 10.1007/s11105-013-0691-z
[9] Soltis P S, Soltis D E. The role of genetic and genomic attributes in the success of polyploids[J]. Proceedings of the National Academy of Sciences, 2000, 97(13): 7051−7057. doi: 10.1073/pnas.97.13.7051
[10] Fu G, Liu Y, Caraballo-Ortiz M A, et al. Characterization of the complete chloroplast genome of the dragonhead herb, Dracocephalum heterophyllum (Lamiaceae), and comparative analyses with related species[J]. Diversity, Multidisciplinary Digital Publishing Institute, 2022, 14(2): 110.
[11] 王鹏良, 吴双成, 杨利平, 等. 巨桉叶绿体基因组密码子偏好性分析[J]. 广西植物, 2019, 39(12): 1583−1592. Wang P L, Wu S C, Yang L P, et al. Codon bias analysis of Chloroplast genome of Eucalyptus grandis[J]. Guangxi Botany, 2019, 39(12): 1583−1592.
[12] 雷慧, 李鸽, 王娜玉. 金莲花叶绿体基因组密码子偏好性分析[J]. 山西农业科学, 2019, 47(8): 1300−1305. Lei H, Li G, Wang N Y. Analysis of codon bias in chloroplast genome of lotus flower[J]. Journal of Shanxi Agricultural Sciences, 2019, 47(8): 1300−1305.
[13] Li G L, Pan Z L, Gao S C, et al. Analysis of synonymous codon usage of chloroplast genome in Porphyra umbilicalis[J]. Genes & Genomics, 2019, 41(10): 1173−1181.
[14] Duan H, Zhang Q, Wang C, et al. Analysis of codon usage patterns of the chloroplast genome in Delphinium grandiflorum L. reveals a preference for AT-ending codons as a result of major selection constraints[J]. PeerJ, 2021, 9(18): e10787.
[15] Wang Z, Xu B, Li B, et al. Comparative analysis of codon usage patterns in chloroplast genomes of six Euphorbiaceae species[J]. Peer Journal, 2020, 8: e8251.
[16] Chakraborty S, Yengkhom S, Uddin A. Analysis of codon usage bias of chloroplast genes in Oryza species[J]. Planta, 2020, 252(4): 67. doi: 10.1007/s00425-020-03470-7
[17] Li Y X, Chen L, Juan L, et al. Suppression subtractive hybridization cloning of cDNAs of differentially expressed genes in dovetree ( Davidia involucrata) bracts[J]. Plant Molecular Biology Reporter, 2002, 20(3): 231−238. doi: 10.1007/BF02782458
[18] Qin H, Jin X, Zhao L. Rare and endangered plants in China[G]//REN H. Conservation and reintroduction of rare and endangered plants in China. Singapore City: Springer, 2020: 21–31.
[19] Jerominek M, Bull-Hereñu K, Arndt M, et al. Live imaging of developmental processes in a living meristem of Davidia involucrata (Nyssaceae)[J]. Frontiers in Plant Science, 2014, 5: 613.
[20] Tang C Q, Dong Y F, Herrando-Moraira S, et al. Potential effects of climate change on geographic distribution of the Tertiary relict tree species Davidia involucrata in China[J]. Scientific Reports, 2017, 7(1): 43822. doi: 10.1038/srep43822
[21] Claßen-Bockhoff R, Arndt M. Flower-like heads from flower-like meristems: pseudanthium development in Davidia involucrata (Nyssaceae)[J]. Journal of Plant Research, 2018, 131(3): 443−458. doi: 10.1007/s10265-018-1029-6
[22] Ren F, Kovalchuk A, Mukrimin M, et al. Tissue microbiome of Norway spruce affected by heterobasidion-induced wood decay[J]. Microbial Ecology, 2019, 77(3): 640−650. doi: 10.1007/s00248-018-1240-y
[23] Yang H, Zhou C, Li G, et al. Reference gene and small RNA data from multiple tissues of Davidia involucrata Baill[J]. Scientific Data, 2019, 6(1): 181. doi: 10.1038/s41597-019-0190-7
[24] Chi X, Zhang F, Dong Q, et al. Insights into comparative genomics, codon usage bias, and phylogenetic relationship of species from biebersteiniaceae and nitrariaceae based on complete chloroplast genomes[J]. Plants, Multidisciplinary Digital Publishing Institute, 2020, 9(11): 1605.
[25] 毛立彦, 黄秋伟, 龙凌云, 等. 7种睡莲属植物叶绿体基因组密码子偏好性分析[J]. 西北林学院学报, 2022, 37(2): 98−107. Mao L Y, Huang Q W, Long L Y, et al. Codon bias analysis of chloroplast genome of seven Nymphaea species[J]. Journal of Northwest Forestry University, 2022, 37(2): 98−107.
[26] 蔡元保, 杨祥燕. 澳洲坚果光壳种叶绿体基因组的密码子使用偏好性及其影响因素分析[J]. 植物科学学报, 2022, 40(2): 229−239. Cai Y B, Yang X Y. Analysis of codon use preference and its influencing factors in chloroplast genome of Macadamia integrifolia[J]. Chinese Journal of Plant Science, 2022, 40(2): 229−239.
[27] 张以忠, 曾文艺, 邓琳琼, 等. 甘蓝S-位点基因SRK、SLG和SP11/SCR密码子偏好性分析[J]. 作物学报, 2022, 48(5): 1152−1168. doi: 10.3724/SP.J.1006.2022.14003 Zhang Y Z, Zeng W Y, Deng L Q, et al. Codon bias analysis of S-locus genes SRK, SLG and SP11/SCR in Brassica oleracea[J]. Acta Agronomica Sinica, 2022, 48(5): 1152−1168. doi: 10.3724/SP.J.1006.2022.14003
[28] 杨祥燕, 蔡元保, 谭秦亮, 等. 菠萝叶绿体基因组密码子偏好性分析[J]. 热带作物学报, 2022, 43(3): 439−446. Yang X Y, Cai Y B, Tan Q L, et al. Analysis of codon bias in chloroplast genome of Pineapple[J]. Chinese Journal of Tropical Crops, 2022, 43(3): 439−446.
[29] Liu H, He R, Zhang H, et al. Analysis of synonymous codon usage in Zea mays[J]. Molecular Biology Reports, 2010, 37(2): 677−684. doi: 10.1007/s11033-009-9521-7
[30] Liu Q P, Feng Y, Xue Q Z. Analysis of factors shaping codon usage in the mitochondrion genome of Oryza sativa[J]. Mitochondrion, 2004, 4: 313−20. doi: 10.1016/j.mito.2004.06.003
[31] Majeed A, Kaur H, Kaur A, et al. Codon usage pattern in Gnetales evolved in close accordance with the Gnetifer hypothesis[J]. Botanical Journal of the Linnean Society, 2021, 196(4): 423−436. doi: 10.1093/botlinnean/boab006
[32] Liu Q, Xue Q. Comparative studies on codon usage pattern of chloroplasts and their host nuclear genes in four plant species[J]. Journal of Genetics, 2005, 84(1): 55−62. doi: 10.1007/BF02715890
[33] Mazumdar P, Binti O R, Mebus K, et al. Codon usage and codon pair patterns in non-grass monocot genomes[J]. Annals of Botany, 2017, 120(6): 893−909. doi: 10.1093/aob/mcx112
[34] 张俊焱, 曾阳, 李锦萍, 等. 2种药用獐牙菜叶绿体基因组密码子偏好性分析[J]. 中国中医药信息杂志, 2022, 29(1): 96−102. Zhang J Y, Zeng Y, Li J P, et al. Analysis of codon usage bias in chloroplast genome of two Medicinal Swertia L.[J]. Chinese Journal of Traditional Chinese Medicine Information, 2022, 29(1): 96−102.
[35] 王琳璇, 王高烽, 谷威, 等. 2种羌活药用植物的叶绿体基因组密码子使用偏性分析[J]. 安徽农业科学, 2022, 50(3): 95−101. Wang L X, Wang G F, Gu W, et al. Codon usage bias in chloroplast genomes of two medicinal species of Notopterygium H. de Boissieu[J]. Journal of Anhui Agricultural Sciences, 2022, 50(3): 95−101.
[36] Monroe J G, Srikant T, Carbonell-Bejerano P, et al. Mutation bias reflects natural selection in Arabidopsis thaliana[J]. Nature, Nature Publishing Group, 2022, 602: 101−105.
[37] Frank W. The effective number of codons used in a gene[J]. Gene, 1990, 317(3): 957−964.
[38] Wang Z J, Wang G Y, Cai Q W, et al. Genome wide comparative analysis of codon usage bias in three sequenced Jatropha curcas[J]. Journal of Genetics, 2021, 100(1): 20. doi: 10.1007/s12041-021-01271-9
[39] 罗群凤, 冯源恒, 贾婕, 等. 马尾松叶绿体基因组测序及特征分析[J]. 广西林业科学, 2018, 47(4): 396−402. Luo Q F, Feng Y H, Jia J, et al. Sequencing and characteristics analysis of chloroplast genome of Pinus massoniana[J]. Journal of Guangxi Forestry Sciences, 2018, 47(4): 396−402.
[40] Hershberg R, Petrov D A. Selection on codon bias[J]. Annual Review of Genetics, 2008, 42: 287−299. doi: 10.1146/annurev.genet.42.110807.091442
[41] 邢朝斌, 曹蕾, 周秘, 等. 刺五加叶绿体基因组密码子的用法分析[J]. 中国中药杂志, 2013, 38(5): 661−665. Xing Z B, Cao L, Zhou M, et al. Analysis on codon usage of chloroplast genome of Eleutherococcus senticosus[J]. China Journal of Chinese Materia Medica, 2013, 38(5): 661−665.
[42] Zhou Z, Dang Y, Zhou M, et al. Codon usage is an important determinant of gene expression levels largely through its effects on transcription[J]. Proceedings of the National Academy of Sciences, 2016, 113(41): E6117−E6125.
[43] Cui G, Wang C, Wei X, et al. Complete chloroplast genome of Hordeum brevisubulatum: genome organization, synonymous codon usage, phylogenetic relationships, and comparative structure analysis[J]. PLoS ONE, 2021, 16(12): e0261196. doi: 10.1371/journal.pone.0261196
[44] Sahoo S, Das S S, Rakshit R. Codon usage pattern and predicted gene expression in Arabidopsis thaliana[J]. Gene: X, 2019, 2: 100012.
[45] Zhou M, Tong C F, Shi J S. A preliminary analysis of synonymous codon usage in poplar species[J]. Journal of Plant Physiology and Molecular Biology, 2007, 33(4): 285−293.
[46] Hershberg R, Petrov D A. General rules for optimal codon choice[J]. PLoS Genetics, 2009, 5(7): e1000556. doi: 10.1371/journal.pgen.1000556
-
期刊类型引用(11)
1. 杨勇,陈林,庞丹波,李学斌,刘瑞亮. 不同凋落物处理对贺兰山三种林分土壤呼吸的影响. 西南农业学报. 2023(03): 593-601 .  百度学术
百度学术
2. 陈炎根,胡艳静,黄莎,刘波,吴继来,王懿祥. 不同间伐强度对杉木人工林土壤呼吸速率的短期影响. 浙江农林大学学报. 2023(05): 1054-1062 .  百度学术
百度学术
3. 沈健,何宗明,董强,郜士垒,林宇,石焱. 不同处理方式下湿地松人工林土壤呼吸及温度敏感性变化. 西北林学院学报. 2023(05): 10-18 .  百度学术
百度学术
4. 刘洪柳,王泽鑫,郭晋平,张芸香. 林分密度对华北落叶松天然次生林林下植被和土壤呼吸的影响. 广西林业科学. 2022(02): 190-196 .  百度学术
百度学术
5. 李素新,覃志杰,刘泰瑞,郭晋平. 模拟氮沉降对华北落叶松人工林土壤微生物碳和微生物氮的动态影响. 水土保持学报. 2020(01): 268-274 .  百度学术
百度学术
6. 段北星,蔡体久,宋浩,肖瑞晗. 寒温带兴安落叶松林凋落物层对土壤呼吸的影响. 生态学报. 2020(04): 1357-1366 .  百度学术
百度学术
7. 巫志龙,周成军,周新年,刘富万,朱奇雄,黄金湧,陈文. 不同强度采伐5年后杉阔混交人工林土壤呼吸速率差异. 林业科学. 2019(06): 142-149 .  百度学术
百度学术
8. 段北星,满秀玲,宋浩,刘家霖. 大兴安岭北部不同类型兴安落叶松林土壤呼吸及其组分特征. 北京林业大学学报. 2018(02): 40-50 .  本站查看
本站查看
9. 李良,夏富才,孙越,张骁. 阔叶红松林下早春植物生物量分配. 北京林业大学学报. 2017(01): 34-42 .  本站查看
本站查看
10. 邵英男,田松岩,刘延坤,李云红,陈瑶,孙志虎. 密度调控对长白落叶松人工林土壤呼吸的影响. 北京林业大学学报. 2017(06): 51-59 .  本站查看
本站查看
11. 赵佳琪,牟长城,吴彬,周雪娇. 造林与间伐对东北温带弃耕地土壤温室气体排放的长期影响. 北京林业大学学报. 2017(10): 13-23 .  本站查看
本站查看
其他类型引用(18)



 下载:
下载: